Un árbol filogenético , filogenia o árbol evolutivo es una representación gráfica que muestra la historia evolutiva entre un conjunto de especies o taxones durante un tiempo específico. [1] [2] En otras palabras, es un diagrama de ramificación o un árbol que muestra las relaciones evolutivas entre varias especies biológicas u otras entidades basadas en similitudes y diferencias en sus características físicas o genéticas. En biología evolutiva, toda la vida en la Tierra es teóricamente parte de un solo árbol filogenético, lo que indica una ascendencia común . La filogenética es el estudio de los árboles filogenéticos. El principal desafío es encontrar un árbol filogenético que represente la ascendencia evolutiva óptima entre un conjunto de especies o taxones. La filogenética computacional (también inferencia de filogenia) se centra en los algoritmos involucrados en la búsqueda del árbol filogenético óptimo en el panorama filogenético. [1] [2]
Los árboles filogenéticos pueden tener raíces o no. En un árbol filogenético con raíces , cada nodo con descendientes representa el ancestro común más reciente inferido de esos descendientes, [3] y las longitudes de los bordes en algunos árboles pueden interpretarse como estimaciones de tiempo. Cada nodo se denomina unidad taxonómica. Los nodos internos generalmente se denominan unidades taxonómicas hipotéticas, ya que no se pueden observar directamente. Los árboles son útiles en campos de la biología como la bioinformática , la sistemática y la filogenética . Los árboles sin raíces ilustran solo la relación de los nodos de las hojas y no requieren que se conozca o infiera la raíz ancestral.
La idea de un árbol de la vida surgió de nociones antiguas de una progresión en forma de escalera desde formas de vida inferiores a superiores (como en la Gran Cadena del Ser ). Las primeras representaciones de árboles filogenéticos "ramificados" incluyen un "diagrama paleontológico" que muestra las relaciones geológicas entre plantas y animales en el libro Geología elemental , de Edward Hitchcock (primera edición: 1840).
Charles Darwin presentó un "árbol" evolutivo esquemático en su libro El origen de las especies de 1859. Más de un siglo después, los biólogos evolucionistas todavía utilizan diagramas de árbol para representar la evolución porque estos diagramas transmiten eficazmente el concepto de que la especiación se produce a través de la división adaptativa y semialeatoria de linajes.
El término filogenético , o filogenia , deriva de las dos palabras griegas antiguas φῦλον ( phûlon ), que significa "raza, linaje", y γένεσις ( génesis ), que significa "origen, fuente". [4] [5]

Un árbol filogenético enraizado (ver los dos gráficos en la parte superior) es un árbol dirigido con un nodo único —la raíz— que corresponde al ancestro común más reciente (generalmente imputado ) de todas las entidades en las hojas del árbol. El nodo raíz no tiene un nodo padre, pero sirve como padre de todos los demás nodos del árbol. Por lo tanto, la raíz es un nodo de grado 2, mientras que otros nodos internos tienen un grado mínimo de 3 (donde "grado" aquí se refiere al número total de aristas entrantes y salientes). [ cita requerida ]
El método más común para enraizar árboles es el uso de un grupo externo no controvertido : lo suficientemente cercano como para permitir la inferencia a partir de datos de rasgos o secuenciación molecular, pero lo suficientemente lejano como para ser un grupo externo claro. Otro método es el enraizamiento de punto medio, o un árbol también puede enraizarse utilizando un modelo de sustitución no estacionaria . [6]

Los árboles sin raíz ilustran la relación de los nodos de las hojas sin hacer suposiciones sobre la ascendencia. No requieren que se conozca o infiera la raíz ancestral. [8] Los árboles sin raíz siempre se pueden generar a partir de árboles con raíz simplemente omitiendo la raíz. Por el contrario, inferir la raíz de un árbol sin raíz requiere algún medio para identificar la ascendencia. Esto normalmente se hace incluyendo un grupo externo en los datos de entrada de modo que la raíz esté necesariamente entre el grupo externo y el resto de los taxones en el árbol, o introduciendo suposiciones adicionales sobre las tasas relativas de evolución en cada rama, como una aplicación de la hipótesis del reloj molecular . [9]
Tanto los árboles con raíz como los que no la tienen pueden ser bifurcadores o multifurcadores. Un árbol bifurcador con raíz tiene exactamente dos descendientes que surgen de cada nodo interior (es decir, forma un árbol binario ), y un árbol bifurcador sin raíz toma la forma de un árbol binario sin raíz , un árbol libre con exactamente tres vecinos en cada nodo interno. Por el contrario, un árbol multifurcador con raíz puede tener más de dos hijos en algunos nodos y un árbol multifurcador sin raíz puede tener más de tres vecinos en algunos nodos. [ cita requerida ]
Tanto los árboles con raíces como los que no las tienen pueden estar etiquetados o no etiquetados. Un árbol etiquetado tiene valores específicos asignados a sus hojas, mientras que un árbol sin etiquetas, a veces llamado forma de árbol, define solo una topología. Algunos árboles basados en secuencias construidos a partir de un pequeño locus genómico, como Phylotree, [10] presentan nodos internos etiquetados con haplotipos ancestrales inferidos.

La cantidad de árboles posibles para una cantidad dada de nodos de hojas depende del tipo específico de árbol, pero siempre hay más árboles etiquetados que no etiquetados, más árboles multifurcantes que bifurcantes y más árboles enraizados que no enraizados. La última distinción es la más relevante biológicamente; surge porque hay muchos lugares en un árbol no enraizado donde poner la raíz. Para los árboles etiquetados bifurcantes, la cantidad total de árboles enraizados es:
Para árboles etiquetados bifurcados, el número total de árboles sin raíz es: [11]
Entre los árboles bifurcados etiquetados, el número de árboles sin raíces con hojas es igual al número de árboles con raíces con hojas. [2]
El número de árboles enraizados crece rápidamente en función del número de puntas. Para 10 puntas, hay más que posibles árboles bifurcadores, y el número de árboles multifurcadores aumenta más rápido, con aproximadamente 7 veces más de estos últimos que de los primeros.
Un dendrograma es un nombre general para un árbol, ya sea filogenético o no, y por lo tanto también para la representación diagramática de un árbol filogenético. [12]
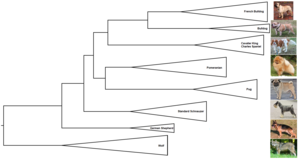
Un cladograma sólo representa un patrón de ramificación; es decir, las longitudes de sus ramas no representan el tiempo o la cantidad relativa de cambio de carácter, y sus nodos internos no representan ancestros. [13]
Un filograma es un árbol filogenético que tiene longitudes de ramas proporcionales a la cantidad de cambio de caracteres. [14]
Un cronograma es un árbol filogenético que representa explícitamente el tiempo a través de la longitud de sus ramas. [15]
Un dahlgrenograma es un diagrama que representa una sección transversal de un árbol filogenético. [ cita requerida ]
Una red filogenética no es, en sentido estricto, un árbol, sino un grafo más general , o un grafo acíclico dirigido en el caso de las redes enraizadas. Se utilizan para superar algunas de las limitaciones inherentes a los árboles.

Un diagrama de husos, o diagrama de burbujas, a menudo se denomina romerograma, después de su popularización por el paleontólogo estadounidense Alfred Romer . [17] Representa la diversidad taxonómica (ancho horizontal) contra el tiempo geológico (eje vertical) para reflejar la variación de la abundancia de varios taxones a través del tiempo. Un diagrama de husos no es un árbol evolutivo: [18] los husos taxonómicos oscurecen las relaciones reales del taxón padre con el taxón hijo [17] y tienen la desventaja de involucrar la parafilia del grupo parental. [19] Este tipo de diagrama ya no se usa en la forma propuesta originalmente. [19]
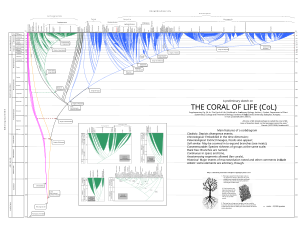
Darwin [20] también mencionó que el coral puede ser una metáfora más adecuada que el árbol . De hecho, los corales filogenéticos son útiles para representar la vida pasada y presente, y tienen algunas ventajas sobre los árboles ( anastomosis permitidas, etc.). [19]
Los árboles filogenéticos compuestos con un número no trivial de secuencias de entrada se construyen utilizando métodos de filogenética computacional . Los métodos de matriz de distancia como neighbor-joining o UPGMA , que calculan la distancia genética a partir de múltiples alineaciones de secuencias , son los más simples de implementar, pero no invocan un modelo evolutivo. Muchos métodos de alineamiento de secuencias como ClustalW también crean árboles utilizando los algoritmos más simples (es decir, los basados en la distancia) de construcción de árboles. La parsimonia máxima es otro método simple para estimar árboles filogenéticos, pero implica un modelo implícito de evolución (es decir, parsimonia). Los métodos más avanzados utilizan el criterio de optimalidad de máxima verosimilitud , a menudo dentro de un marco bayesiano , y aplican un modelo explícito de evolución a la estimación del árbol filogenético. [2] Identificar el árbol óptimo utilizando muchas de estas técnicas es NP-hard , [2] por lo que se utilizan métodos de búsqueda y optimización heurística en combinación con funciones de puntuación de árboles para identificar un árbol razonablemente bueno que se ajuste a los datos.
Los métodos de construcción de árboles se pueden evaluar sobre la base de varios criterios: [21]
Las técnicas de construcción de árboles también han llamado la atención de los matemáticos. Los árboles también pueden construirse utilizando la teoría T. [22]
Los árboles se pueden codificar en varios formatos diferentes, todos los cuales deben representar la estructura anidada de un árbol. Pueden codificar o no longitudes de ramas y otras características. Los formatos estandarizados son fundamentales para distribuir y compartir árboles sin depender de una salida gráfica que es difícil de importar al software existente. Los formatos más utilizados son
Aunque los árboles filogenéticos producidos sobre la base de genes secuenciados o datos genómicos en diferentes especies pueden proporcionar una visión evolutiva, estos análisis tienen limitaciones importantes. Lo más importante es que los árboles que generan no son necesariamente correctos: no necesariamente representan con precisión la historia evolutiva de los taxones incluidos. Como ocurre con cualquier resultado científico, están sujetos a falsificación por estudios posteriores (por ejemplo, recopilación de datos adicionales, análisis de los datos existentes con métodos mejorados). Los datos en los que se basan pueden ser ruidosos ; [23] el análisis puede verse confundido por la recombinación genética , [24] la transferencia horizontal de genes , [25] la hibridación entre especies que no eran vecinas más cercanas en el árbol antes de que se produjera la hibridación y las secuencias conservadas .
Además, existen problemas al basar un análisis en un solo tipo de carácter, como un solo gen o proteína o solo en un análisis morfológico, porque dichos árboles construidos a partir de otra fuente de datos no relacionada a menudo difieren del primero y, por lo tanto, se necesita mucho cuidado al inferir relaciones filogenéticas entre especies. Esto es más cierto en el caso del material genético que está sujeto a transferencia lateral de genes y recombinación , donde diferentes bloques de haplotipos pueden tener diferentes historias. En estos tipos de análisis, el árbol de salida de un análisis filogenético de un solo gen es una estimación de la filogenia del gen (es decir, un árbol genético) y no la filogenia de los taxones (es decir, el árbol de especies) de los que se tomaron muestras de estos caracteres, aunque idealmente, ambos deberían ser muy cercanos. Por esta razón, los estudios filogenéticos serios generalmente utilizan una combinación de genes que provienen de diferentes fuentes genómicas (por ejemplo, de genomas mitocondriales o plastídicos vs. nucleares), [26] o genes que se esperaría que evolucionaran bajo diferentes regímenes selectivos, de modo que sería poco probable que la homoplasia (falsa homología ) fuera el resultado de la selección natural.
Cuando se incluyen especies extintas como nodos terminales en un análisis (en lugar de, por ejemplo, limitar los nodos internos), se considera que no representan ancestros directos de ninguna especie actual. Las especies extintas no suelen contener ADN de alta calidad .
La gama de materiales de ADN útiles se ha ampliado con los avances en las tecnologías de extracción y secuenciación. El desarrollo de tecnologías capaces de inferir secuencias a partir de fragmentos más pequeños o de patrones espaciales de productos de degradación del ADN ampliaría aún más la gama de ADN considerado útil.
Los árboles filogenéticos también pueden inferirse a partir de una variedad de otros tipos de datos, incluida la morfología, la presencia o ausencia de tipos particulares de genes, eventos de inserción y eliminación, y cualquier otra observación que se considere que contiene una señal evolutiva.
Las redes filogenéticas se utilizan cuando los árboles bifurcados no son adecuados, debido a estas complicaciones que sugieren una historia evolutiva más reticulada de los organismos muestreados.
No hay resumen disponible