Phycodnaviridae es una familia de virus de ADN bicatenario grandes (100–560 kb) que infectan algas eucariotas marinas o de agua dulce. Los virus dentro de esta familia tienen una morfología similar, con una cápside icosaédrica (poliedro con 20 caras). En 2014, había 33 especies en esta familia, divididas entre 6 géneros. [1] [2] Esta familia pertenece a un supergrupo de virus grandes conocidos como virus de ADN grande nucleocitoplasmáticos . En 2014 se publicaron pruebas que sugerían que cepas específicas de Phycodnaviridae podrían infectar a los humanos en lugar de solo a las especies de algas, como se creía anteriormente. [3] La mayoría de los géneros de esta familia ingresan a la célula huésped por endocitosis del receptor celular y se replican en el núcleo. Phycodnaviridae desempeñan importantes funciones ecológicas al regular el crecimiento y la productividad de sus huéspedes algas. Las especies de algas como Heterosigma akashiwo y el género Chrysochromulina pueden formar floraciones densas que pueden ser perjudiciales para la pesca, lo que resulta en pérdidas en la industria de la acuicultura. [4] Se ha sugerido el uso del virus Heterosigma akashiwo (HaV) como agente microbiano para prevenir la recurrencia de mareas rojas tóxicas producidas por esta especie de alga. [5] Phycodnaviridae causa la muerte y lisis de especies de algas de agua dulce y marinas, liberando carbono orgánico, nitrógeno y fósforo en el agua, proporcionando nutrientes para el ciclo microbiano. [6]
Grupo: ADN de doble cadena
[2]
La taxonomía de esta familia se basó inicialmente en el rango de hospedadores: los clorovirus infectan algas verdes similares a la clorella de aguas dulces; mientras que los miembros de los otros cinco géneros infectan microalgas marinas y algunas especies de macroalgas pardas. Esto se confirmó posteriormente mediante el análisis de sus polimerasas de ADN de la familia B, que indicaron que los miembros de Phycodnaviridae están más estrechamente relacionados entre sí, en comparación con otros virus de ADN bicatenario, formando un grupo monofilético. [7] [8] [9] Los ficodnavirus contienen seis géneros: Coccolithovirus , Chlorovirus , Phaeovirus , Prasinovirus , Prymnesiovirus y Raphidovirus . Los géneros se pueden distinguir entre sí, por ejemplo, por las diferencias en el ciclo de vida y el contenido genético. [8]
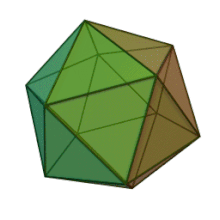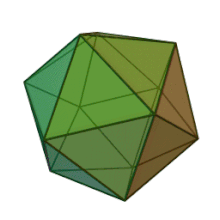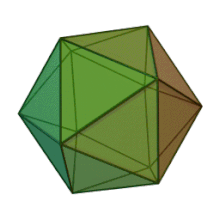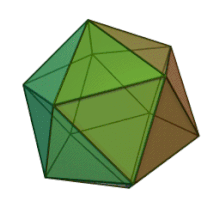
Los seis géneros de la familia Phycodnaviridae tienen una estructura y morfología de virión similares. Son viriones grandes que pueden tener entre 100 y 220 nm de diámetro. Tienen un genoma de ADN de doble cadena y un núcleo proteico rodeado por una bicapa lipídica y una cápside icosaédrica. [10] La cápside tiene ejes de simetría de 2, 3 y 5 pliegues con 20 caras de triángulos equiláteros compuestas de subunidades proteicas. En todos los miembros conocidos de Phycodnaviridae, la cápside está compuesta de subestructuras ordenadas con 20 trisimetrones y 12 pentasimetrones formados por capsómeros triméricos en forma de rosquilla, donde cada capsómero está formado por tres monómeros de la proteína principal de la cápside. Si todos los capsómeros triméricos son idénticos en estructura, la cápside del virión contiene 5040 copias de la proteína principal de la cápside en total con un número de triangulación de 169. En los vértices quíntuples hay 12 capsómeros pentámeros que constan de diferentes proteínas. La(s) proteína(s) que se pueden encontrar debajo del canal axial de cada pentámero pueden ser responsables de digerir la pared celular del huésped durante la infección viral. La especie de virus Phaeocystis puchetii del género Prymnesiovirus tiene la estructura de cápside más grande de la familia Phycodnaviridae . [11]
La membrana de bicapa lipídica en los ficodnavirus no se comprende bien ni se investiga bien. Algunos estudios sugirieron que la membrana se origina en el retículo endoplasmático y también puede adquirirse directamente de la membrana de la célula huésped durante el ensamblaje viral. Aunque los miembros de la familia Phycodnaviridae son muy diversos, comparten genes muy conservados involucrados en la morfología o estructura del virión. [ cita requerida ] A pesar de la similitud de la estructura de la cápside de los ficodnavirus, experimentos recientes han identificado diferencias morfológicas entre los miembros de esta familia. El virus Emiliania huxleyi 86 (EhV-86), una cepa de cocolitovirus, se diferencia de sus contrapartes de virus de algas en que su cápside está envuelta por una membrana lipídica. [12] Además, experimentos recientes de reconstrucción 3D revelaron que el virus de la clorella PBCV-1 tiene una espiga cilíndrica de 250A de largo que se extiende desde uno de sus vértices. EhV-86 también puede poseer una estructura de espiga o cola. [13]
Los ficodnavirus son conocidos por sus grandes genomas de ADN bicatenario que van desde 100 kb hasta más de 550 kb con un contenido de GC del 40% al 50%. [8] Actualmente, hay secuencias genómicas completas disponibles para varios miembros de la familia Phycodnaviridae (incluidos seis clorovirus, dos feovirus, varios prasinovirus y un cocolitovirus) y también hay algunas secuencias parciales disponibles para un cocolitovirus diferente. [14] [15] [16] [17]
Las estructuras genómicas de los ficodnavirus presentan una variación considerable. El clorovirus PBCV-1 tiene un genoma lineal de 330 kb con ADN bicatenario no permutado que está cerrado covalentemente por extremos en horquilla. De manera similar, el feovirus EsV-1 tiene un genoma de ADN bicatenario lineal con repeticiones invertidas que tienen una homología casi perfecta. Estas repeticiones invertidas podrían facilitar la circularización efectiva del genoma y durante un período de tiempo se ha sospechado que EsV-1 tiene un genoma circular. [15] Se sugiere que el cocolitovirus EhV-86 tiene genomas tanto lineales como circulares en diferentes fases durante el empaquetamiento del ADN. La amplificación por PCR revela salientes A/T aleatorios, detección de ligasas de ADN y endonucleasas que insinúan que un genoma lineal puede empaquetarse y circularizarse durante la replicación del ADN. [16] [18] Los ficodnavirus tienen genomas compactos para una replicación eficiente con aproximadamente un gen por cada 900 a 1000 pb de secuencias genómicas. [16] El feovirus EsV-1 es una excepción con 231 genes codificadores de proteínas, lo que significa que tiene un gen por aproximadamente 1450 pb. A pesar de los genomas compactos que se encuentran típicamente en los virus, los genomas de Phycodnaviridae tienen regiones repetitivas generalmente cerca de los extremos terminales y ciertas repeticiones en tándem ubicadas a lo largo del genoma. Se sugiere que estas secuencias repetitivas pueden desempeñar un papel en la recombinación genética que permite al virus intercambiar información genética con otros virus o la célula huésped. [19]
![La historia evolutiva se infirió utilizando el método de máxima verosimilitud basado en el modelo basado en la matriz JTT [1]. El árbol de consenso bootstrap inferido a partir de 100 réplicas se toma para representar la historia evolutiva de los taxones analizados. Las ramas correspondientes a las particiones reproducidas en menos del 50% de las réplicas bootstrap se contraen. El porcentaje de árboles de réplicas en los que los taxones asociados se agruparon en la prueba bootstrap (100 réplicas) se muestra mediante el tamaño del nodo rojo en cada rama. Los árboles iniciales para la búsqueda heurística se obtuvieron automáticamente aplicando los algoritmos Neighbor-Join y BioNJ a una matriz de distancias por pares estimadas utilizando un modelo JTT y luego seleccionando la topología con un valor de verosimilitud logarítmica superior. El análisis involucró 26 secuencias de aminoácidos. Hubo un total de 2599 posiciones en el conjunto de datos final. Los análisis evolutivos se llevaron a cabo en MEGA7.](http://upload.wikimedia.org/wikipedia/commons/thumb/4/48/Molecular_Phylogenetic_analysis_of_NCLDV_members_by_Maximum_Likelihood_method.png/274px-Molecular_Phylogenetic_analysis_of_NCLDV_members_by_Maximum_Likelihood_method.png)
Los virus pertenecientes a Phycodnaviridae albergan genomas de ADN bicatenario con tamaños de varios 100 kbp, que junto con otros Megavirales (por ejemplo, Iridoviridae , Pandoraviridae y Mimiviridae ) se denominan virus de ADN grande nucleocitoplasmáticos . Debido a sus grandes tamaños de genoma y varias proteínas que están codificadas, los virus de Phycodnaviridae están desafiando los conceptos tradicionales de que los virus son pequeños y simples "organismos al borde de la vida". Los análisis filogenéticos de genes centrales basados en la concatenación de genes, [21] filogenias individuales de la ADN polimerasa, [22] y la proteína principal de la cápside, [23] indican las estrechas relaciones evolutivas entre los miembros de Phycodnaviridae y entre Phycodnaviridae y otras familias de virus de ADN grande nucleocitoplasmáticos.
[ cita requerida ]
En Raphidovirus (probablemente mal escrito Rhaphidovirus ), solo hay una especie, el virus Heterosigma akashiwo (HaV), que infecta al alga unicelular, Heterosigma akashiwo . H. akashiwo es un miembro de la clase Raphidophyceae , una especie formadora de floraciones y está ampliamente distribuida en aguas templadas y neríticas . [21] Se han aislado varios otros tipos de virus que infectan a H. akashiwo y no deben confundirse con HaV, como el virus ARN de H. akashiwo (HaRNAV). [25] y el virus de inclusión nuclear de H. akashiwo (HaNIV). [26] [4] Como HaV se aisló y caracterizó por primera vez en 1997, [4] la información sobre el ciclo de vida es limitada.
El virus de la hepatitis A infecta específicamente a H. akashiwo y no infecta a otras especies de fitoplancton marino analizadas. [4] Los mecanismos que determinan la especificidad del virus-huésped no se comprenden bien. Tomaru et al. (2008) [4] sugieren que la especificidad del virus-huésped puede deberse a interacciones únicas entre un ligando viral y un receptor del huésped. En un estudio de Nagaski et al., se encontraron partículas de virus dentro del citoplasma del huésped a las 24 horas posteriores a la infección. Se estimó que el período de latencia o ciclo lisogénico era de 30 a 33 h con un tamaño de ráfaga promedio (número de virus producidos después de la lisis) de 770 por célula. Se encontraron partículas de virus en el área subsuperficial y en el área del viroplasma [5] .

En 2009, MacKinder et al. dilucidaron el mecanismo de entrada del género Coccolithovirus . [12] Utilizando microscopía confocal y electrónica , los investigadores demostraron que la cepa viral EhV-86 utiliza un mecanismo de infección único, que difiere de otros virus de algas, y muestra una mayor similitud con las estrategias de entrada y salida observadas en virus de ADN bicatenario grande nucleocitoplasmático similares a animales (virus de ADN grande nucleocitoplasmático). EhV-86 se diferencia de sus contrapartes de algas en que su cápside está envuelta por una membrana lipídica. EhV-86 ingresa a las células por endocitosis (el proceso por el cual las partículas de alimento o líquido son absorbidas por la célula por una vesícula) o fusión directa (la envoltura viral se fusiona con la membrana del huésped). La entrada de EhV-86 por endocitosis da como resultado una capa de membrana adicional que rodea el genoma encapsulado de la cápside. Independientemente del mecanismo de entrada, la cápside ingresa intacta al citoplasma. Después de entrar en la célula, la cápside viral se desmonta y el ADN se libera en el citoplasma del huésped o directamente en el núcleo. El EhV-86 es único entre otros ficodnavirus, ya que codifica seis subunidades de la ARN polimerasa. Ni PBCV-1 ni ESV-1, por ejemplo, codifican componentes de la ARN polimerasa. [8] Los genes de la ARN polimerasa viral no se transcriben hasta al menos 2 horas después de la infección (pi). A las 3-4 pi, los viriones se ensamblan en el citoplasma, con la ayuda de la ATPasa (una proteína de empaquetamiento del ADN) y se transportan a la membrana plasmática donde se liberan del huésped a través de un mecanismo de gemación. En este mecanismo de gemación, el EhV-86 obtiene una membrana externa de la membrana del huésped. [12] El tamaño de la ráfaga varía de 400 a 1000 partículas por célula. [8]
Se ha identificado un grupo de genes productores de esfingolípidos en EhV-86. Los investigadores han descubierto que la producción de esfingolípidos virales producidos durante la etapa lítica está involucrada en la muerte celular programada en poblaciones de cocolitóforos. Se encontró una alta correlación entre la producción de glicoesfingolípidos (GSL) y la actividad de caspasa durante la etapa lítica en células infectadas. Las caspasas son una familia de enzimas proteasas involucradas en la muerte celular programada. Los investigadores también descubrieron que se requiere una concentración crítica de GSL (>0,06 mg/ml) para iniciar la lisis celular. Por lo tanto, los autores sugieren que la producción de GSL a una concentración crítica puede ser parte de un mecanismo de sincronización para el ciclo lítico. Los autores también sugieren que estas biomoléculas pueden ser capaces de inducir la muerte celular programada en otras células no afectadas, actuando así como una señal de terminación de la floración de algas. [27]
Se ha descrito que los cocolitovirus y los feovirus tienen estrategias de vida opuestas. El cocolitovirus posee una estrategia de vida aguda caracterizada por altas tasas de reproducción y mutación y una mayor dependencia de poblaciones hospedadoras densas para la transmisión. Los feovirus poseen una estrategia de vida persistente en la que la infección puede o no causar enfermedad, y el genoma se transmite de padres a hijos. [28]
Los feovirus infectan a las algas pardas Ectocarpales , que son un orden de algas pardas filamentosas. Uno de los feovirus más estudiados es el virus Ectocarpus siliculosus , más comúnmente conocido como EsV-1. [28] El virus EsV-1 solo infecta los gametos o esporas unicelulares de E. siliculosus . Las células vegetativas son inmunes a la infección, ya que están protegidas por una pared celular rígida. [29] Después de la infección, una copia del ADN viral se incorpora al genoma del huésped. Luego, el genoma viral EsV-1 se replica y los viriones se ensamblan en los esporangios o gametangios de las plantas infectadas. [30] Posteriormente, los virus se liberan a través de la lisis de las células reproductivas, estimulada por cambios en las condiciones ambientales, como un aumento de la temperatura. [31] En plantas sanas, los estímulos ambientales sincronizan la liberación de gametos y zoosporas en el agua circundante. [31] Las partículas virales libres pueden luego reinfectar gametos o esporas de plantas sanas que nadan libremente. Los gametos o esporas infectadas experimentan mitosis, formando plantas infectadas y todas las células de la planta progenie contienen ADN viral. Sin embargo, las partículas virales solo se producen en las células reproductivas de las algas, mientras que los virus permanecen latentes en las células vegetativas. En los esporofitos infectados , las células experimentan meiosis y producen esporas haploides. El genoma de EsV se transmite de manera mendeliana, donde la mitad de la progenie contiene ADN viral. A menudo, las algas de esporas infectadas son indistinguibles de las algas derivadas de esporas sanas, pero son parcial o totalmente incapaces de reproducirse. [29] [30]
Los clorovirus son los únicos virus caracterizados hasta ahora que infectan a las algas de agua dulce. [32] Los huéspedes de los clorovirus son las zooclorellas, que son algas verdes endosimbióticas comúnmente asociadas con los huéspedes Paramecium bursaria , el celentéreo Hydra viridis o el heliozoo Acanthocystis turfacea . [33] En el ciliado Paramecium bursaria , por ejemplo, las algas viven dentro de las células del huésped, proporcionando nutrientes a través de la fotosíntesis. Vivir dentro de las células del ciliado ofrece protección para las algas y un modo de transporte. Las zooclorellas son resistentes a la infección en su estado simbiótico. Cuando la relación entre las algas y el huésped se interrumpe, por ejemplo, a través del pastoreo de copépodos, se permite la infección por clorovirus. [34]
El ciclo de vida del clorovirus que infecta a Paramecium bursaria , conocido como PBCV-1, se ha estudiado en detalle [ cita requerida ] . La microscopía crioelectrónica y la reconstrucción 3D de la cápside viral muestran que hay una estructura larga de "espiga" que primero contacta con la pared celular y probablemente sirve para perforar la pared celular del huésped. El virus PBCV-1 es específico de su huésped y el reconocimiento está mediado por la interacción de las proteínas de la superficie del virus con los carbohidratos de la superficie de las algas. Después de la unión del virus a la pared celular del huésped, las enzimas glucolíticas unidas a la cápside descomponen la pared celular. La membrana viral probablemente se fusiona con la membrana del huésped, lo que permite que el ADN viral ingrese al citoplasma, dejando una cápside vacía en el exterior. Como PBCV-1 carece de un gen de ARN polimerasa, el virus debe usar la maquinaria de la célula huésped para producir ARN viral. Por lo tanto, el ADN viral se mueve rápidamente al núcleo donde se inicia la transcripción temprana 5 a 10 minutos después de la infección. A los pocos minutos de la infección, se produce la degradación cromosómica del huésped, lo que inhibe la transcripción del mismo. A los 20 minutos de la infección, la mayoría de los ARNm de la célula infectada son ARNm virales. Las proteínas traducidas de la etapa temprana de la transcripción participan en el inicio de la replicación del ADN viral, que ocurre entre 60 y 90 minutos después de la infección. La segunda fase de las proteínas se traduce en el citoplasma y el ensamblaje de las cápsides del virus comienza aproximadamente entre 2 y 3 horas después de la infección. Los viriones maduros se forman con la adición de ADN viral recién replicado del núcleo del huésped, probablemente facilitado por una ATPasa de empaquetamiento de ADN codificada por el virus. Aproximadamente entre 5 y 6 horas después de la infección por PBCV-1, el citoplasma se llena de viriones y la lisis se produce entre 6 y 8 horas después de la infección, liberándose aproximadamente 1000 partículas por célula. [32] [35]
El género Prymnesiovirus contiene actualmente solo una especie, conocida como virus Chrysochromulina brevifilum PW1 (CbV-PW1). CbV-PW1 infecta dos especies de fitoplancton marino, Chrysochromulina brevifilum y C. strobilus , pertenecientes al género Chrysochromulina . [36] [37] Según la base de datos AlgaeBase, actualmente hay 63 nombres de especies marinas y de agua dulce en el género, de los cuales 48 son reconocidos como nombres taxonómicamente aceptables. [38] Chrysochromulina es un género particularmente importante ya que puede comprender más del 50% de las células nanoplanctónicas fotosintéticas en el océano. [36]
Se sabe poco sobre el ciclo de vida del virus que infecta a estas especies planctónicas que contienen flagelados, Chrysochromulina brevifilum y C. strobilus . Suttle y Chan (1995) fueron los primeros en aislar virus que infectan a Prymnesiophytes o haptophytes. En este estudio, se prepararon secciones ultrafinas de virus dentro de Chyrsochromulina brevifilum y se observaron utilizando microscopía electrónica de transmisión. [36] Las micrografías electrónicas en la etapa temprana de la infección sugieren que la replicación del virus ocurre en el citoplasma dentro de un viroplasma . Un viroplasma es un área localizada en el citoplasma, o alrededor del núcleo de la célula, que sirve como una "fábrica de replicación viral". El viroplasma contiene componentes como material genético del virus, proteínas del huésped y ribosomas necesarios para la replicación. Los virosomas a menudo están rodeados por una membrana; se encontró que la membrana que rodea el virosoma contenido en las células infectadas en el estudio consistía en una matriz fibrilar. [36] Los viriones se liberan de las células infectadas tras la ruptura de los orgánulos y la lisis de la membrana de la célula huésped. Suttle y Chan (1995) contaron más de 320 virus en una sección ultrafina de una célula infectada. [36] Las estimaciones de los tamaños de ráfaga varían de 320 a 600 virus por célula. [39]
Los miembros del género Prasinovirus infectan pequeñas algas verdes unicelulares del orden Mamiellales , que se encuentran comúnmente en aguas marinas costeras. [40] Una especie del género Prasinovirus es el virus Micromonas pusilla SP1 (MpV-SP1), [41] que se aisló de una muestra de agua recolectada en San Diego. [42] El prasinovirus MpV-SP1 infecta a Micromonas pusilla , que es un picoeucariota marino fotosintético dominante. [43] y que infecta a Micromonas pusilla (UTEX 991, Plymouth 27). Los huéspedes comunes de prasinovirus incluyen miembros de los géneros Ostreococcus y Micromonas . Se han identificado tres especies potenciales de Ostreococcus y difieren según sus requisitos de luz. [44] Uno de los prasinovirus más estudiados, la cepa OtV5 cuyo genoma está completamente secuenciado, infecta a Ostreococcus tauri , el eucariota de vida libre más pequeño conocido actualmente. [45]
Los prasinovirus emplean una estrategia de replicación nucleocitoplasmática en la que los viriones se adhieren a la superficie de la célula huésped, seguida de una inyección de ADN en el citoplasma del huésped. [45] Los investigadores descubrieron que los virus OtV5 "vacíos", o virus con solo la cápside unida a la membrana del huésped, rara vez se veían en ninguna etapa de la infección, lo que sugiere que los viriones se desprenden de la membrana del huésped después de la inyección de su ADN. Los autores también descubrieron que una alta proporción de virus no se adhirieron a las células después de la inoculación y sugieren que la unión viral puede ser un paso limitante en la infección. El ADN viral luego se replica dentro del núcleo por la maquinaria de la célula huésped. Las partículas virales se ensamblan en el citoplasma, generalmente ocupando un espacio cerca de la cara interna del núcleo. Debido al tamaño extremadamente pequeño de las células de las algas, se encontró que el tamaño promedio de la explosión era de 25 partículas virales por célula. [45]
Recientemente se ha observado la producción viral sin lisis celular en células de O. tauri . Thomas et al. (2011) descubrieron que en células hospedadoras resistentes, el genoma viral se replicaba y los virus se liberaban mediante un mecanismo de gemación. [46] Esta baja tasa de liberación viral mediante gemación permite una supervivencia prolongada del hospedador y la progenie del virus, lo que da como resultado una coexistencia estable. [47]
El virus Ectocarpus siliculosus (EsV-1), perteneciente al género Phaeovirus , y el virus Paramecium bursaria chlorella (PBCV-1), perteneciente al género Chlorovirus , son dos virus muy estudiados, cuyos genomas han demostrado codificar muchas proteínas. Estas proteínas funcionan en la estabilidad del virus, la síntesis de ADN, la transcripción y otras interacciones importantes con el huésped. [ cita requerida ]
El PBCV-1 tiene una proteína de la cápside principal glicosilada de 54 kDa, que comprende aproximadamente el 40% de la proteína viral total. [12] A diferencia de la mayoría de las proteínas estructurales virales que están glicosiladas en el retículo endoplasmático (RE) y el aparato de Golgi por las glicosiltransferasas codificadas por el huésped , [48] el PBCV-1 glicosila su proteína de la cápside principal de forma independiente al codificar la mayoría de las enzimas necesarias para construir los oligosacáridos complejos, que luego se unen a la proteína de la cápside principal del PBCV-1 para formar la glicoproteína. Por lo tanto, la glicosilación de la proteína de la cápside principal del PBCV-1 ocurre independientemente del RE y el aparato de Golgi en las células huésped. [49]
La primera proteína viral conocida que funciona como un canal iónico selectivo de potasio se encontró en PBCV-1. [50] La proteína (llamada Kcv) consta de 94 aminoácidos y está codificada a partir de un pequeño marco de lectura abierto ( ORF ) (ORF A250R) en PBCV-1, que puede producir conductancia selectiva de potasio y sensible al voltaje en ovocitos de Xenopus . [50] La supuesta proteína PBCV-1 tiene un extremo N citoplasmático corto (12 aminoácidos) que contiene un sitio de consenso de la proteína quinasa C y tiene 2 dominios transmembrana. Las diferentes secuencias de aminoácidos y la falta de cola citoplasmática COOH-terminal hacen que la proteína Kcv sea diferente de otros canales de potasio. [50] [29]
EsV-1 codifica un ORF de 124 codones que tiene una similitud significativa de aminoácidos con PBCV-1 Kcv (41% de identidad de aminoácidos). [29] Sin embargo, la proteína EsV-1 tiene un extremo N-terminal más largo (35 aminoácidos) que contiene dos sitios de consenso de la proteína quinasa C y tiene tres dominios transmembrana. [29] Se desconoce si la proteína EsV-1 puede formar un canal funcional en células heterólogas. El genoma de EsV-1 también codifica varias proteínas con regiones ricas en aminoácidos hidrofóbicos que se asemejan a dominios transmembrana helicoidales. Entre estas proteínas, el dominio de entrada de la supuesta His-quinasa híbrida 186 y el ORF 188 se asemejan a proteínas de canal iónico. [45]
Tanto EsV-1 como PBCV-1 codifican ADN polimerasa que pertenece a la familia de ADN polimerasa-δ, y todos contienen un dominio exonucleasa 3'-5' de corrección de pruebas . [51] Además, tanto PBCV-1 como EsV-1 codifican una proteína de factor de procesividad de abrazadera deslizante (PCNA), que interactúa con proteínas involucradas en la replicación de ADN, así como proteínas involucradas en la reparación del ADN y el procesamiento posreplicativo (por ejemplo, metilasas de ADN y transposasas de ADN). [52]
El factor de replicación heteropentamérico C (RFC) es un complejo que es responsable de la carga dependiente de ATP de PCNA en el ADN; [53] [54] EsV-1 codifica cinco proteínas que pueden formar un complejo RFC. PBCV-1 codifica una sola proteína que se asemeja a la que se encuentra en el complejo RFC de Archae. [45] PBCV-1 también codifica otras proteínas involucradas en la replicación del ADN, incluyendo una ADN ligasa dependiente de ATP , [55] una ADN topoisomerasa tipo II y RNasa H. [ 29] Aunque tanto EsV-1 como PBCV-1 poseen genes para elementos esenciales del sistema de replicación eucariota, ninguno tiene genes replicativos completos, ya que todos carecen de genes para primasa. [12] [29]
Ni EsV-1 ni PBCV-1 codifican una ARN polimerasa completa , pero producen varias proteínas similares a factores de transcripción para ayudar al sistema de transcripción del huésped. [ cita requerida ]
EsV-1 codifica dos polipéptidos pequeños (ORF 193 y ORF 196) para la regulación transcripcional; las proteínas se asemejan al dominio α/β/α de la subunidad TFIID -18. [45] El complejo TFIID es necesario para la transcripción de eucariotas, ya que se une a la caja TATA en el promotor central del gen para iniciar el ensamblaje de la ARN polimerasa. Además, los polipéptidos se asemejan a los dominios SET, BTB/POZ (es decir, Broad Complex, Tramtrack y Bric-a-brac/poxvirus y zinc finger) (ORF 40) y BAF60b (ORF 129) también están codificados por ESV-1 para regular la remodelación de la cromatina y la represión de la transcripción. [45] [12] [56]
Se han encontrado cuatro proteínas similares a factores de transcripción en PBSV-1, incluyendo TFIIB (A107L), TFIID (A552R), TFIIS (A125L) y un factor de transcripción tipo VLTF-2 (A482R). [29] Además, PBCV-1 también codifica dos enzimas involucradas en la formación de una estructura de tapa de ARNm, una ARN trifosfatasa [57] y una ARN guanililtransferasa . [58] Las enzimas de PBCV-1 están más estrechamente relacionadas con las enzimas de levadura que con las enzimas de tapa de ARN multifuncionales de poxvirus según su tamaño, secuencia de aminoácidos y propiedades bioquímicas. [59] [58] PBCV-1 también codifica RNasa III , que está involucrada en el procesamiento de ARNm del virus. [29]
Para suministrar desoxinucleótidos para la producción viral en las células huésped de baja proliferación, los virus de ADN grandes poseen genes para codificar las enzimas de síntesis de desoxinucleótidos por sí mismos. [29] Se han encontrado trece enzimas metabólicas de nucleótidos en PBCV-1, dos de las cuales incluyen dUTP pirofosfatasa y dCMP deaminasa , que pueden producir dUMP (es decir, el sustrato para la timidilato sintetasa). [60] En comparación, EsV-1 solo codifica una ATPasa (ORF 26), así como ambas subunidades de la ribonucleótido reductasa (ORF 128 y 180), que es una enzima clave en la síntesis de desoxinucleótidos. [45]
También se encontraron otras enzimas como metiltransferasas , endonucleasas de restricción de ADN e integrasas en PBCV-1. [12] [29] PBCV-1 también codifica una proteína de 187 aminoácidos que se asemeja a la SOD Cu-Zn con todos los residuos de aminoácidos conservados para la unión de cobre y zinc, que pueden descomponer el superóxido acumulado rápidamente en las células huésped durante la infección, beneficiando así la replicación del virus. [61]

Heterosigma akashiwo forma floraciones densas y dañinas en aguas templadas y subárticas, que se producen en densidades de hasta 5 × 10 6 células/ml. [62] Estas floraciones de algas pueden ser extremadamente dañinas para la vida acuática, causando la mortalidad en peces silvestres y cultivados, como el salmón, el jurel y el besugo. [5] La gravedad y duración de estas floraciones varía de un año a otro, y el daño a la acuicultura causado por H. akashiwo ha ido en aumento. En 1989, una floración de algas nocivas frente a las costas de Nueva Zelanda provocó la pérdida de salmón Chinook por valor de diecisiete millones de dólares neozelandeses. En 1995 y 1997, en las aguas costeras japonesas de la bahía de Kagoshimo, murieron peces por valor de 1.090 millones y 327 millones de yenes, respectivamente. [5]
Se ha demostrado que el virus HaV, que infecta a H. akashiwo , es un factor en la terminación de la floración. Suttle et al. (1990) sugirieron que la infección viral de las algas podría tener un papel en la regulación de las densidades de población de las comunidades de fitoplancton, teniendo así papeles significativos en su dinámica en los océanos. [63] Estudios anteriores, como el estudio de Nagasaki et al. (1993), exploraron la dinámica entre HaV y H. akashiwo . Se obtuvieron muestras de algas en las etapas medias o finales de una marea roja en la bahía de Hiroshima , Japón. Utilizando microscopía electrónica de transmisión , Nagaski et al. identificaron el virus HaV en y alrededor del área nuclear de las células de H. akashiwo . [63] Un estudio realizado por Nagaski et al. (1994) proporcionó más apoyo al papel del virus HaV en la terminación de la floración. Nagaski et al. (1994) encontraron que la proporción de células que contenían virus aumentaba rápidamente antes de la terminación de la marea roja; No se detectaron células que contuvieran virus tres días antes de la terminación de la marea roja y la muestra recolectada el último día reveló una alta frecuencia (11,5%) de células que contenían virus. [64]
Estudios posteriores de Tarutani et al. (2000) también encontraron una asociación entre una disminución en la densidad celular de H. akashiwo con un aumento en la abundancia de HaV. Los investigadores encontraron que HaV no solo juega un papel importante en el control de la biomasa, sino que también influye en la composición clonal o las características de las células de H. akashiwo . Los investigadores encontraron que la mayoría de los aislamientos después de la terminación de la floración eran resistentes a los aislamientos clonales de HaV, mientras que durante la formación de la floración las células resistentes eran un componente menor. Los autores sugieren que la infección viral, durante el período de terminación de la floración, influye en las propiedades de las células dominantes en las poblaciones de H. akashiwo . [65] La presión selectiva ejercida por los virus en la etapa posterior de la infección puede promover la diversidad genética, lo que permite que la población de H. akashiwo prospere después de la terminación de la floración. [ cita requerida ]
Como se mencionó, las floraciones de H. akashiwo son perjudiciales para las poblaciones de peces en aguas templadas y subárticas, y siguen planteando graves amenazas para la acuicultura. Nagasaki et al. (1999) examinaron las características de crecimiento de HaV y sugirieron que HaV podría usarse como un agente microbiano contra las mareas rojas de H. akashiwo . Las ventajas de usar HaV es que infecta específicamente a H. akashiwo incluso cuando otros microorganismos están presentes. Además, tiene una alta tasa de crecimiento y se puede producir a bajo costo. El uso de HaV como agente microbiano es una solución prometedora para eliminar las mareas rojas y proteger la pesca y la vida marina, pero como concluyeron los autores, los efectos de varios clones de HaV en las poblaciones de H. akashiwo deben explorarse con mayor detalle antes de que el virus se use para aplicaciones a gran escala. [5]

El cocolitovirus (EhV) infecta al cocolitóforo Emiliania huxleyi ( E. huxleyi ). Los cocolitóforos son haptofitos marinos que están rodeados de placas microscópicas hechas de carbonato de calcio . [66] Viven en las capas superiores de los océanos del mundo y representan el tercer grupo más abundante de fitoplancton, que contiene alrededor de 300 especies. [67] E. huxleyi es reconocido como el más destacado y ecológicamente importante de los cocolitóforos. E. huxleyi tiene una distribución global desde los trópicos hasta las aguas subárticas y ocasionalmente forma floraciones densas que pueden cubrir cientos de miles de kilómetros cuadrados. [67] Estos billones de cocolitóforos producidos, luego mueren y se hunden hasta el fondo de los océanos, lo que contribuye a la formación de sedimentos, y son los mayores productores de calcita en los océanos. [66] Por lo tanto, los cocolitos tienen un papel importante en la fijación global de carbono y el ciclo del carbono, así como en el ciclo del azufre. [67] Con el tiempo, los cocolitóforos han dado forma a las características geológicas de nuestro planeta. Por ejemplo, los acantilados blancos de Dover se formaron a partir de tiza blanca , o carbonato de calcio producido por los cocolitóforos durante millones de años. [ cita requerida ]
Las floraciones de cocolitóforos no suelen ser dañinas para la vida marina en el océano. Como estos organismos prosperan en condiciones pobres en nutrientes, los cocolitóforos ofrecen una fuente de nutrición para peces pequeños y zooplancton . [66] Se ha demostrado que los virus E. huxylei (EhV) están vinculados a la terminación de estas floraciones. La etapa de terminación de la floración se indica mediante un cambio de color en el agua. Cuando grandes cantidades de cocolitos (capa de carbonato que rodea a E. huxylei ) se desprenden de las células de E. huxylei debido a la muerte celular o lisis, el agua se vuelve blanca o turquesa. En áreas de terminación densa de floración, el color blanco es reflectante y se puede ver en imágenes satelitales. [67] Wilson et al. (2002) utilizaron citometría de flujo analítica para medir la abundancia de virus en diferentes ubicaciones dentro y alrededor del área de floración. Los investigadores encontraron que las concentraciones de virus eran más altas dentro del "área de alta reflectancia", lo que sugiere que la lisis inducida por virus de las células de E. huxleyi resultó en el desprendimiento de cocolitos. [68] Otros estudios de Martínez et al. (2007) y Bratbak et al. (1993) encontraron concentraciones más altas de virus EhV a medida que la floración de E. huxleyi disminuía, lo que indica que la infección viral lítica fue la principal causa de la terminación de la floración. [69] [70] Por lo tanto, los virus EhV tienen papeles importantes en la regulación de la producción de biomasa en entornos marinos y la sucesión ecológica. Esta regulación de las poblaciones de cocolitóforos por los virus EhV tiene, por lo tanto, efectos significativos en los ciclos biogeoquímicos , particularmente el ciclo del carbono . [ cita requerida ]

Uno de los feovirus mejor estudiados, el EsV-1, infecta a las pequeñas algas filamentosas marrones E. siliculosus , que tienen una distribución cosmopolita (se encuentran en la mayoría de los océanos del mundo). [29] Los Ectocarpales están estrechamente relacionados con el grupo de algas pardas, las Laminariales , que son el grupo de algas pardas económicamente más importante y tienen una amplia gama de aplicaciones en la industria cosmética y alimentaria. [71]
Muller et al. (1990) fueron de los primeros en explorar las causas de los defectos del gametangio en E. siliculosus originario de Nueva Zelanda. Los investigadores identificaron células reproductivas de E. siliculosus llenas de partículas hexagonales que luego se liberaron en el medio de cultivo cuando las células estallaron. Después de la liberación de estas partículas, los esporofitos se infectaron, lo que se mostró por síntomas patológicos, lo que sugiere que las partículas son virus. [72] Estos estudios permitieron la evaluación del potencial de infección de los virus de E. siliculosus . Utilizando la amplificación por PCR de un fragmento de gen viral, Muller et al. (2005) monitorearon los niveles de infección por patógenos en muestras de Ectocarpus de la isla de Gran Canaria, el Atlántico Norte y el sur de Chile. Los investigadores encontraron altos niveles de prevalencia de patógenos; 40-100% de los especímenes de Ectocarpus contenían ADN viral. [73] Estimaciones similares han sido dadas por Sengco et al. (1996) quienes estimaron que al menos el 50% de las plantas de Ectocarpus en el mundo contienen ADN viral. [74] Esta alta frecuencia de infección viral entre las plantas de Ectocarpus distribuidas globalmente tiene implicaciones ecológicas. La infección viral por EsV-1 en plantas de E. siliculosus , como se mencionó, limita el éxito reproductivo de las plantas infectadas. Por lo tanto, el virus EsV-1 juega un papel clave en la regulación de las poblaciones de E. siliculosus , teniendo efectos adicionales en la dinámica del ecosistema local. [ cita requerida ]
.jpg/440px-Stichotricha_secunda_-_400x_(14974779356).jpg)
Los miembros del género Chlorovirus se encuentran en fuentes de agua dulce en todo el mundo e infectan a los simbiontes de algas verdes zoochlorellae. Existe una falta de información sobre el papel que juegan los clorovirus en la ecología de agua dulce. [75] A pesar de esto, los clorovirus se encuentran en aguas nativas en 1-100 unidades formadoras de placa (PFU) / ml y se han obtenido mediciones de hasta 100.000 PFU / ml de agua nativa. [8] Una unidad formadora de placa es el número de partículas capaces de formar estructuras visibles dentro de un cultivo celular, conocidas como placas. [ cita requerida ] Las abundancias de clorovirus varían con la estación, y las mayores abundancias se dan en la primavera. [8] Los clorovirus, como PBCV-1, juegan un papel en la regulación de las poblaciones hospedadoras de zoochlorella. Como se mencionó anteriormente, la infección de zoochlorella ocurre solo cuando se interrumpe la relación simbiótica con su hospedador. La infección de las algas durante esta etapa de independencia entre el hospedador y las algas impedirá que se restablezca la relación entre el hospedador y las algas, lo que reducirá la capacidad de supervivencia de los hospedadores endosimbióticos de las zooclorellas, como Paramecium bursaria. Por lo tanto, los clorovirus desempeñan un papel importante en los ecosistemas de agua dulce , no solo regulando las poblaciones de su hospedador, las zooclorellas, sino también regulando, hasta cierto punto, las poblaciones de hospedadores de zooclorellas. Los clorovirus y los virus en general causan la muerte y lisis de sus hospedadores, liberando carbono orgánico disuelto, nitrógeno y fósforo en el agua. Estos nutrientes pueden ser absorbidos por las bacterias, contribuyendo así al ciclo microbiano. La liberación de materiales orgánicos disueltos permite el crecimiento bacteriano, y las bacterias son una fuente importante de alimento para los organismos en niveles tróficos superiores. En consecuencia, los clorovirus tienen efectos significativos en los flujos de carbono y nutrientes, lo que influye en la dinámica de los ecosistemas de agua dulce. [6]
El Prymnesiovirus, CbV-PW1, como se mencionó, infecta al género de algas Chyrsochromulina . Chyrsochromulina , que se encuentra en aguas dulces y marinas globales, ocasionalmente forma floraciones densas que pueden producir toxinas dañinas, lo que tiene efectos negativos en las pesquerías. [36] Una especie particularmente tóxica llamada C. polylepis ha causado enormes daños a las pesquerías comerciales en Escandinavia. En 1988, esta floración causó una pérdida de 500 toneladas de peces enjaulados, por un valor de 5 millones de dólares estadounidenses. [76] Dado que Chyrsochromulina es una especie ampliamente distribuida y tiene una importancia ecológica significativa, es probable que la infección viral y la lisis de los miembros del género tengan impactos significativos en los ciclos biogeoquímicos, como el reciclaje de nutrientes en entornos acuáticos. Suttle y Chan sugieren que la presencia de virus debería tener un fuerte efecto regulador en las poblaciones de Chyrsochromulina , evitando así la formación de floraciones o permitiendo la terminación de las mismas, lo que explica por qué las floraciones persistentes son un fenómeno inusual en la naturaleza. [36]
Un prasinovirus comúnmente estudiado, OtV5, como se mencionó, infecta al eucariota más pequeño conocido actualmente, Ostreococcus tauri . O. tauri tiene aproximadamente 0,8 micrómetros de diámetro y está dentro de la fracción picosize (0,2-2 micrómetros). Los picoeucariotas, como Ostreococcus tauri , están ampliamente distribuidos y contribuyen significativamente a la biomasa microbiana y la productividad primaria total. En entornos oligotróficos, el picofitoplancton marino representa hasta el 90% de la biomasa autótrofa y, por lo tanto, son una fuente de alimento importante para los protistos nanoplanctónicos y fagotróficos. [77] Como los picoeucariotas sirven como base para las redes alimentarias microbianas marinas, son intrínsecos a la supervivencia de los niveles tróficos superiores. Ostreococcus tauri tiene una tasa de crecimiento rápida y se han observado floraciones densas en las costas de Long Island y California. [77] Se encontró que las muestras recolectadas de la bahía de Long Island contenían muchas partículas similares a virus, una causa probable de la disminución de la floración. [78] A pesar de la gran abundancia de picoeucariotas, estos organismos unicelulares son superados en número por los virus en una proporción de aproximadamente diez a uno. [79] Los virus como OtV5, juegan un papel importante en la regulación de las poblaciones de fitoplancton y, a través de la lisis de las células, contribuyen al reciclaje de nutrientes hacia otros microorganismos, también conocido como derivación viral . [80]
Como se mencionó, el prasinovirus MpV-SP1 infecta a Micromonas pusilla , que es un componente importante del picofitoplancton de los océanos del mundo. M. pusilla vive en ecosistemas marinos desde tropicales hasta polares. [81] Cottrell y Suttle (1995) encontraron que entre el 2 y el 10 % de la población de M. pusilla en un entorno costero se lizaba por día, con un promedio del 4,4 %. [43] Evans et al. (2003) han proporcionado estimaciones más altas, quienes sugieren que los virus de M. pusilla pueden lisar hasta el 25 % de la población de Micromonas por día. [82] Esto sugiere que los virus son responsables de una cantidad moderada de mortalidad en las poblaciones de M. pusilla . [43] A mayor escala, la infección viral de M. pusilla es responsable del reciclaje de nutrientes y energía en las redes alimentarias acuáticas, lo que aún está por cuantificar. [ cita requerida ]
Hasta hace poco se creía que los ficodnavirus infectaban exclusivamente a las especies de algas. Recientemente, se aisló ADN homólogo al virus 1 del clorovirus Acanthocystis turfacea (ATCV-1) de las superficies de la mucosa nasofaríngea humana. La presencia de ATCV-1 en el microbioma humano se asoció con un rendimiento disminuido en las evaluaciones cognitivas. La inoculación de ATCV-1 en animales de experimentación se asoció con un rendimiento disminuido en la memoria y la activación sensoriomotora, así como con una expresión alterada de genes en el hipocampo relacionados con la plasticidad sináptica , el aprendizaje, la formación de la memoria y la respuesta inmunitaria viral. [3]