Las topoisomerasas de ADN (o topoisomerasas ) son enzimas que catalizan cambios en el estado topológico del ADN , interconvirtiendo formas relajadas y superenrolladas, especies enlazadas (catenadas) y no enlazadas, y ADN anudado y no anudado. [1] [2] Los problemas topológicos en el ADN surgen debido a la naturaleza entrelazada de su estructura de doble hélice, que, por ejemplo, puede conducir al enrollamiento excesivo del dúplex de ADN durante la replicación y transcripción del ADN . Si no se modifica, esta torsión eventualmente detendría las polimerasas de ADN o ARN involucradas en estos procesos de continuar a lo largo de la hélice de ADN. Un segundo desafío topológico resulta del enlace o enredo del ADN durante la replicación. Si no se resuelven, los enlaces entre el ADN replicado impedirán la división celular. Las topoisomerasas de ADN previenen y corrigen este tipo de problemas topológicos. Lo hacen uniéndose al ADN y cortando la cadena principal de azúcar y fosfato de una de las cadenas de ADN (topoisomerasas de tipo I) o de ambas (topoisomerasas de tipo II). Esta ruptura transitoria permite desenredar o desenrollar el ADN y, al final de estos procesos, la cadena principal del ADN se vuelve a sellar. Dado que la composición química general y la conectividad del ADN no cambian, el sustrato y el producto del ADN son isómeros químicos, que difieren solo en su topología.
La primera topoisomerasa de ADN fue descubierta en bacterias por James C. Wang en 1971 y fue inicialmente llamada proteína ω (omega); [3] ahora se llama topoisomerasa I de Escherichia coli ( E. coli ) (topo I) y es un representante de la familia de enzimas de tipo IA. Posteriormente, James Champoux y Renato Dulbecco encontraron una actividad similar en células eucariotas (hígado de rata) ; [4] la enzima responsable, la topo I eucariota, tiene un mecanismo distinto y es representativa de la familia de tipo IB. La primera topoisomerasa de tipo II que se descubrió fue la ADN girasa de bacterias, por Martin Gellert y colaboradores en 1976, [5] y también caracterizada por Nicholas Cozzarelli y colaboradores. [6] La ADN girasa cataliza la introducción de superenrollamientos negativos en el ADN y es la única enzima de tipo II que hace esto, todas las demás catalizan la relajación del ADN. Las enzimas de tipo II se diferencian mecánicamente de las de tipo I porque dependen del ATP y escinden transitoriamente ambas cadenas de ADN en lugar de solo una. Las topoisomerasas de tipo II se identificaron posteriormente en virus bacterianos y eucariotas. [7] [8] [9] Los códigos de las topoisomerasas EC son los siguientes: independiente del ATP (tipo I), EC 5.6.2.1; dependiente del ATP (tipo II): EC 5.6.2.2. La excepción entre las topoisomerasas de tipo I, la girasa inversa, que contiene un dominio de helicasa (EC 3.6.4.12) e introduce un superenrollamiento positivo de manera dependiente del ATP. Por lo tanto, es la única topoisomerasa de tipo I clasificada como EC 5.6.2.2 (Tabla 1).
La estructura de doble hélice del ADN implica el entrelazamiento de las dos cadenas de polinucleótidos una alrededor de la otra, lo que potencialmente da lugar a problemas topológicos. La topología del ADN se refiere al cruce de las dos cadenas de ADN que altera el giro de la doble hélice y da lugar a conformaciones terciarias del ADN, como superenrollamientos, nudos y catenanos. [10] Los posibles problemas topológicos asociados con la estructura de doble hélice del ADN se reconocieron poco después de que su estructura fuera elucidada por primera vez en 1953 por James Watson, Francis Crick y Rosalind Franklin [11] [12] [13] y desarrollada aún más por el trabajo de Max Delbruck y John Cairns. [14] [15] El ADN bicatenario circular cerrado se puede describir mediante 3 parámetros: número de enlace (Lk), giro (Tw) y contorsión (Wr) (Fig. 1). Donde Lk se refiere al número de veces que las dos hebras están unidas, Tw se refiere al número de vueltas helicoidales en el ADN, medido en relación con el eje helicoidal, y Wr cuantifica el enrollamiento de la trayectoria de la hélice de ADN en el espacio y a menudo se equipara con el "superenrollamiento".

Los 3 parámetros están relacionados de la siguiente manera: Lk = Tw + Wr. Esta es una identidad matemática obtenida originalmente por Călugăreanu en 1959 [16] y se conoce como el teorema de Călugăreanu, o Călugăreanu–White–Fuller. [17] [18] Lk no se puede alterar sin romper una o ambas hebras de la hélice; Tw y Wr son interconvertibles y dependen de las condiciones de la solución. El superenrollamiento es un término vernáculo para el ADN con una diferencia de enlace distinta de cero, más correctamente denominada diferencia de enlace específica (σ = ΔLk/Lk 0 , donde Lk 0 es el número de enlace medio del círculo de ADN relajado). Se dice que el ADN está superenrollado positivamente si Lk es mayor que Lk 0 para el estado relajado (Lk-Lk o = ΔLk, ΔLk>0); Esto significa que Tw y/o Wr aumentan en relación con la molécula relajada. Por el contrario, el ADN está superenrollado negativamente si el Lk de la molécula es menor que el Lk 0 (ΔLk<0).
Las consecuencias de las perturbaciones topológicas en el ADN se ejemplifican con la replicación del ADN durante la cual las hebras del dúplex se separan; esta separación conduce a la formación de superenrollamientos positivos (sobreenrollamiento o sobretorsión del ADN) por delante de la horquilla de replicación y al entrelazamiento de las hebras hijas (precatenanes) por detrás [10] [19] (Fig. 2). Si los superenrollamientos positivos no se relajan, se impide la progresión de la horquilla de replicación, mientras que la imposibilidad de desvincular las hebras hijas impide la segregación del genoma, que es necesaria para la división celular. [20] La transcripción por la ARN polimerasa también genera un superenrollamiento positivo por delante y un superenrollamiento negativo por detrás del complejo transcripcional (Fig. 2). Este efecto se conoce como el modelo de dominio superenrollado gemelo, como lo describieron Leroy Liu y James Wang en 1987. [21] Estas perturbaciones topológicas deben resolverse para que el metabolismo del ADN continúe, lo que permite que la célula replique, transcriba y divida eficientemente el genoma para permitir la división celular y la vitalidad. Los nudos en el ADN se pueden encontrar en bacteriófagos y como productos de reacciones de recombinación. [10] En general, los nudos en el ADN son perjudiciales y deben eliminarse (por topoisomerasas). Los catenanos de ADN se forman tras la replicación de moléculas circulares y deben resolverse por topoisomerasas o recombinasas para permitir la separación adecuada de las moléculas hijas durante la división celular. Además de los aspectos perjudiciales de la topología del ADN que requieren resolución, también hay aspectos beneficiosos. Por ejemplo, la replicación del plásmido requiere un superenrollamiento negativo del origen, lo que facilita la fusión local y expone el ADN monocatenario necesario para iniciar la replicación. De manera similar, el inicio de la replicación desde el origen bacteriano principal oriC también requiere un superenrollamiento negativo. [22] [23] Además, la compactación del genoma de E. coli se logra en parte mediante superenrollamiento negativo.

Las topoisomerasas del ADN son enzimas que han evolucionado para resolver problemas topológicos en el ADN (Tabla 2). [10] Lo hacen mediante la rotura transitoria de una o ambas cadenas de ADN. Esto ha llevado a la clasificación de las topoisomerasas en dos tipos: tipo I, que catalizan reacciones que implican roturas transitorias de una sola cadena, y tipo II, que catalizan reacciones que implican roturas transitorias de doble cadena (Fig. 3; Tabla 2). Existen subtipos dentro de estas clasificaciones.
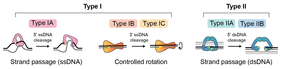
Estas enzimas catalizan cambios en la topología del ADN a través de rupturas transitorias de la cadena simple del ADN. Las reacciones pueden ocurrir en sustratos de ADN tanto de cadena simple como de cadena doble y pueden proceder a través de un mecanismo de "giro" o "paso de cadena" (Fig. 3). La gama de reacciones incluye: relajación de la superenrollación del ADN, desanudamiento de círculos de cadena simple y decatenación, siempre que al menos un socio tenga una región de cadena simple. En el caso de la enzima arqueal, la girasa inversa, es posible la superenrollación positiva del ADN. [24]
Los de tipo IA son monoméricos y se unen a segmentos monocatenarios de ADN. Introducen una ruptura transitoria de la cadena sencilla a través de la formación de un enlace tirosil-fosfato entre una tirosina en la enzima y un 5'-fosfato en el ADN. El segmento de ADN en el que se produce la ruptura se denomina "puerta" o segmento G, y su escisión permite el paso de otro segmento de ADN, el "transporte" o segmento T, en un proceso de "paso de cadena". [25] A esto le sigue la ligadura del segmento G. Para que se produzca el paso de cadena, el topo IA debe sufrir un cambio conformacional para abrir la puerta del ADN y permitir la transferencia del segmento T. Durante una reacción de relajación del ADN, este proceso cambia el número de enlace del ADN en +/-1 (Fig. 4). Entre los ejemplos de topoisomerasas de tipo IA se incluyen las topoisomerasas procariotas I y III, las topoisomerasas eucariotas IIIα y IIIβ y la enzima arqueal girasa inversa. La girasa inversa, que se encuentra en las arqueas termófilas, comprende una topoisomerasa de tipo IA acoplada a una helicasa y es la única enzima conocida que puede introducir superenrollamientos positivos en el ADN. [24] El gen que codifica la girasa inversa también se encuentra en algunos grupos de bacterias termófilas, a donde probablemente se transfirió mediante transferencia horizontal de genes desde las arqueas. [26]

Las topoisomerasas de tipo IB catalizan reacciones que implican roturas transitorias de una sola hebra del ADN mediante la formación de un enlace tirosil-fosfato entre una tirosina de la enzima y un fosfato 3' del ADN. En lugar de utilizar un mecanismo de paso de hebra, estas enzimas funcionan mediante un "giro" o "rotación controlada" de la hebra escindida alrededor de la hebra intacta. [27] Este mecanismo de rotación controlada se describió por primera vez para Vaccinia topo I [27] [28] y permite la rotación del ADN del extremo libre alrededor de la hebra intacta, a una velocidad controlada por la "fricción" dentro de la cavidad de la enzima, antes de que la muesca se vuelva a ligar (Fig. 3). Esto da como resultado un cambio variable del número de enlaces por cada evento de escisión y religación. Este mecanismo es distinto del de las enzimas de tipo IA, y los dos grupos de enzimas no están relacionados estructural y evolutivamente. Los ejemplos de topoisomerasas de tipo IB incluyen la topo I nuclear y mitocondrial eucariota, además de la topo I viral , aunque se han identificado en los tres dominios de la vida.
Las topoisomerasas de tipo IC comparten un mecanismo similar al de las enzimas de tipo IB, pero son estructuralmente distintas. El único representante es la topo V, que se encuentra en el hipertermófilo Methanopyrus kandleri . [29]
Las topoisomerasas de tipo II catalizan cambios en la topología del ADN a través de roturas transitorias de la doble cadena del ADN. Las reacciones ocurren en sustratos de ADN de doble cadena y proceden a través de un mecanismo de paso de cadena (Fig. 5). La gama de reacciones incluye relajación del ADN, superenrollamiento del ADN, desanudamiento y decatenación. Mientras que todas las topoisomerasas de tipo II pueden catalizar la relajación del ADN, la girasa, una topoisomerasa bacteriana arquetípica, también puede introducir superenrollamientos negativos. A diferencia de las topoisomerasas de tipo I que son generalmente monoméricas, las topoisomerasas de tipo II son homodímeras o heterotetrámeras. Se clasifican en dos subtipos según consideraciones evolutivas, estructurales y mecanísticas. El mecanismo general de paso de cadena para las topoisomerasas de tipo II comienza con la unión de un dúplex de ADN, denominado segmento de compuerta (segmento G), en la compuerta del ADN. Otro dúplex, denominado segmento de transporte (segmento T), es capturado por una pinza operada por ATP y pasa a través de una ruptura transitoria en el segmento G, que involucra enlaces de fosfotirosina 5ʹ en ambas cadenas, antes de ser liberado a través de la compuerta C y el segmento G se vuelve a ligar (Fig. 5). La renovación enzimática requiere la unión e hidrólisis del ATP.

Las topoisomerasas de tipo IIA catalizan roturas transitorias de doble cadena en el ADN a través de la formación de enlaces tirosil-fosfato entre tirosinas en la enzima (una en cada subunidad) y 5'-fosfatos escalonados por 4 bases en cadenas de ADN opuestas. La reacción de paso de cadena puede ser intra o intermolecular (Fig. 5), lo que permite cambios en el superenrollamiento y el anudamiento, o desvinculación, respectivamente. Este proceso cambia el número de enlaces del ADN en +/-2. Los ejemplos de topoisomerasas de tipo IIA incluyen topo IIα y topo IIβ eucariotas , además de la girasa bacteriana y la topo IV. La girasa de ADN se ajusta al mismo mecanismo de paso de doble cadena que otras enzimas de tipo II, pero tiene características únicas relacionadas con su capacidad para introducir superenrollamientos negativos en el ADN. El segmento G es parte de un fragmento de ADN mucho más largo (>100 pb) que se enrolla alrededor de la enzima, un brazo del cual forma el segmento T que pasa a través de la rotura de doble cadena (Fig. 5). En el caso de la girasa, una cantidad sustancial de la energía libre de la hidrólisis del ATP se transduce en tensión torsional en el ADN, es decir, el superenrollamiento es un proceso que requiere energía. [30] Además, en ausencia de ATP, la girasa puede eliminar los superenrollamientos negativos en una reacción de relajación del ADN más lenta.
Las enzimas de tipo IIB también catalizan roturas transitorias de doble cadena a través de la formación de enlaces tirosil-fosfato entre las tirosinas de la enzima y los 5′-fosfatos en cadenas opuestas del ADN, pero en el caso de las enzimas IIB las roturas de doble cadena tienen un escalonamiento de 2 bases. Las enzimas de tipo IIB muestran diferencias estructurales importantes, pero están relacionadas evolutivamente con las enzimas de tipo IIA. Estas diferencias incluyen la falta de una de las "puertas" de las proteínas (la puerta C) (Fig. 5). Se encontraron originalmente en las arqueas, pero también se han encontrado en eucariotas y, en particular, en plantas; los ejemplos incluyen topo VI y topo VIII. Topo VI es la enzima mejor estudiada de este subtipo y se cree que es una decatenasa preferencial. [31]
Para los no especialistas, quizás el aspecto más importante de las topoisomerasas es su papel como dianas farmacológicas tanto para la quimioterapia antibacteriana como para la anticancerígena; varios fármacos antibacterianos y anticancerígenos dirigidos por las topoisomerasas están incluidos en la Lista Modelo de Medicamentos Esenciales de la Organización Mundial de la Salud de 2019. La razón de esta prominencia es que sus reacciones se producen a través de roturas transitorias en el ADN, que, si se estabilizan mediante la unión del fármaco, pueden provocar la muerte celular debido a la generación de roturas tóxicas mono o bicatenarias en el ADN genómico. La mayoría de los fármacos dirigidos a las topoisomerasas actúan de esta manera, es decir, estabilizan el intermediario de escisión covalente enzima-ADN. [32] [33] [34]
Aunque los topos de tipo I, como el topo I bacteriano, son objetivos antibióticos viables, [35] actualmente no existen compuestos en uso clínico que tengan como objetivo estas enzimas. Sin embargo, las enzimas de tipo II, la ADN girasa y la ADN topoisomerasa IV, han tenido un enorme éxito como objetivos para los antibióticos fluoroquinolónicos ampliamente utilizados (Fig. 6).
Los compuestos antibacterianos de quinolona se desarrollaron por primera vez en la década de 1960 y se han utilizado clínicamente desde la década de 1980. [36] Los derivados de FQ, como ciprofloxacino, levofloxacino y moxifloxacino (Fig. 6) han tenido un gran éxito. Estos compuestos funcionan al interactuar con su diana (girasa o topo IV) y el ADN en el sitio de escisión para estabilizar el intermediario de escisión covalente ADN-proteína. Específicamente, se intercalan en el ADN y evitan el paso de religación del ADN de la reacción de la topoisomerasa (Fig. 5). Este es un mecanismo de inhibición altamente efectivo que también es utilizado por varios medicamentos contra el cáncer dirigidos contra la topoisomerasa. A pesar de su éxito espectacular, la resistencia a las FQ es un problema grave. [36] Una variedad de otros compuestos, como las quinazolindionas y las imidazolpirazinonas, [37] funcionan de manera similar y se espera que algunos de ellos reemplacen a las FQ en el futuro.
Las aminocumarinas (Fig. 6), como la novobiocina, la clorobiocina y la cumermicina A 1 , son productos naturales de Streptomyces que inhiben la reacción ATPasa de la girasa y la topo IV. [37] Aunque pueden ser muy potentes contra su objetivo, sufren problemas de permeabilidad y toxicidad y, por lo tanto, no han disfrutado del nivel de éxito clínico de las FQ.
Existen varios inhibidores proteicos de la girasa, incluidas las toxinas bacterianas CcdB, MccB17 y ParE, [38] [39] [40] que estabilizan el complejo de escisión, de manera similar a las FQ. Aunque estas proteínas no son viables como antibacterianos, su modo de acción podría inspirar el desarrollo de nuevos compuestos antibacterianos. Otros inhibidores proteicos de la girasa impiden la unión del ADN por la topoisomerasa en lugar de estabilizar los complejos de escisión. Estos incluyen YacG [41] y proteínas de repetición de pentapéptidos, como QnrB1 y MfpA; [42] [43] estos inhibidores proteicos también confieren resistencia a las fluoroquinolonas.

Tanto la topo I como la topo II humana (tanto las isoformas α como β) pueden ser el objetivo de la quimioterapia contra el cáncer (Fig. 7). [32] [33] [44] [45] [46] [47] La mayoría de estos compuestos actúan de manera similar a las FQ, es decir, estabilizando el complejo de escisión covalente proteína-ADN; por esto se les conoce como venenos de la topoisomerasa, distintos de los inhibidores catalíticos. [33] [34] [48] Varios inhibidores de la topoisomerasa humana están incluidos en la Lista de medicamentos esenciales de la Organización Mundial de la Salud .
La camptotecina (Fig. 7), derivada originalmente del árbol Camptotheca acuminata , se dirige a la topo I humana y los derivados como el topotecán y el irinotecán se utilizan ampliamente en la quimioterapia contra el cáncer. [33] La camptotecina y sus derivados actúan estabilizando el complejo de escisión de la topo I, evitando la religación de la mella mediada por proteínas en el ADN. Estos inhibidores interfaciales se estabilizan mediante interacciones de apilamiento con el ADN mellado y la unión de hidrógeno a la enzima. Aunque los derivados de CPT estabilizan un complejo de escisión de cadena sencilla, se cree que las colisiones posteriores con la maquinaria de replicación o transcripción generan roturas tóxicas del ADN bicatenario. Estos compuestos se utilizan como terapias de primera o segunda línea para tratar cánceres como el colorrectal, de ovario, de pulmón, de mama y de cuello uterino. Sin embargo, los derivados de CPT sufren limitaciones asociadas con la toxicidad y vidas medias terapéuticas limitadas debido a la inestabilidad química. Los nuevos inhibidores de la topo I, las indenoisoquinolinas y las fluoroindenoisoquinolinas, superan las limitaciones de los derivados de la CPT y actualmente se encuentran en ensayos clínicos. [49]
El etopósido (Fig. 7) y su pariente cercano, el tenipósido (VM-26), son derivados de la epipodofilotoxina obtenidos del rizoma de la mandrágora silvestre que actúan sobre la topo II estabilizando el complejo de escisión covalente y evitando la religación del ADN escindido. [46] Estos se utilizan típicamente junto con otros fármacos de quimioterapia para tratar cánceres, incluidos tumores testiculares, cáncer de pulmón de células pequeñas y leucemia. El tratamiento con etopósido puede provocar leucemias secundarias que surgen de translocaciones genómicas específicas, que involucran principalmente a la topo IIβ. [46]

La doxorrubicina (Fig. 7) y los derivados relacionados daunorrubicina, epirrubicina e idarrubicina son antraciclinas obtenidas de la bacteria Streptomyces [48] que se dirigen a la topo II humana, estabilizando el complejo de escisión de manera similar a otros venenos de la topoisomerasa. La mitoxantrona es una antracenodiona sintética que es química y funcionalmente similar a las antraciclinas. [47] Las antraciclinas fueron los primeros inhibidores de la topoisomerasa utilizados para tratar el cáncer y siguen estando entre los tratamientos más utilizados y efectivos para una amplia gama de cánceres, incluidos el cáncer de mama, el linfoma, las leucemias, los carcinomas, los sarcomas y otros tumores. [47] Estos compuestos son agentes intercalantes del ADN y, como tales, pueden afectar una amplia gama de procesos del ADN celular, además de envenenar específicamente la topo II. [33] La citotoxicidad adicional proviene de reacciones redox que involucran antraciclinas que generan especies reactivas de oxígeno. La generación de oxígeno reactivo, junto con el envenenamiento por topo IIβ, da como resultado la cardiotoxicidad limitante de la dosis de las antraciclinas. [33]
La merbarona es un derivado del ácido tiobarbitúrico y el dexrazoxano (ICRF-187), uno de los varios derivados relacionados de la bisdioxopiperazina (Fig. 7), son ejemplos de inhibidores catalíticos de la topo II, es decir, impiden la finalización del ciclo catalítico de la topo II pero no estabilizan el complejo de escisión del ADN. Si bien estos inhibidores catalíticos presentan citotoxicidad y se han probado en ensayos clínicos, actualmente no se utilizan clínicamente para la terapia del cáncer. [47] Sin embargo, el dexrazoxano, que bloquea la hidrólisis de ATP por la topo II, se utiliza para prevenir la cardiotoxicidad asociada con las antraciclinas. [50] [51]
Al menos una topoisomerasa, la ADN topoisomerasa II beta (topo IIβ), tiene un papel regulador en la transcripción génica. Las roturas de doble cadena de ADN dependientes de la topoisomerasa IIβ y los componentes de la maquinaria de reparación de daños en el ADN son importantes para la expresión rápida de genes tempranos inmediatos , así como para la regulación génica sensible a señales. [52] [53] [54] [55] La topoisomerasa IIβ, con otras enzimas asociadas, [54] parece ser importante para la liberación de la ARN polimerasa en pausa en genes largos o altamente transcritos. [56] [57] [58]
Las roturas de doble cadena de ADN inducidas por estímulos (DSB) que se limitan a un corto plazo (10 minutos a 2 horas) son inducidas por topo IIβ en las regiones promotoras de genes regulados por señales. Estas DSB permiten una rápida regulación positiva de la expresión de dichos genes sensibles a señales en varios sistemas (ver la Tabla a continuación). Estos genes regulados por señales incluyen genes activados en respuesta a la estimulación con estrógeno , suero , insulina , glucocorticoides (como dexametasona ) y activación de neuronas. Cuando la rotura de doble cadena de ADN inducida se ha reparado, entonces la transcripción del gen sensible a señales regresa a un nivel basal bajo. [52]

Se encontró que Topo IIβ y PARP-1 estaban constitutivamente presentes en un nivel moderado cerca del sitio de inicio de la transcripción de un promotor de un gen sensible a señales. Después de que se produjo la señal, topo IIβ causó una rotura de doble cadena y PARP-1 estuvo involucrado en reemplazar la histona H1 por HMGB1 / HMGA2 , que puede promover la transcripción. [55] Topo IIβ y PARP-1 aumentaron en el sitio de la rotura de doble cadena y los componentes de la vía de reparación del ADN de unión de extremos no homólogos , incluyendo DNA-PKcs, Ku70/Ku80 y ADN ligasa IV ensamblados con topo IIβ y PARP-1. Este ensamblaje estaba todo presente en el ADN de enlace adyacente a un solo nucleosoma en la región promotora de un gen (ver Figura). El nucleosoma estaba cerca del sitio de inicio de la transcripción del gen. [55] Los componentes de la vía de reparación del ADN mediante la unión de extremos no homólogos fueron esenciales para el cierre de la rotura de la doble cadena del ADN. [52]
La ARN polimerasa II frecuentemente tiene un sitio de pausa que se encuentra aproximadamente entre 30 y 60 nucleótidos aguas abajo del sitio de inicio de la transcripción de un gen. [64] [65] Se cree que la pausa de la ARN polimerasa II en estos sitios y la liberación controlada de la pausa tienen un papel regulador en la transcripción génica. Como señalaron Singh et al., [58] "alrededor del 80% de los genes altamente expresados en células HeLa están en pausa". Las roturas de doble cadena de ADN inducidas por topo IIβ a muy corto plazo, pero no reselladas inmediatamente, ocurren en los sitios de pausa de la ARN polimerasa II, y parecen ser necesarias para la liberación eficiente del estado de pausa y la progresión a la transcripción génica. [56] [57] [58] Para los genes en los que ocurre, se cree que la rotura de doble cadena de ADN inducida por TOP2B es parte del proceso de regulación de la expresión génica.