El aprendizaje automático en bioinformática es la aplicación de algoritmos de aprendizaje automático a la bioinformática , [1] incluyendo genómica , proteómica , microarrays , biología de sistemas , evolución y minería de texto . [2] [3]
Antes de la aparición del aprendizaje automático, los algoritmos bioinformáticos tenían que programarse a mano; para problemas como la predicción de la estructura de las proteínas , esto resultó difícil. [4] Las técnicas de aprendizaje automático, como el aprendizaje profundo , pueden aprender características de conjuntos de datos en lugar de requerir que el programador las defina individualmente. El algoritmo puede aprender además cómo combinar características de bajo nivel en características más abstractas, y así sucesivamente. Este enfoque de múltiples capas permite que estos sistemas realicen predicciones sofisticadas cuando se entrenan adecuadamente. Estos métodos contrastan con otros enfoques de biología computacional que, si bien explotan los conjuntos de datos existentes, no permiten que los datos se interpreten y analicen de formas imprevistas.
Los algoritmos de aprendizaje automático en bioinformática se pueden utilizar para la predicción, la clasificación y la selección de características. Los métodos para lograr esta tarea son variados y abarcan muchas disciplinas; los más conocidos son el aprendizaje automático y la estadística. Las tareas de clasificación y predicción tienen como objetivo construir modelos que describan y distingan clases o conceptos para futuras predicciones. Las diferencias entre ellos son las siguientes:
Debido al crecimiento exponencial de las tecnologías de la información y de los modelos aplicables, entre ellos la inteligencia artificial y la minería de datos, además del acceso a conjuntos de datos cada vez más completos, se han creado nuevas y mejores técnicas de análisis de la información, basadas en su capacidad de aprendizaje. Dichos modelos permiten llegar más allá de la descripción y proporcionar información en forma de modelos comprobables.
Las redes neuronales artificiales en bioinformática se han utilizado para: [5]
La forma en que se extraen las características, a menudo vectores en un espacio multidimensional, de los datos del dominio es un componente importante de los sistemas de aprendizaje. [6] En genómica, una representación típica de una secuencia es un vector de frecuencias de k-meros , que es un vector de dimensión cuyas entradas cuentan la aparición de cada subsecuencia de longitud en una secuencia dada. Dado que para un valor tan pequeño como la dimensionalidad de estos vectores es enorme (por ejemplo, en este caso la dimensión es ), se utilizan técnicas como el análisis de componentes principales para proyectar los datos a un espacio dimensional más bajo, seleccionando así un conjunto más pequeño de características de las secuencias. [6] [ cita(s) adicional(es) necesaria(s) ]
En este tipo de tarea de aprendizaje automático, el resultado es una variable discreta. Un ejemplo de este tipo de tarea en bioinformática es el etiquetado de nuevos datos genómicos (como genomas de bacterias no cultivables) basándose en un modelo de datos ya etiquetados. [6]
Los modelos ocultos de Markov (HMM) son una clase de modelos estadísticos para datos secuenciales (a menudo relacionados con sistemas que evolucionan con el tiempo). Un HMM se compone de dos objetos matemáticos: un proceso observado dependiente del estado y un proceso no observado (oculto) . En un HMM, el proceso de estado no se observa directamente (es una variable "oculta" o "latente"), sino que se realizan observaciones de un proceso dependiente del estado (o proceso de observación) que es impulsado por el proceso de estado subyacente (y que, por lo tanto, puede considerarse como una medición ruidosa de los estados del sistema de interés). [7] Los HMM se pueden formular en tiempo continuo. [8] [9]
Los HMM se pueden utilizar para perfilar y convertir una alineación de secuencias múltiples en un sistema de puntuación de posición específica adecuado para buscar secuencias homólogas en bases de datos de forma remota. [10] Además, los fenómenos ecológicos se pueden describir mediante HMM. [11]
Las redes neuronales convolucionales (CNN) son una clase de red neuronal profunda cuya arquitectura se basa en pesos compartidos de núcleos de convolución o filtros que se deslizan a lo largo de las características de entrada, proporcionando respuestas equivalentes a la traducción conocidas como mapas de características. [12] [13] Las CNN aprovechan el patrón jerárquico en los datos y ensamblan patrones de complejidad creciente utilizando patrones más pequeños y simples descubiertos a través de sus filtros. [14]
Las redes convolucionales se inspiraron en procesos biológicos [15] [16] [17] [18] en que el patrón de conectividad entre neuronas se asemeja a la organización de la corteza visual animal . Las neuronas corticales individuales responden a estímulos solo en una región restringida del campo visual conocida como campo receptivo . Los campos receptivos de diferentes neuronas se superponen parcialmente de tal manera que cubren todo el campo visual.
Las CNN utilizan relativamente poco preprocesamiento en comparación con otros algoritmos de clasificación de imágenes . Esto significa que la red aprende a optimizar los filtros (o núcleos) mediante aprendizaje automático, mientras que en los algoritmos tradicionales estos filtros se diseñan a mano . Esta menor dependencia del conocimiento previo del analista y de la intervención humana en la extracción manual de características hace que las CNN sean un modelo deseable. [14]
Una red neuronal convolucional filogenética (Ph-CNN) es una arquitectura de red neuronal convolucional propuesta por Fioranti et al. en 2018 para clasificar datos metagenómicos . [19] En este enfoque, los datos filogenéticos están dotados de distancia patrística (la suma de las longitudes de todas las ramas que conectan dos unidades taxonómicas operativas [OTU]) para seleccionar k vecindarios para cada OTU, y cada OTU y sus vecinos se procesan con filtros convolucionales.
A diferencia de los métodos supervisados, los métodos de aprendizaje autosupervisados aprenden representaciones sin depender de datos anotados. Esto es muy adecuado para la genómica, donde las técnicas de secuenciación de alto rendimiento pueden crear cantidades potencialmente grandes de datos no etiquetados. Algunos ejemplos de métodos de aprendizaje autosupervisados aplicados a la genómica incluyen DNABERT y Self-GenomeNet. [20] [21]

Los bosques aleatorios (RF) clasifican mediante la construcción de un conjunto de árboles de decisión y la generación de la predicción promedio de los árboles individuales. [22] Esta es una modificación de la agregación bootstrap (que agrega una gran colección de árboles de decisión) y se puede utilizar para la clasificación o la regresión . [23] [24]
Como los bosques aleatorios proporcionan una estimación interna del error de generalización, no es necesaria la validación cruzada. Además, producen proximidades que pueden utilizarse para imputar valores faltantes y que permiten nuevas visualizaciones de datos. [25]
Desde el punto de vista computacional, los bosques aleatorios son atractivos porque manejan de manera natural tanto la regresión como la clasificación (multiclase), son relativamente rápidos de entrenar y predecir, dependen solo de uno o dos parámetros de ajuste, tienen una estimación incorporada del error de generalización, se pueden usar directamente para problemas de alta dimensión y se pueden implementar fácilmente en paralelo. Desde el punto de vista estadístico, los bosques aleatorios son atractivos por sus características adicionales, como medidas de importancia variable, ponderación de clase diferencial, imputación de valores faltantes, visualización, detección de valores atípicos y aprendizaje no supervisado. [25]
La agrupación (la partición de un conjunto de datos en subconjuntos disjuntos, de modo que los datos de cada subconjunto estén lo más cerca posible entre sí y lo más lejos posible de los datos de cualquier otro subconjunto, de acuerdo con una función de distancia o similitud definida ) es una técnica común para el análisis estadístico de datos.
La agrupación es fundamental para gran parte de la investigación bioinformática basada en datos y sirve como un poderoso método computacional mediante el cual se ha estudiado y utilizado durante mucho tiempo en entornos de aprendizaje automático clásico mediante la clasificación jerárquica, basada en centroides, basada en distribución, basada en densidad y mapas autoorganizados. En particular, la agrupación ayuda a analizar datos no estructurados y de alta dimensión en forma de secuencias, expresiones, textos, imágenes, etc. La agrupación también se utiliza para obtener información sobre los procesos biológicos a nivel genómico , por ejemplo, funciones genéticas, procesos celulares, subtipos de células, regulación genética y procesos metabólicos. [26]
Los algoritmos de agrupamiento de datos pueden ser jerárquicos o particionales. Los algoritmos jerárquicos encuentran clústeres sucesivos utilizando clústeres previamente establecidos, mientras que los algoritmos particionales determinan todos los clústeres a la vez. Los algoritmos jerárquicos pueden ser aglomerativos (de abajo hacia arriba) o divisivos (de arriba hacia abajo).
Los algoritmos aglomerativos comienzan con cada elemento como un grupo separado y los fusionan en grupos sucesivamente más grandes. Los algoritmos divisivos comienzan con todo el conjunto y proceden a dividirlo en grupos sucesivamente más pequeños. El agrupamiento jerárquico se calcula utilizando métricas en espacios euclidianos , la más utilizada es la distancia euclidiana calculada al encontrar el cuadrado de la diferencia entre cada variable, sumando todos los cuadrados y encontrando la raíz cuadrada de dicha suma. Un ejemplo de un algoritmo de agrupamiento jerárquico es BIRCH , que es particularmente bueno en bioinformática por su complejidad temporal casi lineal dados conjuntos de datos generalmente grandes. [27] Los algoritmos de partición se basan en especificar un número inicial de grupos y reasignar iterativamente los objetos entre los grupos hasta la convergencia. Este algoritmo generalmente determina todos los grupos a la vez. La mayoría de las aplicaciones adoptan uno de los dos métodos heurísticos populares: algoritmo k-means o k-medoids . Otros algoritmos no requieren un número inicial de grupos, como la propagación de afinidad . En un entorno genómico, este algoritmo se ha utilizado tanto para agrupar grupos de genes biosintéticos en familias de grupos de genes (GCF) como para agrupar dichos GCF. [28]
Normalmente, un flujo de trabajo para aplicar el aprendizaje automático a los datos biológicos pasa por cuatro pasos: [2]
En general, un sistema de aprendizaje automático puede ser entrenado para reconocer elementos de una determinada clase dadas suficientes muestras. [30] Por ejemplo, los métodos de aprendizaje automático pueden ser entrenados para identificar características visuales específicas, como sitios de empalme. [31]
Las máquinas de vectores de soporte se han utilizado ampliamente en estudios genómicos del cáncer. [32] Además, el aprendizaje profundo se ha incorporado a algoritmos bioinformáticos. Las aplicaciones de aprendizaje profundo se han utilizado para la genómica regulatoria y la obtención de imágenes celulares. [33] Otras aplicaciones incluyen la clasificación de imágenes médicas, el análisis de secuencias genómicas, así como la clasificación y predicción de la estructura de las proteínas. [34] El aprendizaje profundo se ha aplicado a la genómica regulatoria, la identificación de variantes y las puntuaciones de patogenicidad. [35] El procesamiento del lenguaje natural y la minería de texto han ayudado a comprender fenómenos como la interacción proteína-proteína, la relación gen-enfermedad, así como a predecir las estructuras y funciones de las biomoléculas. [36]
Los algoritmos de procesamiento del lenguaje natural personalizan la medicina para pacientes que padecen enfermedades genéticas, combinando la extracción de información clínica y los datos genómicos disponibles de los pacientes. Institutos como Health-funded Pharmacogenomics Research Network se centran en encontrar tratamientos para el cáncer de mama. [37]
La medicina de precisión tiene en cuenta la variabilidad genómica individual, que se logra gracias a bases de datos biológicas a gran escala. El aprendizaje automático se puede aplicar para realizar la función de correspondencia entre (grupos de pacientes) y modalidades de tratamiento específicas. [38]
Las técnicas computacionales se utilizan para resolver otros problemas, como el diseño eficiente de cebadores para PCR , el análisis de imágenes biológicas y la retrotraducción de proteínas (que es, dada la degeneración del código genético, un problema combinatorio complejo). [2]
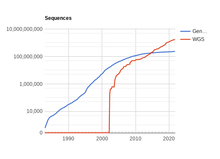
Si bien los datos de secuencias genómicas han sido históricamente escasos debido a la dificultad técnica de secuenciar un fragmento de ADN, la cantidad de secuencias disponibles está creciendo. En promedio, la cantidad de bases disponibles en el repositorio público GenBank se ha duplicado cada 18 meses desde 1982. [39] Sin embargo, si bien los datos sin procesar se estaban volviendo cada vez más disponibles y accesibles, a partir de 2002 [update], la interpretación biológica de estos datos se estaba produciendo a un ritmo mucho más lento. [40] Esto generó una creciente necesidad de desarrollar herramientas de genómica computacional , incluidos sistemas de aprendizaje automático, que puedan determinar automáticamente la ubicación de los genes que codifican proteínas dentro de una secuencia de ADN dada (es decir, predicción de genes ). [40]
La predicción de genes se realiza comúnmente a través de búsquedas extrínsecas e intrínsecas . [40] Para la búsqueda extrínseca, la secuencia de ADN de entrada se ejecuta a través de una gran base de datos de secuencias cuyos genes se han descubierto previamente y sus ubicaciones se han anotado e identificando los genes de la secuencia objetivo determinando qué cadenas de bases dentro de la secuencia son homólogas a secuencias de genes conocidas. Sin embargo, no todos los genes en una secuencia de entrada dada se pueden identificar solo a través de la homología, debido a los límites en el tamaño de la base de datos de secuencias de genes conocidas y anotadas. Por lo tanto, se necesita una búsqueda intrínseca donde un programa de predicción de genes intenta identificar los genes restantes solo a partir de la secuencia de ADN. [40]
El aprendizaje automático también se ha utilizado para el problema de la alineación de secuencias múltiples , que implica alinear muchas secuencias de ADN o aminoácidos para determinar regiones de similitud que podrían indicar una historia evolutiva compartida. [2] También se puede utilizar para detectar y visualizar reordenamientos del genoma. [41]

Las proteínas , cadenas de aminoácidos , obtienen gran parte de su función del plegamiento de proteínas , donde se adaptan a una estructura tridimensional, que incluye la estructura primaria , la estructura secundaria ( hélices alfa y láminas beta ), la estructura terciaria y la estructura cuaternaria .
La predicción de la estructura secundaria de las proteínas es un foco principal de este subcampo, ya que las estructuras terciarias y cuaternarias se determinan en función de la estructura secundaria. [4] Resolver la estructura verdadera de una proteína es costoso y requiere mucho tiempo, lo que aumenta la necesidad de sistemas que puedan predecir con precisión la estructura de una proteína analizando la secuencia de aminoácidos directamente. [4] [2] Antes del aprendizaje automático, los investigadores necesitaban realizar esta predicción manualmente. Esta tendencia comenzó en 1951 cuando Pauling y Corey publicaron su trabajo sobre la predicción de las configuraciones de enlaces de hidrógeno de una proteína a partir de una cadena polipeptídica. [42] El aprendizaje automático de características alcanza una precisión del 82-84%. [4] [43] El estado del arte actual en la predicción de la estructura secundaria utiliza un sistema llamado DeepCNF (campos neuronales convolucionales profundos) que se basa en el modelo de aprendizaje automático de redes neuronales artificiales para lograr una precisión de aproximadamente el 84% cuando se le asigna la tarea de clasificar los aminoácidos de una secuencia de proteína en una de tres clases estructurales (hélice, lámina o bobina). [43] [ Necesita actualización ] El límite teórico para la estructura secundaria de proteínas de tres estados es del 88 al 90 %. [4]
El aprendizaje automático también se ha aplicado a problemas de proteómica como la predicción de cadenas laterales de proteínas , el modelado de bucles de proteínas y la predicción de mapas de contacto de proteínas . [2]
La metagenómica es el estudio de las comunidades microbianas a partir de muestras de ADN ambiental. [44] Actualmente, las limitaciones y los desafíos predominan en la implementación de herramientas de aprendizaje automático debido a la cantidad de datos en las muestras ambientales. [45] Las supercomputadoras y los servidores web han facilitado el acceso a estas herramientas. [46] La alta dimensionalidad de los conjuntos de datos del microbioma es un desafío importante en el estudio del microbioma; esto limita significativamente el poder de los enfoques actuales para identificar diferencias verdaderas y aumenta la posibilidad de descubrimientos falsos. [47] [ se necesita una mejor fuente ]
A pesar de su importancia, las herramientas de aprendizaje automático relacionadas con la metagenómica se han centrado en el estudio de la microbiota intestinal y la relación con enfermedades digestivas, como la enfermedad inflamatoria intestinal (EII), la infección por Clostridioides difficile (CDI), el cáncer colorrectal y la diabetes , buscando un mejor diagnóstico y tratamientos. [46] Se desarrollaron muchos algoritmos para clasificar las comunidades microbianas de acuerdo con el estado de salud del huésped, independientemente del tipo de datos de secuencia, por ejemplo, ARNr 16S o secuenciación del genoma completo (WGS), utilizando métodos como el clasificador de operador de selección y contracción absoluta mínima, bosque aleatorio , modelo de clasificación supervisada y modelo de árbol potenciado por gradiente. Se han agregado redes neuronales , como redes neuronales recurrentes (RNN), redes neuronales convolucionales (CNN) y redes neuronales de Hopfield . [46] Por ejemplo, en 2018, Fioravanti et al. desarrolló un algoritmo llamado Ph-CNN para clasificar muestras de datos de pacientes sanos y pacientes con síntomas de EII (para distinguir pacientes sanos y enfermos) mediante el uso de árboles filogenéticos y redes neuronales convolucionales. [48]
Además, los métodos de bosque aleatorio (RF) y las medidas de importancia implementadas ayudan en la identificación de especies de microbioma que pueden usarse para distinguir muestras enfermas y no enfermas. Sin embargo, el rendimiento de un árbol de decisión y la diversidad de árboles de decisión en el conjunto influyen significativamente en el rendimiento de los algoritmos de RF. El error de generalización para RF mide qué tan precisos son los clasificadores individuales y su interdependencia. Por lo tanto, los problemas de alta dimensionalidad de los conjuntos de datos de microbioma plantean desafíos. Los enfoques efectivos requieren muchas combinaciones posibles de variables, lo que aumenta exponencialmente la carga computacional a medida que aumenta el número de características. [47]
Para el análisis del microbioma en 2020, Dang y Kishino [47] desarrollaron un nuevo proceso de análisis. El núcleo del proceso es un clasificador RF acoplado con una selección de variables de reenvío (RF-FVS), que selecciona un conjunto básico de especies microbianas o firmas funcionales de tamaño mínimo que maximizan el rendimiento del clasificador predictivo. El marco combina:
Demostraron su rendimiento analizando dos conjuntos de datos publicados de estudios de casos y controles a gran escala:
El enfoque propuesto mejoró la precisión del 81% al 99,01% para CDI y del 75,14% al 90,17% para CRC.
El uso del aprendizaje automático en muestras ambientales ha sido menos explorado, tal vez debido a la complejidad de los datos, especialmente de WGS. Algunos trabajos muestran que es posible aplicar estas herramientas en muestras ambientales. En 2021, Dhungel et al., [49] diseñaron un paquete R llamado MegaR. Este paquete permite trabajar con ARNr 16S y secuencias metagenómicas completas para realizar perfiles taxonómicos y modelos de clasificación mediante modelos de aprendizaje automático. MegaR incluye un entorno de visualización cómodo para mejorar la experiencia del usuario. El aprendizaje automático en metagenómica ambiental puede ayudar a responder preguntas relacionadas con las interacciones entre las comunidades microbianas y los ecosistemas, por ejemplo, el trabajo de Xun et al., en 2021 [50] donde el uso de diferentes métodos de aprendizaje automático ofreció información sobre la relación entre el suelo, la biodiversidad del microbioma y la estabilidad del ecosistema.
Los microarrays , un tipo de laboratorio en un chip , se utilizan para recopilar automáticamente datos sobre grandes cantidades de material biológico. El aprendizaje automático puede ayudar en el análisis y se ha aplicado a la identificación de patrones de expresión, la clasificación y la inducción de redes genéticas. [2]

Esta tecnología es especialmente útil para monitorear la expresión genética, ayudando a diagnosticar el cáncer al examinar qué genes se expresan. [51] Una de las tareas principales es identificar qué genes se expresan en función de los datos recopilados. [2] Además, debido a la gran cantidad de genes sobre los que se recopilan datos mediante el microarray, es un desafío seleccionar la gran cantidad de datos irrelevantes para la tarea de identificación de genes expresados. El aprendizaje automático presenta una solución potencial, ya que se pueden utilizar varios métodos de clasificación para realizar esta identificación. Los métodos más utilizados son las redes de funciones de base radial , el aprendizaje profundo , la clasificación bayesiana , los árboles de decisión y el bosque aleatorio . [51]
La biología de sistemas se centra en el estudio de los comportamientos emergentes a partir de interacciones complejas de componentes biológicos simples en un sistema. Dichos componentes pueden incluir ADN, ARN, proteínas y metabolitos. [52]
El aprendizaje automático se ha utilizado para ayudar a modelar estas interacciones en dominios como redes genéticas, redes de transducción de señales y vías metabólicas. [2] Los modelos gráficos probabilísticos , una técnica de aprendizaje automático para determinar la relación entre diferentes variables, son uno de los métodos más utilizados para modelar redes genéticas. [2] Además, el aprendizaje automático se ha aplicado a problemas de biología de sistemas como la identificación de sitios de unión de factores de transcripción mediante la optimización de la cadena de Markov . [2] Los algoritmos genéticos , técnicas de aprendizaje automático que se basan en el proceso natural de la evolución, se han utilizado para modelar redes genéticas y estructuras reguladoras. [2]
Otras aplicaciones de aprendizaje automático en biología de sistemas incluyen la tarea de predicción de funciones enzimáticas, análisis de datos de microarrays de alto rendimiento, análisis de estudios de asociación de todo el genoma para comprender mejor los marcadores de enfermedades y predicción de funciones proteicas. [53]
Este dominio, en particular la reconstrucción de árboles filogenéticos , utiliza las características de las técnicas de aprendizaje automático. Los árboles filogenéticos son representaciones esquemáticas de la evolución de los organismos. Inicialmente, se construyeron utilizando características como las características morfológicas y metabólicas. Más tarde, debido a la disponibilidad de secuencias genómicas, la construcción del algoritmo del árbol filogenético utilizó el concepto basado en la comparación de genomas. Con la ayuda de técnicas de optimización, se realizó una comparación mediante el alineamiento de secuencias múltiples. [54]
Los métodos de aprendizaje automático para el análisis de datos de neuroimagen se utilizan para ayudar a diagnosticar accidentes cerebrovasculares . Históricamente, múltiples enfoques para este problema involucraban redes neuronales. [55] [56]
Existen múltiples enfoques para detectar accidentes cerebrovasculares que utilizan el aprendizaje automático. Como propuso Mirtskhulava, [57] se probaron redes de propagación hacia adelante para detectar accidentes cerebrovasculares utilizando imágenes neuronales. Como propuso Titano [58] se probaron técnicas de 3D-CNN en la clasificación supervisada para examinar imágenes de TC de cabeza en busca de eventos neurológicos agudos. A menudo se utilizan métodos tridimensionales de CNN y SVM . [56]
El aumento de las publicaciones biológicas aumentó la dificultad de buscar y recopilar información relevante disponible sobre un tema determinado. Esta tarea se conoce como extracción de conocimiento . Es necesaria para la recopilación de datos biológicos que luego pueden, a su vez, introducirse en algoritmos de aprendizaje automático para generar nuevos conocimientos biológicos. [2] [59] El aprendizaje automático se puede utilizar para esta tarea de extracción de conocimiento utilizando técnicas como el procesamiento del lenguaje natural para extraer la información útil de los informes generados por humanos en una base de datos. Text Nailing , un enfoque alternativo al aprendizaje automático, capaz de extraer características de las notas narrativas clínicas, se introdujo en 2017.
Esta técnica se ha aplicado a la búsqueda de nuevos objetivos farmacológicos, ya que esta tarea requiere el examen de la información almacenada en bases de datos y revistas biológicas. [59] Las anotaciones de proteínas en bases de datos de proteínas a menudo no reflejan el conjunto completo de conocimientos conocidos de cada proteína, por lo que se debe extraer información adicional de la literatura biomédica. El aprendizaje automático se ha aplicado a la anotación automática de la función de genes y proteínas, la determinación de la localización subcelular de proteínas , el análisis de matrices de expresión de ADN , el análisis de interacciones de proteínas a gran escala y el análisis de interacciones de moléculas. [59]
Otra aplicación de la minería de texto es la detección y visualización de distintas regiones de ADN dados suficientes datos de referencia. [60]
Las comunidades microbianas son conjuntos complejos de diversos microorganismos, [61] donde los socios simbiontes producen constantemente diversos metabolitos derivados del metabolismo primario y secundario (especializado), del cual el metabolismo juega un papel importante en la interacción microbiana. [62] Los datos metagenómicos y metatranscriptómicos son una fuente importante para descifrar señales de comunicación.
Los mecanismos moleculares producen metabolitos especializados de diversas maneras. Los grupos de genes biosintéticos (BGC) atraen la atención, ya que varios metabolitos son agentes clínicamente valiosos, antimicrobianos, antifúngicos, antiparasitarios, antitumorales e inmunosupresores producidos por la acción modular de grupos de genes multienzimáticos y multidominios, como las sintetasas de péptidos no ribosomales (NRPS) y las sintetasas de policétidos (PKS). [63] Diversos estudios [64] [65] [66] [67] [68] [69] [ 70] [71] muestran que agrupar los BGC que comparten genes centrales homólogos en familias de grupos de genes (GCF) puede brindar información útil sobre la diversidad química de las cepas analizadas y puede respaldar la vinculación de los BGC con sus metabolitos secundarios. [65] [67] Los GCF se han utilizado como marcadores funcionales en estudios de salud humana [72] [73] y para estudiar la capacidad del suelo para suprimir patógenos fúngicos. [74] Dada su relación directa con las enzimas catalíticas y los compuestos producidos a partir de sus vías codificadas, los BGC/GCF pueden servir como un proxy para explorar el espacio químico del metabolismo secundario microbiano. La catalogación de los GCF en genomas microbianos secuenciados proporciona una visión general de la diversidad química existente y ofrece información sobre las prioridades futuras. [64] [66] Herramientas como BiG-SLiCE y BIG-MAP [75] han surgido con el único propósito de revelar la importancia de los BGC en entornos naturales.
El aumento de péptidos sintetizados ribosómicamente y modificados postraduccionalmente (RiPPs) caracterizados experimentalmente , junto con la disponibilidad de información sobre su secuencia y estructura química, seleccionada de bases de datos como BAGEL, BACTIBASE, MIBIG y THIOBASE, brindan la oportunidad de desarrollar herramientas de aprendizaje automático para decodificar la estructura química y clasificarlos.
En 2017, investigadores del Instituto Nacional de Inmunología de Nueva Delhi, India, desarrollaron el software RiPPMiner [76] , un recurso bioinformático para decodificar las estructuras químicas de RiPP mediante la minería del genoma. El servidor web RiPPMiner consta de una interfaz de consulta y la base de datos RiPPDB. RiPPMiner define 12 subclases de RiPP, prediciendo el sitio de escisión del péptido líder y el enlace cruzado final de la estructura química de RiPP.
Muchos estudios metabolómicos basados en espectrometría de masas en tándem ( MS/MS ) , como la comparación de bibliotecas y las redes moleculares, utilizan la similitud espectral como un indicador de la similitud estructural. El algoritmo Spec2vec [77] proporciona una nueva forma de puntuación de similitud espectral, basada en Word2Vec . Spec2Vec aprende relaciones fragmentarias dentro de un gran conjunto de datos espectrales, con el fin de evaluar similitudes espectrales entre moléculas y clasificar moléculas desconocidas a través de estas comparaciones.
Para la anotación sistémica, algunos estudios de metabolómica se basan en ajustar los espectros de masas de fragmentación medidos a los espectros de la biblioteca o contrastarlos mediante análisis de redes. Las funciones de puntuación se utilizan para determinar la similitud entre pares de espectros de fragmentos como parte de estos procesos. Hasta ahora, ninguna investigación ha sugerido puntuaciones que sean significativamente diferentes de la similitud basada en coseno que se utiliza comúnmente . [78]
Una parte importante de la bioinformática es la gestión de grandes conjuntos de datos, conocidos como bases de datos de referencia. Existen bases de datos para cada tipo de datos biológicos, por ejemplo, para grupos de genes biosintéticos y metagenomas.
El Centro Nacional de Información Biotecnológica (NCBI) [79] ofrece una amplia gama de recursos en línea para obtener información y datos biológicos, incluida la base de datos de secuencias de ácidos nucleicos GenBank y la base de datos PubMed de citas y resúmenes de revistas científicas publicadas. Muchas de las aplicaciones web se complementan con implementaciones personalizadas del programa BLAST optimizado para buscar conjuntos de datos especializados. Los recursos incluyen PubMed Data Management, RefSeq Functional Elements, descarga de datos genómicos, API de servicios de variación, Magic-BLAST, QuickBLASTp y Identical Protein Groups. Se puede acceder a todos estos recursos a través del NCBI. [80]
antiSMASH permite la rápida identificación, anotación y análisis de grupos de genes de biosíntesis de metabolitos secundarios en genomas bacterianos y fúngicos. Se integra y se enlaza con una gran cantidad de herramientas de análisis de metabolitos secundarios in silico. [81]
gutSMASH es una herramienta que evalúa sistemáticamente el potencial metabólico bacteriano al predecir grupos de genes metabólicos anaeróbicos (MGC) conocidos y nuevos del microbioma intestinal .
MIBiG, [82] la especificación de información mínima sobre un grupo de genes biosintéticos, proporciona un estándar para anotaciones y metadatos sobre grupos de genes biosintéticos y sus productos moleculares. MIBiG es un proyecto del Consorcio de Estándares Genómicos que se basa en el marco de información mínima sobre cualquier secuencia (MIxS). [83]
MIBiG facilita la deposición y recuperación estandarizada de datos de grupos de genes biosintéticos, así como el desarrollo de herramientas integrales de análisis comparativo. Potencia la investigación de próxima generación sobre la biosíntesis, la química y la ecología de amplias clases de metabolitos secundarios bioactivos socialmente relevantes , guiada por evidencia experimental sólida y ricos componentes de metadatos. [84]
SILVA [85] es un proyecto interdisciplinario entre biólogos y científicos informáticos que reúne una base de datos completa de secuencias de genes de ARN ribosómico (ARNr), tanto de subunidades pequeñas ( 16S , 18S , SSU) como grandes ( 23S , 28S , LSU), que pertenecen a los dominios de bacterias, arqueas y eucariotas. Estos datos están disponibles de forma gratuita para uso académico y comercial. [86]
Greengenes [87] es una base de datos completa de genes ARNr 16S que proporciona detección de quimeras, alineación estándar y una taxonomía curada basada en la inferencia de árboles de novo. [88] [89] Descripción general:
Open Tree of Life Taxonomy (OTT) [90] tiene como objetivo construir un árbol de la vida completo, dinámico y disponible digitalmente mediante la síntesis de árboles filogenéticos publicados junto con datos taxonómicos. Los árboles filogenéticos se han clasificado, alineado y fusionado. Las taxonomías se han utilizado para rellenar regiones dispersas y huecos dejados por las filogenias. OTT es una base que se ha utilizado poco para análisis de secuenciación de la región 16S, sin embargo, tiene un mayor número de secuencias clasificadas taxonómicamente hasta el nivel de género en comparación con SILVA y Greengenes. Sin embargo, en términos de clasificación a nivel de borde, contiene una menor cantidad de información [91].
Ribosomal Database Project (RDP) [92] es una base de datos que proporciona secuencias de ARN ribosómico (ARNr) de subunidades pequeñas de dominio bacteriano y arqueológico ( 16S ); y secuencias de ARNr fúngico de subunidades grandes ( 28S ). [93]