El Proyecto 1000 Genomas ( 1KGP ), que se llevó a cabo entre enero de 2008 y 2015, fue un esfuerzo de investigación internacional para establecer el catálogo más detallado de la variación genética humana en ese momento. Los científicos planearon secuenciar los genomas de al menos mil participantes sanos anónimos de varios grupos étnicos diferentes en los siguientes tres años, utilizando avances en tecnologías recientemente desarrolladas . En 2010, el proyecto terminó su fase piloto, que se describió en detalle en una publicación en la revista Nature . [1] En 2012, la secuenciación de 1092 genomas se anunció en una publicación de Nature . [2] En 2015, dos artículos en Nature informaron los resultados y la finalización del proyecto y las oportunidades para futuras investigaciones. [3] [4]
Se identificaron muchas variaciones raras, restringidas a grupos estrechamente relacionados, y se analizaron ocho clases de variación estructural. [5]
El proyecto unió a equipos de investigación multidisciplinarios de institutos de todo el mundo, incluidos China , Italia , Japón , Kenia , Nigeria , Perú , el Reino Unido y los Estados Unidos , que contribuyeron al conjunto de datos de secuencias y a un mapa refinado del genoma humano de libre acceso a través de bases de datos públicas para la comunidad científica y el público en general por igual. [2]
El Recurso Internacional de Muestras del Genoma se creó para albergar y ampliar el conjunto de datos una vez finalizado el proyecto. [6]

Desde la finalización del Proyecto Genoma Humano, los avances en la genética de poblaciones humanas y la genómica comparativa permitieron un mayor conocimiento de la diversidad genética. [7] La comprensión de las variaciones estructurales (inserciones/deleciones ( indels ), variaciones del número de copias (CNV), retroelementos ), polimorfismos de un solo nucleótido (SNP) y selección natural se estaba consolidando. [8] [9] [10] [11]
La diversidad de la variación genética humana, como los indeles, se estaban descubriendo y se estaban investigando las variaciones genómicas humanas [ cita requerida ]
También se propuso proporcionar evidencia que pueda utilizarse para explorar el impacto de la selección natural en las diferencias poblacionales. Los patrones de polimorfismos de ADN pueden utilizarse para detectar de manera confiable las firmas de selección y pueden ayudar a identificar genes que podrían ser la base de la variación en la resistencia a las enfermedades o el metabolismo de los fármacos. [12] [13] Estos conocimientos podrían mejorar la comprensión de las variaciones fenotípicas , los trastornos genéticos y la herencia mendeliana y sus efectos en la supervivencia y/o reproducción de diferentes poblaciones humanas.
El Proyecto 1000 Genomas fue diseñado para cerrar la brecha de conocimiento entre las variantes genéticas raras que tienen un efecto severo predominantemente en rasgos simples (por ejemplo, fibrosis quística , enfermedad de Huntington ) y las variantes genéticas comunes que tienen un efecto leve y están implicadas en rasgos complejos (por ejemplo, cognición , diabetes , enfermedad cardíaca ). [14]
El objetivo principal de este proyecto era crear un catálogo completo y detallado de las variaciones genéticas humanas , que se pueda utilizar para estudios de asociación que relacionen la variación genética con las enfermedades. El consorcio tenía como objetivo descubrir >95 % de las variantes (por ejemplo, SNP, CNV, indels) con frecuencias de alelos menores tan bajas como el 1 % en todo el genoma y el 0,1-0,5 % en las regiones genéticas, así como estimar las frecuencias poblacionales, los antecedentes haplotípicos y los patrones de desequilibrio de ligamiento de los alelos variantes. [15]
Los objetivos secundarios incluyeron el apoyo a una mejor selección de SNP y sondas para plataformas de genotipado en estudios futuros y la mejora de la secuencia de referencia humana . Se esperaba que la base de datos completa fuera una herramienta útil para estudiar las regiones bajo selección, la variación en múltiples poblaciones y la comprensión de los procesos subyacentes de mutación y recombinación . [15]
El genoma humano consta de aproximadamente 3 mil millones de pares de bases de ADN y se estima que contiene alrededor de 20.000 genes codificadores de proteínas . Al diseñar el estudio, el consorcio tuvo que abordar varias cuestiones críticas relacionadas con las métricas del proyecto, como los desafíos tecnológicos, los estándares de calidad de los datos y la cobertura de secuencias. [15]
En el transcurso de los próximos tres años, los científicos del Instituto Sanger , BGI Shenzhen y la Red de Secuenciación a Gran Escala del Instituto Nacional de Investigación del Genoma Humano planearon secuenciar un mínimo de 1.000 genomas humanos. Debido a la gran cantidad de datos de secuenciación que se requerían, se mantuvo el reclutamiento de participantes adicionales. [14]
Se debían secuenciar casi 10 mil millones de bases por día durante un período de dos años de la fase de producción, lo que equivale a más de dos genomas humanos cada 24 horas. El conjunto de datos de secuencias previsto debía comprender 6 billones de bases de ADN, 60 veces más datos de secuencias que lo que se había publicado en las bases de datos de ADN hasta el momento. [14]
Para determinar el diseño final del proyecto completo, se llevaron a cabo tres estudios piloto durante el primer año del proyecto. El primer piloto pretende genotipar a 180 personas de 3 grupos geográficos principales con una cobertura baja (2×). Para el segundo estudio piloto, se secuenciarán los genomas de dos familias nucleares (ambos padres y un hijo adulto) con una cobertura profunda (20× por genoma). El tercer estudio piloto implica la secuenciación de las regiones codificantes ( exones ) de 1000 genes en 1000 personas con una cobertura profunda (20×). [14] [15]
Se estimó que el proyecto probablemente costaría más de 500 millones de dólares si se utilizaran tecnologías de secuenciación de ADN estándar. Se aplicarían varias tecnologías más nuevas (por ejemplo, Solexa , 454 , SOLiD ), lo que reduciría los costos esperados a entre 30 y 50 millones de dólares. El principal apoyo lo brindarán el Wellcome Trust Sanger Institute en Hinxton, Inglaterra; el Beijing Genomics Institute , Shenzhen (BGI Shenzhen), China; y el NHGRI , parte de los Institutos Nacionales de Salud (NIH). [14]
De acuerdo con los principios de Fort Lauderdale Archivado el 28 de diciembre de 2013 en Wayback Machine , todos los datos de la secuencia del genoma (incluidas las llamadas variantes) están disponibles gratuitamente a medida que avanza el proyecto y se pueden descargar a través de FTP desde la página web del proyecto 1000 genomas.
En función de los objetivos generales del proyecto, las muestras se seleccionarán para que aporten potencia en poblaciones en las que se están realizando estudios de asociación de enfermedades comunes. Además, las muestras no necesitan tener información médica o fenotípica, ya que el catálogo propuesto será un recurso básico sobre la variación humana. [15]
Para los estudios piloto se secuenciarán muestras del genoma humano de la colección HapMap . Será útil centrarse en muestras que tengan datos adicionales disponibles (como la secuencia ENCODE , los genotipos de todo el genoma, la secuencia del extremo de los fósmidos , los ensayos de variación estructural y la expresión génica ) para poder comparar los resultados con los de otros proyectos. [15]
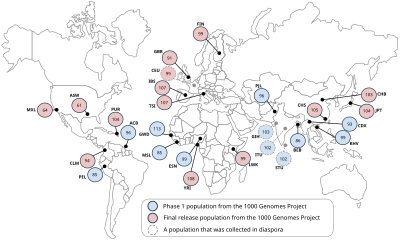
Cumpliendo con extensos procedimientos éticos, el Proyecto 1000 Genomas utilizará muestras de donantes voluntarios. Las siguientes poblaciones se incluirán en el estudio: Yoruba en Ibadan (YRI), Nigeria ; Japoneses en Tokio (JPT); Chinos en Beijing (CHB); Residentes de Utah con ascendencia del norte y oeste de Europa (CEU); Luhya en Webuye , Kenia (LWK); Maasai en Kinyawa, Kenia (MKK); Toscani en Italia (TSI); Peruanos en Lima , Perú (PEL); Indios Gujarati en Houston (GIH); Chinos en el área metropolitana de Denver (CHD); Personas de ascendencia mexicana en Los Ángeles (MXL); y Personas de ascendencia africana en el suroeste de los Estados Unidos (ASW). [14]
* Población que fue recolectada en diáspora
Los datos generados por el Proyecto 1000 Genomas son ampliamente utilizados por la comunidad genética, lo que convierte al primer Proyecto 1000 Genomas en uno de los artículos más citados en biología. [17] Para apoyar a esta comunidad de usuarios, el proyecto celebró una reunión de análisis de la comunidad en julio de 2012 que incluyó charlas que destacaron los descubrimientos clave del proyecto, su impacto en la genética de poblaciones y los estudios de enfermedades humanas, y resúmenes de otros estudios de secuenciación a gran escala. [18]
La fase piloto consistió en tres proyectos:
Se descubrió que, en promedio, cada persona porta alrededor de 250 a 300 variantes con pérdida de función en genes anotados y entre 50 y 100 variantes previamente implicadas en trastornos hereditarios. Con base en los dos tríos, se estima que la tasa de mutación de novo en la línea germinal es de aproximadamente 10 −8 por base por generación. [1]