El ligamiento genético es la tendencia de las secuencias de ADN que están muy juntas en un cromosoma a heredarse juntas durante la fase de meiosis de la reproducción sexual . Es poco probable que dos marcadores genéticos que están físicamente cerca uno del otro se separen en cromátidas diferentes durante el cruce cromosómico y, por lo tanto, se dice que están más vinculados que los marcadores que están muy separados. En otras palabras, cuanto más cerca estén dos genes en un cromosoma, menor será la posibilidad de recombinación entre ellos y mayor será la probabilidad de que se hereden juntos. Los marcadores en diferentes cromosomas están perfectamente desvinculados , aunque la penetrancia de alelos potencialmente nocivos puede verse influenciada por la presencia de otros alelos, y estos otros alelos pueden estar ubicados en otros cromosomas distintos de aquel en el que se encuentra un alelo potencialmente nocivo en particular. [1]
El ligamiento genético es la excepción más destacada a la Ley de Surtido Independiente de Gregor Mendel . El primer experimento para demostrar el vínculo se llevó a cabo en 1905. En ese momento, se desconocía la razón por la cual ciertos rasgos tienden a heredarse juntos. Trabajos posteriores revelaron que los genes son estructuras físicas relacionadas por la distancia física.
La unidad típica de ligamiento genético es el centimorgan (cM). Una distancia de 1 cM entre dos marcadores significa que los marcadores se separan en diferentes cromosomas en promedio una vez por cada 100 productos meióticos, es decir, una vez por cada 50 meiosis.
La Ley de Surtido Independiente de Gregor Mendel establece que cada rasgo se hereda independientemente de cualquier otro rasgo. Pero poco después de que se redescubriera la obra de Mendel , se encontraron excepciones a esta regla. En 1905, los genetistas británicos William Bateson , Edith Rebecca Saunders y Reginald Punnett cruzaron plantas de guisantes en experimentos similares a los de Mendel. [2] [3] Estaban interesados en la herencia de rasgos en el guisante de olor y estaban estudiando dos genes: el gen del color de la flor ( P , púrpura y p , rojo) y el gen que afecta la forma de los granos de polen ( L , largo , y l , redondo). Cruzaron las líneas puras PPLL y ppll y luego autocruzaron las líneas PpLl resultantes.
Según la genética mendeliana , los fenotipos esperados se producirían en una proporción de 9:3:3:1 de PL:Pl:pL:pl. Para su sorpresa, observaron una mayor frecuencia de PL y pl y una disminución de la frecuencia de Pl y pL:
Su experimento reveló un vínculo entre los alelos P y L y los alelos p y l . La frecuencia de que P aparezca junto con L y de que p aparezca junto con l es mayor que la de Pl y pL recombinantes . La frecuencia de recombinación es más difícil de calcular en un cruce F2 que en un retrocruzamiento, [4] pero la falta de ajuste entre los números de progenie observados y esperados en la tabla anterior indica que es inferior al 50%. Esto indicó que dos factores interactuaron de alguna manera para crear esta diferencia al enmascarar la apariencia de los otros dos fenotipos. Esto llevó a la conclusión de que algunos rasgos están relacionados entre sí debido a su proximidad entre sí en un cromosoma.
La comprensión del vínculo se amplió con el trabajo de Thomas Hunt Morgan . La observación de Morgan de que la cantidad de cruce entre genes vinculados difiere llevó a la idea de que la frecuencia de cruce podría indicar la distancia que separa los genes en el cromosoma . El centimorgan , que expresa la frecuencia de cruce, lleva su nombre en su honor.
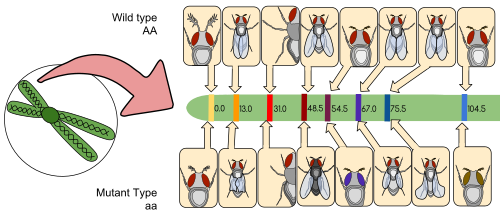
Un mapa de ligamiento (también conocido como mapa genético ) es una tabla para una especie o población experimental que muestra la posición de sus genes conocidos o marcadores genéticos entre sí en términos de frecuencia de recombinación, en lugar de una distancia física específica a lo largo de cada cromosoma. . Los mapas de vinculación fueron desarrollados por primera vez por Alfred Sturtevant , un estudiante de Thomas Hunt Morgan .
Un mapa de ligamiento es un mapa basado en las frecuencias de recombinación entre marcadores durante el cruce de cromosomas homólogos . Cuanto mayor es la frecuencia de recombinación (segregación) entre dos marcadores genéticos, más separados se supone que están. Por el contrario, cuanto menor es la frecuencia de recombinación entre los marcadores, menor es la distancia física entre ellos. Históricamente, los marcadores utilizados originalmente eran fenotipos detectables (producción de enzimas, color de ojos) derivados de secuencias codificantes de ADN ; eventualmente, se han utilizado secuencias de ADN no codificantes confirmadas o supuestas , como microsatélites o aquellas que generan polimorfismos de longitud de fragmentos de restricción ( RFLP ).
Los mapas de vinculación ayudan a los investigadores a localizar otros marcadores, como otros genes, mediante pruebas de vinculación genética de los marcadores ya conocidos. En las primeras etapas del desarrollo de un mapa de vinculación, los datos se utilizan para ensamblar grupos de vinculación , un conjunto de genes que se sabe que están vinculados. A medida que avanza el conocimiento, se pueden agregar más marcadores a un grupo, hasta que el grupo cubra un cromosoma completo. [6] Para organismos bien estudiados, los grupos de enlace se corresponden uno a uno con los cromosomas.
Un mapa de vinculación no es un mapa físico (como un mapa híbrido de radiación reducida ) ni un mapa genético .
El análisis de ligamiento es un método genético que busca segmentos cromosómicos que se cosegregan con el fenotipo de la enfermedad a través de familias. [7] Puede utilizarse para mapear genes de rasgos tanto binarios como cuantitativos. [7] El análisis de ligamiento puede ser paramétrico (si conocemos la relación entre la similitud fenotípica y genética) o no paramétrico. El análisis de ligamiento paramétrico es el enfoque tradicional, mediante el cual la probabilidad de que un gen importante para una enfermedad esté vinculado a un marcador genético se estudia a través de la puntuación LOD, que evalúa la probabilidad de que un determinado pedigrí, donde la enfermedad y el marcador están cosegregados, sea debido a la existencia de vínculo (con un valor de vínculo determinado) o al azar. El análisis de ligamiento no paramétrico, a su vez, estudia la probabilidad de que un alelo sea idéntico por descendencia a sí mismo.

La puntuación LOD (logaritmo (base 10) de probabilidades), desarrollada por Newton Morton , [8] es una prueba estadística que se utiliza a menudo para el análisis de ligamiento en poblaciones humanas, animales y vegetales. La puntuación LOD compara la probabilidad de obtener los datos de la prueba si los dos loci están realmente vinculados, con la probabilidad de observar los mismos datos simplemente por casualidad. Las puntuaciones LOD positivas favorecen la presencia de vinculación, mientras que las puntuaciones LOD negativas indican que la vinculación es menos probable. El análisis computarizado de la puntuación LOD es una forma sencilla de analizar genealogías familiares complejas para determinar el vínculo entre los rasgos mendelianos (o entre un rasgo y un marcador, o dos marcadores).
Strachan y Read describen el método con mayor detalle.[1] Brevemente, funciona de la siguiente manera:
La puntuación LOD se calcula de la siguiente manera:
NR indica el número de descendencia no recombinante y R indica el número de descendencia recombinante. La razón por la que se utiliza 0,5 en el denominador es que cualquier alelo que esté completamente desvinculado (por ejemplo, alelos en cromosomas separados) tiene un 50% de posibilidades de recombinación, debido a la distribución independiente. θ es la fracción recombinante, es decir, la fracción de nacimientos en los que se ha producido una recombinación entre el marcador genético estudiado y el gen putativo asociado con la enfermedad. Por tanto, es igual a R /( NR + R ) .
Por convención, una puntuación LOD superior a 3,0 se considera evidencia de vinculación, ya que indica probabilidades de 1000 a 1 de que la vinculación observada no ocurrió por casualidad. Por otro lado, una puntuación LOD inferior a -2,0 se considera evidencia para excluir el vínculo. Aunque es muy poco probable que se obtenga una puntuación LOD de 3 a partir de un único pedigrí, las propiedades matemáticas de la prueba permiten combinar datos de varios pedigríes sumando sus puntuaciones LOD. Una puntuación LOD de 3 se traduce en un valor p de aproximadamente 0,05, [9] y no se requiere corrección de pruebas múltiples (por ejemplo, corrección de Bonferroni ). [10]
El análisis de vinculación tiene una serie de limitaciones metodológicas y teóricas que pueden aumentar significativamente la tasa de error tipo 1 y reducir el poder para mapear loci de rasgos cuantitativos (QTL) humanos. [11] Si bien el análisis de vinculación se utilizó con éxito para identificar variantes genéticas que contribuyen a trastornos raros como la enfermedad de Huntington , no funcionó tan bien cuando se aplicó a trastornos más comunes como enfermedades cardíacas o diferentes formas de cáncer . [12] Una explicación para esto es que los mecanismos genéticos que afectan los trastornos comunes son diferentes de los que causan algunos trastornos raros. [13]
La frecuencia de recombinación es una medida del vínculo genético y se utiliza en la creación de un mapa de vínculo genético. La frecuencia de recombinación ( θ ) es la frecuencia con la que se producirá un único cruce cromosómico entre dos genes durante la meiosis . Un centimorgan (cM) es una unidad que describe una frecuencia de recombinación del 1%. De esta forma podemos medir la distancia genética entre dos loci, en función de su frecuencia de recombinación. Ésta es una buena estimación de la distancia real. Los cruces dobles no darían lugar a ninguna recombinación. En este caso no podemos decir si se produjeron cruces. Si los loci que estamos analizando están muy cerca (menos de 7 cM), es muy poco probable que se produzca un doble cruce. Cuando las distancias aumentan, aumenta la probabilidad de un doble cruce. A medida que aumenta la probabilidad de un doble cruce, se podría subestimar sistemáticamente la distancia genética entre dos loci, a menos que se utilice un modelo matemático apropiado.
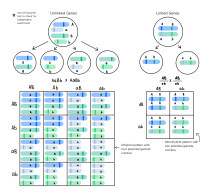
Durante la meiosis, los cromosomas se clasifican aleatoriamente en gametos , de modo que la segregación de los alelos de un gen es independiente de los alelos de otro gen. Así está establecido en la Segunda Ley de Mendel y se conoce como ley del surtido independiente . La ley de distribución independiente siempre es válida para genes que están ubicados en cromosomas diferentes, pero no siempre es válida para genes que están en el mismo cromosoma.
Como ejemplo de selección independiente, consideremos el cruce de la cepa parental homocigota de raza pura con genotipo AABB con una cepa de raza pura diferente con genotipo aabb . A y a y B y b representan los alelos de los genes A y B. El cruce de estas cepas parentales homocigotas dará como resultado una descendencia de la generación F1 que es doble heterocigota con genotipo AaBb. La descendencia F1 AaBb produce gametos que son AB , Ab , aB y ab con frecuencias iguales (25%) porque los alelos del gen A se seleccionan independientemente de los alelos del gen B durante la meiosis. Tenga en cuenta que 2 de los 4 gametos (50%) ( Ab y aB) no estaban presentes en la generación parental. Estos gametos representan gametos recombinantes . Los gametos recombinantes son aquellos gametos que difieren de los dos gametos haploides que formaron la célula diploide original . En este ejemplo, la frecuencia de recombinación es del 50% ya que 2 de los 4 gametos eran gametos recombinantes.
La frecuencia de recombinación será del 50% cuando dos genes se localicen en cromosomas diferentes o cuando estén muy separados en un mismo cromosoma. Esta es una consecuencia del surtido independiente.
Cuando dos genes están muy juntos en el mismo cromosoma, no se distribuyen de forma independiente y se dice que están vinculados. Mientras que los genes ubicados en diferentes cromosomas se agrupan de forma independiente y tienen una frecuencia de recombinación del 50%, los genes vinculados tienen una frecuencia de recombinación inferior al 50%.
Como ejemplo de vinculación, consideremos el experimento clásico de William Bateson y Reginald Punnett . [14] Estaban interesados en la herencia de rasgos en el guisante de olor y estaban estudiando dos genes: el gen del color de la flor ( P , púrpura y p , rojo) y el gen que afecta la forma de los granos de polen ( L , largo y l , redondo). Cruzaron las líneas puras PPLL y ppll y luego autocruzaron las líneas PpLl resultantes. Según la genética mendeliana , los fenotipos esperados se producirían en una proporción de 9:3:3:1 de PL:Pl:pL:pl. Para su sorpresa, observaron una mayor frecuencia de PL y pl y una disminución de la frecuencia de Pl y pL (consulte la tabla a continuación).
Su experimento reveló un vínculo entre los alelos P y L y los alelos p y l . La frecuencia de que P aparezca junto con L y de que p aparezca junto con l es mayor que la de Pl y pL recombinantes . La frecuencia de recombinación es más difícil de calcular en un cruce F2 que en un retrocruzamiento, [4] pero la falta de ajuste entre los números de progenie observados y esperados en la tabla anterior indica que es inferior al 50%.
En este caso, la descendencia recibió dos alelos dominantes unidos en un cromosoma (lo que se conoce como acoplamiento o disposición cis ). Sin embargo, después del cruce, alguna progenie podría haber recibido un cromosoma parental con un alelo dominante para un rasgo (p. ej., morado) vinculado a un alelo recesivo para un segundo rasgo (p. ej., redondo), mientras que ocurre lo contrario para el otro cromosoma parental (p. ej., rojo). y largo). Esto se conoce como repulsión o acuerdo trans . El fenotipo aquí seguiría siendo morado y largo, pero un cruce de prueba de este individuo con el progenitor recesivo produciría una progenie con una proporción mucho mayor de los dos fenotipos cruzados. Si bien tal problema puede no parecer probable en este ejemplo, aparecen vínculos de repulsión desfavorables cuando se mejoran para obtener resistencia a enfermedades en algunos cultivos.
Las dos posibles disposiciones, cis y trans, de los alelos en un heterocigoto doble se denominan fases gaméticas , y la fase gamética es el proceso de determinar cuál de los dos está presente en un individuo determinado.
Cuando dos genes están ubicados en el mismo cromosoma, la posibilidad de que un cruce produzca una recombinación entre los genes está relacionada con la distancia entre los dos genes. Así, el uso de frecuencias de recombinación se ha utilizado para desarrollar mapas de ligamiento o mapas genéticos .
Sin embargo, es importante señalar que la frecuencia de recombinación tiende a subestimar la distancia entre dos genes vinculados. Esto se debe a que a medida que los dos genes están ubicados más separados, también aumenta la posibilidad de que se produzcan el doble o incluso un número de cruces entre ellos. El doble o un número par de cruces entre los dos genes da como resultado su cosegregación en el mismo gameto, lo que produce una progenie parental en lugar de la progenie recombinante esperada. Como se mencionó anteriormente, las transformaciones de Kosambi y Haldane intentan corregir múltiples cruces. [15] [16]
A principios de la década de 1950, la opinión predominante era que los genes de un cromosoma son entidades discretas, indivisibles por recombinación genética y dispuestas como cuentas en un hilo. Entre 1955 y 1959, Benzer realizó experimentos de recombinación genética utilizando mutantes rII del bacteriófago T4 . Descubrió que, basándose en pruebas de recombinación, los sitios de mutación podían mapearse en orden lineal. [17] [18] Este resultado proporcionó evidencia de la idea clave de que el gen tiene una estructura lineal equivalente a una longitud de ADN con muchos sitios que pueden mutar de forma independiente.
Édgar et al. [19] realizaron experimentos de mapeo con mutantes r del bacteriófago T4 que muestran que las frecuencias de recombinación entre mutantes rII no son estrictamente aditivas. La frecuencia de recombinación de un cruce de dos mutantes rII (axd) suele ser menor que la suma de las frecuencias de recombinación para subintervalos internos adyacentes (axb) + (bxc) + (cxd). Aunque no es estrictamente aditiva, se observó una relación sistemática [20] que probablemente refleja el mecanismo molecular subyacente de la recombinación genética .
Si bien la recombinación de cromosomas es un proceso esencial durante la meiosis, existe un amplio rango de frecuencia de cruces entre organismos y dentro de especies. Las tasas de recombinación sexualmente dimórficas se denominan heteroquiasmia y se observan con más frecuencia que una tasa común entre hombres y mujeres. En los mamíferos, las hembras suelen tener una mayor tasa de recombinación en comparación con los machos. Se teoriza que existen selecciones únicas que actúan o impulsores meióticos que influyen en la diferencia de tasas. La diferencia en las tasas también puede reflejar los entornos y condiciones muy diferentes de la meiosis en la ovogénesis y la espermatogénesis. [21]
Las mutaciones en genes que codifican proteínas implicadas en el procesamiento del ADN a menudo afectan la frecuencia de recombinación . En el bacteriófago T4 , las mutaciones que reducen la expresión de la ADN polimerasa replicativa [producto génico 43 (gp43)] aumentan la recombinación (disminuyen el enlace) varias veces. [22] [23] El aumento en la recombinación puede deberse a errores de replicación cometidos por la ADN polimerasa defectuosa que son en sí mismos eventos de recombinación, como cambios de plantilla, es decir, eventos de recombinación de elección de copia. [24] La recombinación también aumenta mediante mutaciones que reducen la expresión de la ADN ligasa (gp30) [25] [23] y dCMP hidroximetilasa (gp42), [22] [23] dos enzimas empleadas en la síntesis de ADN .
La recombinación se reduce (el enlace aumenta) por mutaciones en genes que codifican proteínas con funciones nucleasas (gp46 y gp47) [25] [23] y una proteína de unión al ADN (gp32) [23] La mutación en el gen del bacteriófago uvsX también reduce sustancialmente la recombinación. . [26] El gen uvsX es análogo al bien estudiado gen recA de Escherichia coli que desempeña un papel central en la recombinación. [27]
Con pedigríes muy grandes o con datos de marcadores genéticos muy densos, como los de la secuenciación del genoma completo, es posible localizar recombinaciones con precisión. Con este tipo de análisis genético, se asigna un indicador de meiosis a cada posición del genoma para cada meiosis en un pedigrí. El indicador indica qué copia del cromosoma parental contribuye al gameto transmitido en esa posición. Por ejemplo, si se transmite el alelo de la "primera" copia del cromosoma parental, se podría asignar un "0" a esa meiosis. Si se transmite el alelo de la "segunda" copia del cromosoma parental, se asignaría un "1" a esa meiosis. Los dos alelos del progenitor procedían, uno de cada uno, de dos abuelos. Estos indicadores se utilizan luego para determinar estados de identidad por descendencia (IBD) o estados de herencia, que a su vez se utilizan para identificar genes responsables de enfermedades.