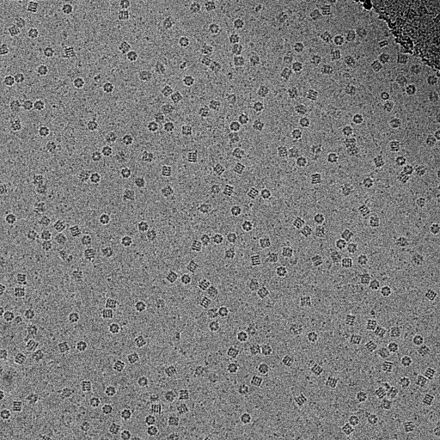
La criomicroscopía electrónica de transmisión ( CryoTEM ), comúnmente conocida como crio-EM , es una forma de microscopía electrónica criogénica , más específicamente un tipo de microscopía electrónica de transmisión (TEM) donde la muestra se estudia a temperaturas criogénicas (generalmente temperaturas de nitrógeno líquido ). [1] Cryo-EM, específicamente microscopía electrónica tridimensional ( 3DEM ), está ganando popularidad en biología estructural . [2]
La utilidad de la criomicroscopía electrónica de transmisión radica en que permite observar especímenes que no han sido teñidos ni fijados de ninguna forma, mostrándolos en su entorno nativo. Esto contrasta con la cristalografía de rayos X , que requiere cristalizar la muestra, lo que puede ser difícil, y colocarla en ambientes no fisiológicos, lo que ocasionalmente puede conducir a cambios conformacionales funcionalmente irrelevantes.
Los avances en la tecnología de detectores de electrones , en particular DED (detectores directos de electrones), así como algoritmos de imágenes de software más potentes, han permitido la determinación de estructuras macromoleculares con una resolución casi atómica. [3] Las macromoléculas fotografiadas incluyen virus , ribosomas , mitocondrias , canales iónicos y complejos enzimáticos . A partir de 2018, la crio-EM podría aplicarse a estructuras tan pequeñas como la hemoglobina (64 kDa ) [4] y con resoluciones de hasta 1,8 Å . [5] En 2019, las estructuras crio-EM representaron el 2,5% de las estructuras depositadas en el Protein Data Bank , [6] y este número sigue creciendo. [7] Una aplicación de la crio-EM es la tomografía crioelectrónica (crio-ET), donde se crea una reconstrucción 3D de la muestra a partir de imágenes 2D inclinadas.
El fundamento original de CryoTEM era un medio para combatir los daños causados por la radiación en especímenes biológicos. La cantidad de radiación requerida para recolectar una imagen de una muestra en el microscopio electrónico es lo suficientemente alta como para ser una fuente potencial de daño a la muestra en estructuras delicadas. Además, el alto vacío requerido en la columna de un microscopio electrónico hace que el entorno para la muestra sea bastante duro.
El problema del vacío se resolvió parcialmente mediante la introducción de tinciones negativas , pero incluso con tinciones negativas las muestras biológicas son propensas al colapso estructural tras la deshidratación de la muestra. Incrustar las muestras en hielo por debajo de la temperatura de sublimación fue una posibilidad que se contempló desde el principio, pero el agua tiende a organizarse en una red cristalina de menor densidad al congelarse y esto puede destruir la estructura de cualquier cosa que esté incrustada en ella.
A principios de la década de 1980, varios grupos que estudiaban la física del estado sólido intentaban producir hielo vítreo por diferentes medios, como la congelación a alta presión o la congelación instantánea. En un artículo fundamental de 1984, el grupo dirigido por Jacques Dubochet en el Laboratorio Europeo de Biología Molecular mostró imágenes de adenovirus incrustados en una capa de agua vitrificada. [8] Generalmente se considera que este artículo marca el origen de Cryo-EM, y la técnica se ha desarrollado hasta el punto de convertirse en rutina en numerosos laboratorios de todo el mundo.
La energía de los electrones utilizados para obtener imágenes (80 a 300 kV) es lo suficientemente alta como para que se puedan romper los enlaces covalentes . Cuando las muestras de imágenes son vulnerables al daño por radiación, es necesario limitar la exposición a los electrones utilizados para adquirir la imagen. Estas bajas exposiciones requieren que las imágenes de miles o incluso millones de moléculas congeladas idénticas sean seleccionadas, alineadas y promediadas para obtener mapas de alta resolución, utilizando software especializado. En 2012 se logró una mejora significativa en las características estructurales mediante la introducción de detectores de electrones directos y mejores algoritmos computacionales. [1] [2]
En 2015, Bridget Carragher y sus colegas del Scripps National Resource for Automated Molecular Microscopy utilizaron técnicas que ella y Clint Potter desarrollaron para determinar la primera estructura crio-EM con una resolución inferior a 3 Å, elevando así a CryoTEM como una herramienta comparable y potencialmente superior. a las técnicas tradicionales de cristalografía de rayos X. [9] [10] Desde entonces, se han logrado resoluciones más altas, incluida una estructura de 2,2 Å de la enzima bacteriana β-galactosidasa en 2015 [11] y una estructura de 1,8 Å de glutamato deshidrogenasa en 2016. [12] Cryo-EM también ha Se ha utilizado para determinar la estructura de varios virus, incluido el virus Zika , [13] y se ha aplicado a grandes complejos como el espliceosoma . [14] En 2017, el Premio Nobel de Química fue otorgado conjuntamente a Jacques Dubochet , Joachim Frank y Richard Henderson , "por el desarrollo de la microscopía crioelectrónica para la determinación estructural de alta resolución de biomoléculas en solución". [15]
El material biológico se extiende sobre una rejilla de microscopía electrónica y se conserva en estado congelado-hidratado mediante congelación rápida, generalmente en etano líquido cerca de la temperatura del nitrógeno líquido . Al mantener las muestras a la temperatura del nitrógeno líquido o más frías, se pueden introducir en el alto vacío de la columna del microscopio electrónico . La mayoría de los especímenes biológicos son extremadamente radiosensibles , por lo que se deben obtener imágenes con técnicas de dosis bajas (de manera útil, la baja temperatura de la criomicroscopía electrónica de transmisión proporciona un factor protector adicional contra el daño por radiación).
En consecuencia, las imágenes son extremadamente ruidosas . Para algunos sistemas biológicos es posible promediar imágenes para aumentar la relación señal-ruido y recuperar información de alta resolución sobre el espécimen utilizando la técnica conocida como análisis de partícula única . Este enfoque en general requiere que los elementos que se promedian sean idénticos, aunque ahora se puede estudiar cierta heterogeneidad conformacional limitada (por ejemplo, ribosoma ). Se han resuelto reconstrucciones tridimensionales a partir de imágenes CryoTEM de complejos proteicos y virus con resolución subnanométrica o casi atómica, lo que permite obtener nuevos conocimientos sobre la estructura y la biología de estos grandes conjuntos.
El análisis de conjuntos ordenados de proteínas, como cristales bidimensionales de proteínas transmembrana o conjuntos helicoidales de proteínas, también permite un tipo de promedio que puede proporcionar información de alta resolución sobre la muestra. Esta técnica se llama cristalografía electrónica .
El método de película delgada se limita a muestras delgadas (típicamente <500 nm) porque los electrones no pueden cruzar muestras más gruesas sin múltiples eventos de dispersión. Las muestras más gruesas se pueden vitrificar mediante congelación por inmersión ( criofijación ) en etano (hasta decenas de μm de espesor) o, más comúnmente, mediante congelación a alta presión (hasta cientos de μm). Luego se pueden cortar en secciones delgadas (de 40 a 200 nm de espesor) con una cuchilla de diamante en un crioultramicrotomo a temperaturas inferiores a -135 °C (temperatura de desvitrificación). Las secciones se recogen en una rejilla de microscopio electrónico y se obtienen imágenes de la misma manera que una muestra vitrificada en una película delgada. Esta técnica se denomina criomicroscopía electrónica de transmisión de cortes vítreos (CEMOVIS) o criomicroscopía electrónica de transmisión de cortes hidratados congelados.
Además de permitir la obtención de imágenes de muestras biológicas vitrificadas, CryoTEM también se puede utilizar para obtener imágenes de muestras de materiales que son demasiado volátiles en el vacío para obtener imágenes mediante microscopía electrónica estándar a temperatura ambiente. Por ejemplo, se pueden extraer secciones vitrificadas de interfaces líquido-sólido para su análisis mediante CryoTEM [16] y el azufre, que es propenso a la sublimación en el vacío de los microscopios electrónicos, se puede estabilizar y obtener imágenes en CryoTEM. [17]
Aunque en la mayoría de los métodos de microscopía electrónica se intenta obtener la imagen de mejor resolución del material, no siempre es así en crio-TEM. Además de todos los beneficios de las imágenes de alta resolución, la relación señal-ruido sigue siendo el principal obstáculo que impide asignar orientación a cada partícula. Por ejemplo, en los complejos de macromoléculas, hay varias estructuras diferentes que se proyectan de 3D a 2D durante la obtención de imágenes y, si no se distinguen, el resultado del procesamiento de imágenes será borroso. Es por ello que los enfoques probabilísticos cobran más fuerza en este tipo de investigaciones. [18] Hay dos enfoques populares que se utilizan ampliamente hoy en día en el procesamiento de imágenes crio-EM, el enfoque de máxima verosimilitud que se descubrió en 1998 [19] y el enfoque bayesiano adaptado relativamente recientemente. [20]
El enfoque de estimación de máxima verosimilitud llega a este campo desde la estadística. Aquí, se suman todas las orientaciones posibles de las partículas para obtener la distribución de probabilidad resultante. Podemos comparar esto con una estimación de mínimos cuadrados típica donde las partículas obtienen orientaciones exactas por imagen. [21] De esta manera, las partículas de la muestra obtienen orientaciones "borrosas" después de los cálculos, ponderadas por las probabilidades correspondientes. Todo el proceso es iterativo y con cada iteración siguiente el modelo mejora. Las buenas condiciones para realizar un modelo que represente fielmente la estructura real es cuando los datos no tienen demasiado ruido y las partículas no tienen ninguna dirección preferencial. La principal desventaja del enfoque de máxima verosimilitud es que el resultado depende de la suposición inicial y la optimización del modelo a veces puede quedarse estancada en el mínimo local. [22]
El enfoque bayesiano que se utiliza actualmente en crio-TEM es empírico por naturaleza. Esto significa que la distribución de partículas se basa en el conjunto de datos original. De manera similar, en el método bayesiano habitual existe una probabilidad previa fija que cambia después de observar los datos. La principal diferencia con la estimación de máxima verosimilitud radica en un término de reconstrucción especial que ayuda a suavizar los mapas resultantes y al mismo tiempo reduce el ruido durante la reconstrucción. [21] El suavizado de los mapas se produce asumiendo que la probabilidad anterior es una distribución gaussiana y analizando los datos en el espacio de Fourier. Dado que se establece la conexión entre el conocimiento previo y el conjunto de datos, hay menos posibilidades de que se produzcan errores de factor humano, lo que potencialmente aumenta la objetividad de la reconstrucción de imágenes. [20]
Con la aparición de nuevos métodos de obtención de imágenes crio-TEM y reconstrucción de imágenes, aparecen nuevas soluciones de software que ayudan a automatizar el proceso. Después de que se implementó el enfoque bayesiano empírico en el programa informático de código abierto RELION (REgularized LIkelihood OptimizatioN) para la reconstrucción 3D, [23] [24] el programa se generalizó en el campo crio-TEM. Ofrece una serie de correcciones que mejoran la resolución de las imágenes reconstruidas, permite implementar scripts versátiles utilizando lenguaje Python y ejecuta las tareas habituales de clasificación de modelos 2D/3D o creación de modelos de novo . [25] [26]
En CryoTEM se pueden utilizar una variedad de técnicas. [27] Las técnicas populares incluyen: