Potyvirus es un género de virus de ARN de cadena positiva (nombrado por su especie tipo, Potato virus Y (PVY) ) en la familia Potyviridae . Las plantas sirven como huéspedes naturales. Al igual que los begomovirus , los miembros de este género pueden causar pérdidas significativas en cultivos agrícolas, pastorales, hortícolas y ornamentales . Más de 200 especies de pulgones propagan potyvirus, y la mayoría son de la subfamilia Aphidinae (géneros Macrosiphum y Myzus ). [ cita requerida ] El género contiene 190 especies y los potyvirus representan aproximadamente el treinta por ciento de todos los virus de plantas conocidos actualmente . [1] [2]
El virión no tiene envoltura y posee una nucleocápside flexuosa y filamentosa , de 680 a 900 nanómetros (nm) de largo y de 11 a 20 nm de diámetro. [1] La nucleocápside contiene alrededor de 2000 copias de la proteína de la cápside. La simetría de la nucleocápside es helicoidal con un paso de 3,4 a 3,5 nm. [1]

El genoma es un ARN monocatenario , lineal y de sentido positivo cuyo tamaño varía entre 9000 y 12 000 bases de nucleótidos . La mayoría de los potyvirus tienen genomas no segmentados, [1] aunque varias especies son bipartitas. Las composiciones de bases típicas de algunas de las cepas no recombinantes más comunes de la especie tipo, PVY, varían entre ~23,4-23,8 % G; ~31-31,6 % A; ~18,2-18,8 % C; y ~26,5-26,8 % U. [3]
En las especies con un genoma monopartito, una proteína VPg ligada al genoma está unida covalentemente al extremo 5' y el extremo 3' está poliadenilado. El genoma codifica un único marco de lectura abierto (ORF) expresado como un precursor de poliproteína de 350 kDa. Esta poliproteína se procesa en diez proteínas más pequeñas: proteína 1 proteasa (P1-Pro), componente auxiliar proteasa (HC-Pro), proteína 3 (P3), inclusión cilíndrica (CI), proteína viral ligada al genoma (Vpg), inclusión nuclear A (NIa), inclusión nuclear B (NIb), proteína de la cápside (CP) y dos pequeñas proteínas putativas conocidas como 6K1 y 6K2. El cistrón P3 también contiene un marco de lectura superpuesto llamado "Pretty interesting Potyviridae ORF " (PIPO). [4] PIPO codifica un extremo C alternativo a la proteína P3, que se genera en un subconjunto de transcripciones por un desplazamiento del marco de lectura +2 causado por un mecanismo de deslizamiento de ribosomas en una secuencia de repetición GA 6 conservada . [5] [6] La proteína resultante se llama P3N-PIPO. Se cree que un mecanismo similar produce un marco de lectura alternativo dentro del cistrón P1, llamado "ORF del potyvirus de la batata bastante interesante" (PISPO), en varios potyvirus que infectan la batata, incluido el virus del moteado plumoso de la batata . [7]
P1 (~33 kilo daltons (kDa) de peso molecular) es una serina proteasa que facilita su propia escisión de la poliproteína en la unión P1-HC-Pro. [8] P1 consta de un dominio de proteasa C-terminal conservado y una región N-terminal que tiene un alto nivel de variación en secuencia y longitud entre especies de potyvirus pero exhibe patrones conservados de desorden intrínseco . P1 también promueve la replicación del ARN viral, aunque no es necesario para ello. [9]
HC-Pro (~52 KDa) es una proteasa de cisteína que escinde un dipéptido glicina -glicina en su propio extremo C. [8] También interactúa con el factor de iniciación eucariota 4 (eIF4) .Actúa como un supresor del silenciamiento del ARN viral a través de sus interacciones con las proteínas AGO del huésped . [10] La actividad de HC-Pro está regulada por la proteína P1 adyacente: antes de que P1 se escinda del intermediario P1-HC-Pro, el extremo P1 reduce la actividad de supresión del silenciamiento del ARN de HC-Pro. [8] Por lo tanto, la tasa de escisión de P1 regula el nivel de supresión de la interferencia del ARN durante la infección. HC-Pro también está involucrado en la transmisión de pulgones. [11] Aunque se desconoce el mecanismo exacto, se ha propuesto que HC-Pro se adhiere a las partes bucales del pulgón huésped a través de su dominio similar a un dedo de zinc N-terminaly ancla viriones a través de sus interacciones con la proteína de la cápside. [12]
P3 (~41 kDa) es una proteína de membrana necesaria para la replicación viral y se acumula en vesículas de replicación viral. [13] Media las interacciones entre vesículas de replicación y proteínas del complejo de movimiento, lo que puede permitir que las vesículas de replicación sean reclutadas al complejo de movimiento para un movimiento intercelular eficiente. [14] P3 también interactúa con la subunidad grande de la ribulosa-1,5-bisfosfato carboxilasa/oxigenasa . [ cita requerida ]
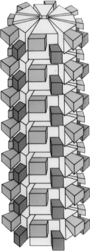
CI (~71 kDa) es una helicasa de ARN con actividad ATPasa . [15] Su propiedad más inusual es su capacidad para formar inclusiones cónicas y cilíndricas grandes y altamente simétricas con un cilindro hueco central desde el cual las láminas laminadas irradian hacia afuera y se pliegan sobre sí mismas en un patrón a menudo descrito como " ruedas de molino ". Estas inclusiones se ven fácilmente en micrografías electrónicas de transmisión de tejidos infectados y se usaron históricamente como un criterio de diagnóstico para infecciones por potyvirus. Las inclusiones de CI son un componente principal del complejo de movimiento potyviral que se ensambla en los plasmodesmos . CI también es necesario para la replicación viral y está presente en las membranas de replicación. Sus contribuciones exactas a la replicación no están claras pero, como helicasa de ARN, es probable que CI facilite la replicación al desmantelar las estructuras secundarias del ARN viral.
El NIa (~50 kDa) forma inclusiones cristalinas en el núcleo del huésped y se escinde en NIa-Pro y VPg.
NIa-Pro (~27 kDa) es una proteasa de cisteína que procesa la mayoría de los sitios de escisión de la poliproteína. [16] Las únicas excepciones son las autoescisiones de P1 y HC-Pro. El alto grado de especificidad y conservación de la secuencia de escisión ha hecho que NIa-Pro (a menudo la del virus del grabado del tabaco ) sea una herramienta valiosa en biotecnología, especialmente en aplicaciones que requieren la eliminación de etiquetas de afinidad de proteínas recombinantes después de la purificación por afinidad . NIa-Pro también ha demostrado exhibir actividad de DNasa independiente de la secuencia e interferir con la metilación del ADN del huésped , lo que sugiere que NIa y/o NIa-Pro están alterando la expresión génica del huésped . [17] La NIa-Pro potyviral comparte un alto nivel de homología con la proteasa 3C picornaviral . [18]
La VPg (~22 kDa) está unida covalentemente al extremo 5' del ARN genómico viral a través de uridilación y se cree que actúa como un cebador para la replicación del genoma viral de manera similar a las proteínas VPg de picornaviridae . [19] Es una proteína altamente desordenada y se ha sugerido que su flexibilidad le permite interactuar con muchas otras proteínas virales. La VPg también interactúa con varias proteínas del huésped, incluido el factor de iniciación eucariota 4E (eIF4E), el factor de elongación eucariota 1A (eEF1A) y la proteína de unión a poli(A) (PABP). [20] [21]
NIb (~59 kDa) es una ARN polimerasa dependiente de ARN de la superfamilia II (RdRp) que polimeriza el ARN viral durante la replicación. [22] Al igual que NIa, NIb forma inclusiones en el núcleo del huésped donde es transportado debido a sus dos secuencias de localización nuclear . NIb tiene la estructura de tres dominios "palma, pulgar y dedos" típica de RdRps.
6K1 (~6 kDa): la función no se conoce, pero debido a que se acumula en vesículas de replicación y tiene un dominio transmembrana , se cree que 6K1 contribuye a la formación de vesículas inducida por el virus. [23]
6K2 (~6 kDa) es una proteína transmembrana que reorganiza las membranas del huésped en estructuras de membrana inducidas por el virus. [24] Interactúa con varias proteínas del sitio de salida del RE para producir extensiones vesiculares y tubulares que eventualmente maduran en vesículas de replicación. [25] 6K2 tiene tres dominios principales: el dominio N-terminal que es necesario para el movimiento de célula a célula, la hélice alfa transmembrana hidrofóbica central y el dominio C-terminal que es necesario para la replicación viral. [26]
P3N-PIPO (~25 kDa) es una proteína de movimiento dedicada que ancla el complejo de movimiento al plasmodesmo . [27] También puede modular el límite de exclusión de tamaño del plasmodesmo al interactuar con proteínas del huésped que cortan los filamentos de actina del plasmodesmo y reducen la deposición de calosa . [28] [29] Interactúa con las subunidades grandes y pequeñas de la ribulosa-1,5-bisfosfato carboxilasa/oxigenasa. [ cita requerida ]
CP (~30 - 35 kDa) es la proteína de la cápside. Tiene dos dominios terminales que están desordenados y expuestos en la superficie del virión. [30] [31] El dominio central contiene un bolsillo de unión de ARN que se une al ARN viral. La estructura de la proteína de la cápside está altamente conservada en los potyvirus, aunque hay un grado relativamente alto de variabilidad de secuencia. Además de encapsidar el virión, el dominio central CP es necesario para el movimiento intercelular y contribuye a la transmisión de la semilla. [32]
Ciertos potyvirus atípicos codifican proteínas adicionales o dominios proteicos, como P1-PISPO, Alkylation B (AlkB) y la inosina trifosfato pirofosfatasa (conocida como ITPasa o HAM1). [33] Estas anomalías suelen estar situadas en la región hipervariable P1-HC-Pro. [8]

La mayoría de los potyvirus son transmitidos por los pulgones cuando exploran los tejidos de las plantas con su estilete mientras se alimentan. [34] No circulan ni se multiplican dentro del pulgón y, por lo general, solo persisten en él durante unos minutos. Se ha demostrado que ciertos potyvirus alteran los patrones de alimentación de sus vectores pulgones, lo que puede manifestarse como períodos más prolongados de tiempo en las plantas infectadas, menor tiempo de alimentación sin exploración y mayor ingestión de savia del floema.
Se ha documentado la transmisión de semillas y polen en ciertas especies de potyvirus, por ejemplo en PVY y el virus del mosaico del nabo (TUMV) . [35] La transmisión vegetativa por tubérculos infectados o material de injerto es de particular preocupación para ciertos cultivos agrícolas, como la papa y los árboles frutales, respectivamente.
La transmisión también puede ocurrir por contacto físico con plantas infectadas o con herramientas, ropa o incluso agua contaminadas. [36]
Después de la entrada, las partículas de potyvirus quedan sin recubrimiento y el ARN genómico se libera en el citoplasma del huésped. El ARN del potyvirus imita al ARNm del huésped : la proteína VPg 5' comparte similitudes funcionales con la tapa 5' y el extremo 3' está poliadenilado . [37] VPg y sus interacciones con eIF4E y eIF4(iso)E permiten que el virus utilice la maquinaria de traducción dependiente de la tapa del huésped para su traducción. De manera similar a la traducción eucariota, la interacción VPg-eIF4E ensambla el complejo eIF4F alrededor del ARN viral.
Se han identificado varios sitios de entrada internos débiles a los ribosomas (IRES) en muchas especies de potyvirus, pero no se sabe si la traducción independiente del capuchón es un mecanismo de traducción importante para los potyvirus. [37]
Al igual que muchos otros virus de ARN de cadena positiva , la replicación de potyvirus está fuertemente asociada con las membranas del huésped. [1] [38] La proteína viral 6K2 coordina la reorganización de las membranas del huésped en varias estructuras asociadas a la infección que, dependiendo de la especie de potyvirus, pueden incluir cualquier cosa desde pequeñas vesículas virales redondas hasta estructuras globulares complejas con muchas cisternas o lóbulos. Estas estructuras están salpicadas de complejos de replicación viral y a menudo se denominan "vesículas de replicación", "viroplasma" o "fábricas virales". Las membranas de las vesículas de replicación se derivan de una variedad de orgánulos del huésped y las fuentes difieren entre las especies de potyvirus. Algunas fuentes de membrana incluyen el RE , los cloroplastos , el aparato de Golgi y las vacuolas .
No se conoce el mecanismo exacto de replicación, pero implica un ARN intermediario de sentido negativo y requiere proteínas virales y del huésped. Las proteínas virales detectadas en complejos de replicación incluyen HC-Pro, P3, 6K1, 6K2, CI, VPg, NIa-Pro y NIb. [39] Los factores del huésped presentes en las vesículas de replicación incluyen eIF4A y varias proteínas de choque térmico .
Al igual que la mayoría de los virus de plantas, los potyvirus han evolucionado para moverse de una célula vegetal a otra a través de plasmodesmos . Sin embargo, a diferencia de algunos virus de plantas bien estudiados, como el virus del mosaico del tabaco , los potyvirus no tienen una sola proteína de movimiento , sino que ensamblan un complejo de movimiento alrededor del plasmodesmo. [40] Este complejo está compuesto principalmente por tres proteínas virales: CI, CP, P3N-PIPO. Las inclusiones cónicas de CI están ancladas a los plasmodesmos por P3N-PIPO durante las primeras etapas de la infección por potyvirus. Esto permite que la inclusión canalice partículas virales o complejos virales de ARN-CP a través del plasmodesmo. Las vesículas de replicación también se reclutan al complejo de movimiento, lo que sugiere que la replicación y el movimiento están acoplados. Las vesículas de replicación son reclutadas por P3N-PIPO, que interactúa tanto con CI como con P3 a través del dominio P3N compartido. [14] La interacción de P3 con 6K2 permite que las vesículas de replicación se unan al complejo de movimiento.
Los potyvirus evolucionaron hace entre 6.600 y 7.250 años. [41] [42] Parece que evolucionaron en el sudoeste de Eurasia o el norte de África . La tasa de mutación estimada es de aproximadamente 1,15 × 10 −4 sustituciones de nucleótidos/sitio/año. [ cita requerida ]
La agricultura se introdujo en Australia en el siglo XVIII. Esta introducción también incluyó patógenos vegetales. Se han aislado treinta y ocho especies de potyvirus en Australia. Dieciocho de ellos se han encontrado solo en Australia y se presume que son endémicos allí. Los veinte restantes parecen haber sido introducidos con la agricultura. [ cita requerida ]
Históricamente, el diagnóstico de potyvirus se basaba en la detección de diversas inclusiones proteínicas en las células vegetales infectadas. Estas pueden aparecer como cristales en el citoplasma o en el núcleo, como cuerpos X amorfos, cuerpos membranosos, viroplasmas o remolinos. [43] Las inclusiones pueden contener o no (según la especie) viriones. [ cita requerida ] Estas inclusiones se pueden ver mediante microscopía óptica en tiras de hojas de tejido vegetal infectado teñidas con naranja-verde (tinción de proteínas) pero no con azul A (tinción de ácido nucleico). [44] [45] [46]
Los métodos de detección modernos se basan principalmente en la PCR de transcripción inversa . [47]
El potyvirus contiene las siguientes especies: [2]
Anteriormente se habían clasificado otros cuatro virus como especies de este género, pero se eliminaron debido a la falta de información sobre la secuencia genética: [48]
Los potyvirus se dividieron en los grupos de especies PVY, SCMV, BYMV y BCMV en 1992. Gibbs y Ohshima en 2010 produjeron una filogenia molecular más extensa con los mismos cuatro grupos, pero también con varios grupos nuevos: BtMV, ChVMV, DaMV, OYDV, PRSV, TEV y TuMV. [42]
Contiene 16 especies, incluida la especie tipo del género (virus Y de la papa). Los hospedadores primarios son: nueve Solanaceae , tres Amaranthus , tres Asteraceae , un Lilium y un Amaryllis . [42]