Paramyxoviridae (del griego para- “al lado de” y myxa “ moco ”) es una familia de virus de ARN de cadena negativa en el orden Mononegavirales . [1] [2] Los vertebrados sirven como huéspedes naturales. [3] Las enfermedades asociadas con esta familia incluyen sarampión , paperas e infecciones del tracto respiratorio . [4] La familia tiene cuatro subfamilias, 17 géneros, tres de los cuales no están asignados a una subfamilia, y 78 especies. [5]
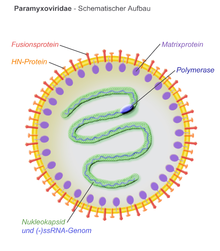
Los viriones tienen una envoltura y pueden ser esféricos o pleomórficos y capaces de producir viriones filamentosos. El diámetro es de alrededor de 150 nm. Los genomas son lineales, de alrededor de 15 kb de longitud. [6] [1] Las proteínas de fusión y las proteínas de unión aparecen como espigas en la superficie del virión. Las proteínas de la matriz dentro de la envoltura estabilizan la estructura del virus. El núcleo de la nucleocápside está compuesto por el ARN genómico, las proteínas de la nucleocápside, las fosfoproteínas y las proteínas de la polimerasa.

El genoma es ARN no segmentado, de sentido negativo, de 15 a 19 kilobases de longitud y contiene de seis a diez genes. Las regiones extracistrónicas (no codificantes) incluyen:
Cada gen contiene señales de inicio/parada de la transcripción al principio y al final, que se transcriben como parte del gen.
La secuencia de genes dentro del genoma se conserva en toda la familia debido a un fenómeno conocido como polaridad transcripcional (ver Mononegavirales ) en el que los genes más cercanos al extremo 3' del genoma se transcriben en mayor abundancia que los que están hacia el extremo 5'. Esto es resultado de la estructura del genoma. Después de que se transcribe cada gen, la ARN polimerasa dependiente de ARN hace una pausa para liberar el nuevo ARNm cuando encuentra una secuencia intergénica. Cuando la ARN polimerasa se detiene, existe la posibilidad de que se disocie del genoma de ARN. Si se disocia, debe volver a ingresar al genoma en la secuencia líder, en lugar de continuar transcribiendo la longitud del genoma. El resultado es que cuanto más alejados estén los genes de la secuencia líder, menos serán transcritos por la ARN polimerasa.
La evidencia de un modelo de promotor único se verificó cuando los virus se expusieron a la luz ultravioleta. La radiación ultravioleta puede causar la dimerización del ARN, lo que impide la transcripción por la ARN polimerasa. Si el genoma viral sigue un modelo de promotor múltiple, el nivel de inhibición de la transcripción debería correlacionarse con la longitud del gen de ARN. Sin embargo, el genoma se describió mejor mediante un modelo de promotor único. Cuando el genoma del paramixovirus se expuso a la luz ultravioleta, el nivel de inhibición de la transcripción fue proporcional a la distancia desde la secuencia líder. Es decir, cuanto más lejos está el gen de la secuencia líder, mayor es la probabilidad de que la dimerización del ARN inhiba la ARN polimerasa.
El virus aprovecha el modelo de promotor único al disponer sus genes en orden relativo a la proteína necesaria para una infección exitosa. Por ejemplo, la proteína de la nucleocápside, N, es necesaria en mayores cantidades que la ARN polimerasa, L.
Los virus de la familia Paramyxoviridae también son antigénicamente estables, lo que significa que las glicoproteínas de los virus son consistentes entre diferentes cepas del mismo tipo. Se postulan dos razones para este fenómeno: la primera es que el genoma no está segmentado, por lo que no puede sufrir reordenamiento genético . Para que ocurra este proceso, los segmentos necesarios para el reordenamiento ocurren cuando los segmentos de diferentes cepas se mezclan para crear una nueva cepa. Sin segmentos, nada se puede mezclar entre sí, por lo que no se produce un cambio antigénico . La segunda razón se relaciona con la idea de la deriva antigénica . Dado que la ARN polimerasa dependiente de ARN no tiene una función de verificación de errores, se realizan muchas mutaciones cuando se procesa el ARN. Estas mutaciones se acumulan y, finalmente, se crean nuevas cepas. Debido a este concepto, uno esperaría que los paramixovirus no fueran antigénicamente estables; sin embargo, se ve que es cierto lo contrario. La hipótesis principal detrás de por qué los virus son antigénicamente estables es que cada proteína y aminoácido tiene una función importante. De esta forma, cualquier mutación provocaría una disminución o pérdida total de la función, lo que a su vez haría que el nuevo virus fuera menos eficiente y no pudiera sobrevivir tanto tiempo como las cepas más virulentas, por lo que se extinguiría.
Muchos genomas de paramixovirus siguen la "regla de seis" . La longitud total del genoma es casi siempre un múltiplo de seis. Esto se debe probablemente a la ventaja de tener todo el ARN unido por la proteína N (ya que N se une a los hexámeros del ARN). Si el ARN queda expuesto, el virus no se replica de manera eficiente. La secuencia del gen es:

La replicación viral es citoplasmática . La entrada en la célula huésped se logra mediante la unión viral a la célula huésped. La replicación y la transcripción siguen los modelos de virus de ARN de cadena negativa . [7] La traducción se lleva a cabo mediante escaneo con fugas, derivación ribosómica y terminación-reiniciación del ARN. El virus sale de la célula huésped mediante gemación. Los vertebrados, incluidos los humanos y las aves, sirven como huéspedes naturales. La vía de transmisión son partículas transportadas por el aire. [1]
Los Paramyxoviridae pueden experimentar edición de ARNm, que produce diferentes proteínas a partir del mismo transcrito de ARNm al retroceder una base para leer en un marco de lectura abierto ( ORF ) diferente debido a la presencia de estructuras secundarias como pseudonudos. Los Paramyxoviridae también experimentan tartamudeo transcripcional para producir la cola de poli (A) al final de las transcripciones de ARNm al retroceder repetidamente un nucleótido a la vez al final de la plantilla de ARN. [8] [9]

Familia: Paramyxoviridae [5]
Los paramixovirus causan una serie de enfermedades humanas importantes, entre ellas las paperas y el sarampión , que causó alrededor de 136.200 muertes en 2022. [11]
Los virus parainfluenza humanos (HPIV) son la segunda causa más común de enfermedades de las vías respiratorias en bebés y niños. Hay cuatro tipos de HPIV, conocidos como HPIV-1, HPIV-2, HPIV-3 y HPIV-4. El HPIV-1 y el HPIV-2 pueden causar síntomas similares a los del resfriado, junto con crup en niños. El HPIV-3 se asocia con bronquiolitis , bronquitis y neumonía . El HPIV-4 es menos común que los otros tipos y se sabe que causa enfermedades de las vías respiratorias de leves a graves. [12]
Los paramixovirus también son responsables de una variedad de enfermedades en otras especies animales, por ejemplo , el virus del moquillo canino ( perros ), el virus del moquillo focino ( focas ), el morbilivirus de los cetáceos ( delfines y marsopas ), el virus de la enfermedad de Newcastle ( aves ) y el virus de la peste bovina ( ganado ).
Algunos paramixovirus, como los henipavirus , son patógenos zoonóticos que se producen de forma natural en un huésped animal, pero que también pueden infectar a los seres humanos. El virus Hendra y el virus Nipah del género Henipavirus han surgido en humanos y ganado en Australia y el sudeste asiático . Ambos virus son contagiosos , altamente virulentos y capaces de infectar a varias especies de mamíferos y causar enfermedades potencialmente mortales. Debido a la falta de una vacuna humana autorizada (existe una vacuna contra el virus Hendra para caballos) o terapias antivirales, el virus Hendra y el virus Nipah se designan como agentes de nivel de bioseguridad (BSL) 4. La estructura genómica de ambos virus es la de un paramixovirus típico. [13]
En las últimas décadas, [¿ cuándo? ] se han descubierto paramixovirus en animales terrestres, voladores y acuáticos, lo que demuestra una amplia gama de hospedadores y una gran diversidad genética viral. A medida que avanza la tecnología molecular y se implementan programas de vigilancia viral, aumenta el descubrimiento de nuevos virus en este grupo. [4]
La evolución de los paramixovirus todavía se debate. Utilizando los neumovirus (familia mononegaviral Pneumoviridae ) como grupo externo, los paramixovirus se pueden dividir en dos clados: uno que consiste en avulavirus y rubulavirus y otro que consiste en respirovirus , henipavirus y morbillivirus . [14] Dentro del segundo clado, los respirovirus parecen ser el grupo basal. El clado respirovirus-henipavirus-morbillivirus puede ser basal al clado avulavirus-rubulavirus.