El ortoreóvirus de los mamíferos (MRV) es un virus de ARN bicatenario . Forma parte de la familia Reoviridae , así como de la subfamilia Spinareovirinae . Como se ve en el nombre, el ortoreóvirus de los mamíferos infecta a numerosas especies de mamíferos y vertebrados que sirven como huéspedes naturales. Algunas enfermedades que ocurren como resultado de este virus o están asociadas con este virus incluyen enfermedad leve de las vías respiratorias superiores y enfermedad gastrointestinal. Ejemplos de estas son: síndromes del tracto respiratorio superior, gastroenteritis (gripe estomacal), atresia biliar , hidrocefalia obstructiva , ictericia , alopecia , conjuntivitis y "cabello graso" asociado con esteatorrea . [3] [4]
Existen cuatro serotipos , o variaciones distintas dentro de la especie viral, del orthoreovirus de mamíferos. Esto significa que hay cuatro cepas distintas de orthoreovirus de mamíferos: tipo 1 Lang, tipo 2 Jones, tipo 3 Dearing, tipo 4 Ndelle. Un ejemplo de estas cepas es el orthoreovirus de mamíferos tipo 3, u orthoreovirus de mamíferos 3-Dearing (MRV-3; cepa Dearing). Induce la muerte celular preferentemente en células transformadas y, por lo tanto, muestra propiedades oncolíticas inherentes. [4] [5]
Se cree que el orthoreovirus de mamíferos causa infecciones subclínicas en humanos más que en otros mamíferos. Esto significa que un humano infectado con orthoreovirus de mamíferos, independientemente del serotipo , es casi o completamente asintomático y, por lo tanto, no presenta signos ni síntomas del virus. Esta teoría se basa en evidencia de que la mayoría de las personas tienen anticuerpos para todos los serotipos, lo que significa que estuvieron expuestas al virus en algún momento y el sistema inmunológico del cuerpo desarrolló inmunidad contra él después de ser infectadas. [3]

Según el esquema de clasificación de Baltimore , el orthoreovirus de mamíferos es un virus del Grupo III . Esto se debe a que el orthoreovirus de mamíferos es un virus de ARN bicatenario y, como se dijo anteriormente, forma parte de la familia Reovirus . El nombre "Reo-" se deriva de enfermedades huérfanas entéricas respiratorias . El término "enfermedades huérfanas" hace referencia al hecho de que algunos Reovirus no se sabía que estuvieran asociados con ninguna enfermedad en el momento de su descubrimiento. La familia Reoviridae también se caracteriza por tener cápsides icosaédricas "desnudas" y 10-12 segmentos de ARN bicatenario lineal . "Desnudo" significa que la cápside icosaédrica no está rodeada por una envoltura viral. [6]
Dentro de la familia Reoviridae , el orthoreovirus de mamíferos forma parte de la subfamilia Spinareovirinae . Esto se basa en el hecho de que los virus dentro de esta subfamilia, como el orthoreovirus de mamíferos , Aquareovirus , Coltivirus , Cypovirus , etc., tienen la presencia de una proteína en torreta en la cápside interna. [5] [7]

La etimología del orthoreovirus de mamíferos se basa en "orto-" traducido del griego como "recto" y "reovirus" de virus huérfano entérico respiratorio . [ 7] El orthoreovirus de mamíferos fue etiquetado como un virus huérfano en la década de 1950 cuando fue descubierto. Fue descrito como una "presencia ubicua" en mamíferos, lo que significa que se encontró prácticamente en todas partes. Las encuestas de suero realizadas en la década de 1950 encontraron anticuerpos neutralizantes al orthoreovirus de mamíferos en humanos , monos , conejos y cobayas . Estudios más recientes sugieren que el MRV sigue siendo una presencia ubicua en humanos . El orthoreovirus de mamíferos está bien investigado y entendido en los niveles bioquímico y estructural, además, su patogénesis en ratones sirve como un sistema modelo para estudiar la patogénesis de los reovirus en general. El género Orthoreovirus se subdivide en fusogénico y no fusogénico. La división se basa en que los orthoreovirus fusogénicos tienen la capacidad de provocar la fusión de células infectadas, lo que da lugar a sincitios celulares multinucleados. Estos orthoreovirus fusogénicos codifican una proteína transmembrana pequeña asociada a la fusión (FAST) que desempeña un papel en esta capacidad. Los orthoreovirus de Mammailian prototípicos no son fusogénicos y no producen sincitios debido a la falta de la proteína transmembrana asociada a la fusión. [4] [7]
Los serotipos de orthoreovirus de mamíferos se han estudiado significativamente a nivel antigénico y de secuencia. Los serotipos 1 (Lang), 2 (Jones) y 3 (Dearing) se reconocieron originalmente basándose en perfiles de neutralización del virus e inhibición de la hemaglutinación. El serotipo 4 (Ndelle) se ha estudiado a nivel antigénico y molecular. [4] [6] [8]
Al igual que el resto de reovirus, el orthoreovirus de mamíferos contiene un genoma segmentado con una disposición genómica lineal, que está encerrado en una cápside proteica de doble capa de 70-80 nm , formada por una capa interna (T = 2) y una capa externa (T = 13). El núcleo viral del MRV, y de los orthoreovirus por igual, consiste en una capa de cápside interna más su genoma viral encerrado . El genoma de ARN bicatenario del orthoreovirus de mamíferos contiene 10 segmentos divididos en tres clases de tamaño (pequeño, mediano y grande) según su movilidad característica durante la electroforesis en gel. El genoma tiene tres segmentos grandes (L1, L2 y L3) que codifican proteínas λ (lambda); tres segmentos medianos (M1, M2 y M3) que codifican proteínas μ (mu); y cuatro segmentos pequeños (S1, S2, S3 y S4) que codifican proteínas σ (sigma). En total, el tamaño del genoma es de aproximadamente 23.500 pares de bases. [6] [7] [8]
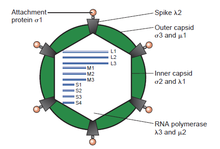
Generalmente, estos segmentos genómicos de ARN bicatenario contienen un solo gen que codifica una sola proteína, aunque hay algunas excepciones. El segmento genómico S1 de los reovirus no fusogénicos (p. ej., ortoreovirus de mamíferos) codifica la proteína de unión celular σ1. Junto con el gen σ1 hay un segundo marco de lectura abierto más pequeño que codifica los σ1 no estructurales. El ARN bicatenario de los reovirus no puede servir como plantilla para la traducción de proteínas o como ARN mensajero ( ARNm ). Debido a la naturaleza del ARN bicatenario , los virus de ARN bicatenario como los reovirus y los ortoreovirus de mamíferos deben transportar y/o codificar las enzimas necesarias dentro de sus viriones para transcribir primero su genoma , producir ARNm y entregar su ARNm infeccioso al citoplasma de la célula huésped. El núcleo viral de ARN bicatenario , que desempeña un papel importante en el empaquetamiento del genoma y la transcripción del ARNm , así como en la maduración, contiene proteínas que están presentes en todo el grupo de ARN bicatenario de tipo 3. En la capa externa de la cápside, las proteínas participan en el papel de la estabilidad ambiental y la adhesión celular a una serie de huéspedes, que son bastante variables incluso dentro del mismo género de Orthoreovirus . [6]
Se ha reconstruido completamente la estructura interna de MRV. λ3 es la replicasa de ARN, mientras que μ2 es un factor de transcripción. [9]
Como se indicó al principio del artículo, los huéspedes naturales del orthoreovirus de mamíferos son los mamíferos , desde los cerdos hasta los humanos, y la mayoría de las especies de mamíferos. Por eso se describe al orthoreovirus de mamíferos como una presencia ubicua, porque se han encontrado anticuerpos para uno (o más) serotipos en prácticamente todas las especies de mamíferos. Esto se debe, al menos en parte, a que el orthoreovirus de mamíferos no depende de los artrópodos para su transmisión como lo hacen muchos virus. En cambio, el orthoreovirus de mamíferos se transmite por vía oral-fecal , lo que significa que de alguna manera las heces de un huésped infectado son ingeridas por otra persona, o por vía de aerosol , lo que significa que las partículas virales viajan a través del aire y son inhaladas por una persona. [6] [7] [10]
Una vez dentro de la persona, el orthoreovirus de mamíferos se adhiere a las células diana a través de la proteína σ1, un trímero filamentoso que sobresale de la capa externa de la cápside. La molécula de adhesión de unión-A es un receptor para el orthoreovirus de mamíferos independientemente del serotipo. El ácido siálico , residente dentro del sistema respiratorio de la mayoría de los mamíferos, es un correceptor para el orthoreovirus de mamíferos tipo 3 (Dearing). Después de unirse a los receptores en la superficie de la célula huésped, el virus es llevado a la célula a través de endocitosis mediada por receptores . Después de la internalización del virus, la cápside externa viral se desmonta dentro del compartimento endocítico (la vesícula ). Este desmontaje de la cápside externa del virus lo realizan las proteasas endocíticas , durante condiciones de pH ácido. Esto conduce a la eliminación de la proteína σ3, lo que da como resultado la exposición de micro1, un mediador de penetración de membrana, así como un cambio conformacional en la proteína de unión σ1. [7] [10] [11] [12]
Después de que las partículas virales sin recubrimiento penetran en los endosomas , la transcripción temprana del genoma de ARN bicatenario por la polimerasa viral ocurre dentro del núcleo viral sin recubrimiento (desnudo) . Este proceso ocurre de esta manera para que el ARN bicatenario viral no quede expuesto al citoplasma , ya que esto haría que la célula atacara la partícula viral. El núcleo viral luego se libera en el citoplasma de la célula huésped, donde ocurre la replicación. La replicación se completa con la ayuda de la proteína λ3, que actúa como ARN polimerasa dependiente de ARN . Se sintetizan transcripciones de cadena positiva de cada uno de los segmentos de ARN bicatenario , estas transcripciones luego se usan como plantillas para la traducción de proteínas, además de hacer el ARN de cadena negativa . La proteína viral μ2, es un cofactor de la transcriptasa que forma parte de la transcripción , también tiene funciones enzimáticas como tapar el ARNm y actúa como ARN helicasa , que separa las cadenas de ARN bicatenario . [7] [11] [13] [14]
Las transcripciones de ARN mensajero del orthoreovirus de mamíferos tienen una región corta no traducida de 5' y no tienen colas de poli A de 3'; algunas carecen de tapas de 5' durante la postinfección. Se desconoce cómo las versiones sin tapa del ARNm del MRV pueden usar un ribosoma de la célula huésped en la traducción . Las proteínas virales del orthoreovirus de mamíferos y sus ARN genómicos se agregan en fábricas virales del citoplasma. Las cadenas de ARN positivas y negativas se emparejarán para crear el genoma viral de ARN bicatenario . El virión se ensambla en partículas subvirales en el citoplasma . Debido a la naturaleza del orthoreovirus de mamíferos, su cápside externa (T = 13) e interna (T = 2) se autoensamblan. Las proteínas de la cápside interna requieren la coexpresión de la proteína T2 y la proteína σ2, para estabilizar la estructura de la cápside interna y ayudar en el ensamblaje. El ensamblaje de la cápside externa depende de la proteína viral σ3, que ayuda a la formación de la cápside icosaédrica . Una vez que el virus se ha ensamblado y madurado por completo, la partícula de ortoreovirus de mamíferos recién formada se libera de la célula huésped; no se sabe con certeza cómo lo hace la partícula viral, pero se presume que sucede después de la muerte celular y la ruptura de la membrana plasmática del huésped . [7] [11] [13]
El orthoreovirus de mamíferos, como se mencionó anteriormente, es omnipresente entre los mamíferos e infecta una gran variedad de especies en todo el mundo. Los ejemplos incluyen cerdos , ganado , caballos , primates , perros , gatos , conejos , ratones , marsupiales y humanos . La infección experimental del orthoreovirus de mamíferos tipo 3 resultó en una mortalidad del 100% en cerdos neonatos, los cerdos desarrollaron gastroenteritis aguda y diarrea severa dentro de las 72 horas posteriores a la infección. Además, se ha informado que el MRV-3 causa enteritis , neumonía , encefalitis y falla reproductiva en cerdos. La infección experimental del orthoreovirus de mamíferos tipo 1 resultó en neumonía , enteritis , fiebre y diarrea . [15] [16]
)
{{cite book}}: |website=ignorado ( ayuda )