
La obtención de imágenes de células vivas es el estudio de células vivas mediante microscopía de lapso de tiempo . Los científicos la utilizan para obtener una mejor comprensión de la función biológica a través del estudio de la dinámica celular. [1] La obtención de imágenes de células vivas fue pionera en la primera década del siglo XXI. Una de las primeras películas microcinematográficas de células con lapso de tiempo jamás realizadas fue realizada por Julius Ries, que muestra la fertilización y el desarrollo del huevo de erizo de mar . [2] Desde entonces, se han desarrollado varios métodos de microscopía para estudiar las células vivas con mayor detalle y con menos esfuerzo. Se ha utilizado un tipo más nuevo de obtención de imágenes mediante puntos cuánticos , ya que se ha demostrado que son más estables. [3] El desarrollo de la microscopía holotomográfi ca ha hecho caso omiso de la fototoxicidad y otras desventajas derivadas de la tinción al implementar la tinción digital basada en el índice de refracción de las células. [4] [5]
Los sistemas biológicos existen como una interacción compleja de innumerables componentes celulares que interactúan en cuatro dimensiones para producir el fenómeno llamado vida. Si bien es común reducir los organismos vivos a muestras no vivas para adaptarse a las herramientas tradicionales de imágenes estáticas, cuanto más se desvía la muestra de las condiciones nativas, más probable es que los delicados procesos en cuestión presenten perturbaciones. [6] Por lo tanto, la onerosa tarea de capturar la verdadera identidad fisiológica del tejido vivo requiere una visualización de alta resolución tanto en el espacio como en el tiempo dentro del organismo original. [7] Los avances tecnológicos de las imágenes de células vivas, diseñadas para proporcionar imágenes espaciotemporales de eventos subcelulares en tiempo real, cumplen una función importante para corroborar la relevancia biológica de los cambios fisiológicos observados durante la experimentación. Debido a su relación contigua con las condiciones fisiológicas, los ensayos de células vivas se consideran el estándar para investigar eventos celulares complejos y dinámicos. [8] A medida que los procesos dinámicos como la migración , el desarrollo celular y el tráfico intracelular se convierten cada vez más en el foco de la investigación biológica, las técnicas capaces de capturar datos tridimensionales en tiempo real para redes celulares ( in situ ) y organismos completos ( in vivo ) se convertirán en herramientas indispensables para comprender los sistemas biológicos. La aceptación general de las imágenes de células vivas ha llevado a una rápida expansión en el número de profesionales y ha establecido la necesidad de una mayor resolución espacial y temporal sin comprometer la salud de la célula. [9]
Antes de la introducción del microscopio de contraste de fases , era difícil observar células vivas. Como las células vivas son translúcidas, deben teñirse para ser visibles en un microscopio óptico tradicional . Desafortunadamente, el proceso de tinción de las células generalmente las mata. Con la invención de la microscopía de contraste de fases se hizo posible observar células vivas no teñidas en detalle. Después de su introducción en la década de 1940, la obtención de imágenes de células vivas rápidamente se hizo popular mediante la microscopía de contraste de fases. [11] El microscopio de contraste de fases se popularizó a través de una serie de películas de lapso de tiempo (ver video), grabadas con una cámara de película fotográfica. [12] Su inventor, Frits Zernike , fue galardonado con el Premio Nobel en 1953. [13] Otras técnicas de contraste de fases posteriores utilizadas para observar células no teñidas son la modulación de Hoffman y la microscopía de contraste de interferencia diferencial .
La microscopía de contraste de fases no tiene la capacidad de observar proteínas específicas u otros compuestos químicos orgánicos que forman la compleja maquinaria de una célula. Por lo tanto, se han desarrollado tinciones fluorescentes sintéticas y orgánicas para marcar dichos compuestos, haciéndolos observables mediante microscopía de fluorescencia (ver video). [15] Sin embargo, las tinciones fluorescentes son fototóxicas , invasivas y blanquean cuando se observan. Esto limita su uso cuando se observan células vivas durante períodos prolongados de tiempo. Por lo tanto, las técnicas de contraste de fases no invasivas se utilizan a menudo como un complemento vital para la microscopía de fluorescencia en aplicaciones de imágenes de células vivas. [16] [17] Sin embargo, los métodos de microscopía de fluorescencia asistida por aprendizaje profundo ayudan a reducir la carga de luz y la fototoxicidad y permiten incluso la obtención repetida de imágenes en vivo de alta resolución. [18]
Como resultado del rápido aumento de la densidad de píxeles de los sensores de imágenes digitales , la microscopía cuantitativa de contraste de fases ha surgido como un método de microscopía alternativo para la obtención de imágenes de células vivas. [20] [21] La microscopía cuantitativa de contraste de fases tiene una ventaja sobre la microscopía fluorescente y de contraste de fases en que es no invasiva y cuantitativa por naturaleza.
Debido a la estrecha profundidad focal de la microscopía convencional, la obtención de imágenes de células vivas se limita en gran medida a la observación de células en un único plano. La mayoría de las implementaciones de la microscopía cuantitativa de contraste de fases permiten crear y enfocar imágenes en diferentes planos focales a partir de una única exposición. Esto abre la posibilidad futura de obtener imágenes tridimensionales de células vivas mediante técnicas de fluorescencia. [22] La microscopía cuantitativa de contraste de fases con barrido rotacional permite obtener imágenes tridimensionales de células vivas con lapso de tiempo y alta resolución. [23] [24] [4]
La holotomografía (HT) es una técnica láser para medir el índice de refracción tridimensional (IR) de una muestra microscópica, como células y tejidos biológicos. Debido a que el IR puede servir como un contraste de imagen intrínseco para objetos transparentes o en fase, las mediciones de los tomogramas de IR pueden proporcionar imágenes cuantitativas sin etiquetas de objetos de fase microscópica. Para medir el tomograma de IR 3D de las muestras, la HT emplea el principio de imágenes holográficas y dispersión inversa . Por lo general, se miden múltiples imágenes holográficas 2D de una muestra en varios ángulos de iluminación, empleando el principio de imágenes interferométricas. Luego, se reconstruye un tomograma de IR 3D de la muestra a partir de estas múltiples imágenes holográficas 2D resolviendo inversamente la dispersión de luz en la muestra.
El principio de la tomografía computarizada con rayos X (TC) es muy similar al de la tomografía computarizada (TC ) . La TC mide múltiples imágenes de rayos X en 2D de un cuerpo humano en varios ángulos de iluminación y luego se recupera un tomograma en 3D (absortividad de rayos X) utilizando la teoría de dispersión inversa. Tanto la TC con rayos X como la TC con láser comparten la misma ecuación rectora: la ecuación de Helmholtz , la ecuación de onda para una longitud de onda monocromática. La TC también se conoce como tomografía por difracción óptica.
La combinación de holografía y escaneo rotacional permite registros de células vivas a largo plazo y sin etiquetas.
La nanoscopia óptica no invasiva puede lograr una resolución lateral de este tipo mediante un esquema de detección cuasi- 2π -holográfico y una deconvolución compleja. Las frecuencias espaciales de la célula captada por la imagen no tienen sentido para el ojo humano, pero estas frecuencias dispersas se convierten en un holograma y sintetizan un paso de banda, que tiene una resolución que duplica la disponible normalmente. Los hologramas se registran desde diferentes direcciones de iluminación en el plano de la muestra y se observan variaciones tomográficas de sublongitud de onda de la muestra. Las aperturas a escala nanométrica sirven para calibrar la reconstrucción tomográfica y caracterizar el sistema de obtención de imágenes mediante la función de transferencia coherente, lo que da lugar a un filtrado inverso realista y garantiza una reconstrucción de campo verdaderamente compleja. [24]
En conclusión, las 2 terminologías de (i) resolución óptica (la real) y (ii) resolución de muestreo (la de la pantalla) se separan para la microscopía holotomográfica 3D.
La obtención de imágenes de células vivas representa un compromiso cuidadoso entre la adquisición de la imagen de mayor resolución y el mantenimiento de las células vivas durante el mayor tiempo posible. [25] Como resultado, los microscopistas de células vivas se enfrentan a un conjunto único de desafíos que a menudo se pasan por alto cuando se trabaja con muestras fijas. Además, la obtención de imágenes de células vivas a menudo emplea especificaciones especiales de sistemas ópticos y detectores. Por ejemplo, idealmente, los microscopios utilizados en la obtención de imágenes de células vivas tendrían altas relaciones señal-ruido , velocidades rápidas de adquisición de imágenes para capturar videos de lapso de tiempo de eventos extracelulares y mantener la viabilidad a largo plazo de las células. [26] Sin embargo, optimizar incluso una sola faceta de la adquisición de imágenes puede requerir muchos recursos y debe considerarse caso por caso.
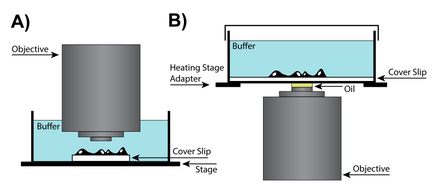
En los casos en los que se requiere espacio adicional entre el objetivo y la muestra para trabajar con ella, se puede utilizar una lente seca, lo que puede requerir ajustes adicionales del collar de corrección, que cambia la ubicación de la lente en el objetivo, para tener en cuenta las diferencias en las cámaras de imágenes. Las lentes de objetivo especiales están diseñadas con collares de corrección que corrigen las aberraciones esféricas al tiempo que tienen en cuenta el grosor del cubreobjetos. En las lentes de objetivo secas de alta apertura numérica (NA), el anillo de ajuste del collar de corrección cambia la posición de un grupo de lentes móviles para tener en cuenta las diferencias en la forma en que el exterior de la lente enfoca la luz en relación con el centro. Aunque las aberraciones de la lente son inherentes a todos los diseños de lentes, se vuelven más problemáticas en las lentes secas, donde la retención de la resolución es clave. [27]
La inmersión en aceite es una técnica que puede aumentar la resolución de la imagen sumergiendo la lente y la muestra en aceite con un alto índice de refracción . Dado que la luz se desvía cuando pasa entre medios con diferentes índices de refracción, al colocar aceite con el mismo índice de refracción que el vidrio entre la lente y el portaobjetos, se pueden evitar dos transiciones entre índices de refracción. [28] Sin embargo, para la mayoría de las aplicaciones se recomienda que se utilice la inmersión en aceite con muestras fijas (muertas) porque las células vivas requieren un entorno acuoso y la mezcla de aceite y agua puede causar aberraciones esféricas graves. Para algunas aplicaciones, se puede utilizar aceite de silicona para producir reconstrucciones de imágenes más precisas. El aceite de silicona es un medio atractivo porque tiene un índice de refracción cercano al de las células vivas, lo que permite producir imágenes de alta resolución al tiempo que minimiza las aberraciones esféricas. [27]
La obtención de imágenes de células vivas requiere una muestra en un entorno acuoso que suele estar a una distancia de entre 50 y 200 micrómetros del cubreobjetos. Por lo tanto, las lentes de inmersión en agua pueden ayudar a lograr un mayor poder de resolución debido a que tanto el entorno como las propias células estarán cerca del índice de refracción del agua. Las lentes de inmersión en agua están diseñadas para ser compatibles con el índice de refracción del agua y, por lo general, tienen un collar correctivo que permite ajustar el objetivo. Además, debido al mayor índice de refracción del agua, las lentes de inmersión en agua tienen una alta apertura numérica y pueden producir imágenes superiores a las lentes de inmersión en aceite cuando se resuelven planos con una profundidad superior a 0 μm. [27]
Otra solución para la obtención de imágenes de células vivas es la lente de inmersión. Estas lentes son un subconjunto de las lentes de inmersión en agua que no requieren un cubreobjetos y se pueden sumergir directamente en el entorno acuoso de la muestra. Una de las principales ventajas de la lente de inmersión es que tiene una distancia de trabajo efectiva larga. [29] Como no se requiere un cubreobjetos, este tipo de lente puede acercarse a la superficie de la muestra y, como resultado, la resolución está limitada por las restricciones impuestas por la aberración esférica en lugar de las limitaciones físicas del cubreobjetos. Aunque las lentes de inmersión pueden ser muy útiles, no son ideales para todos los experimentos, ya que el acto de "sumergir" la lente puede alterar las células de la muestra. Además, como la cámara de incubación debe estar abierta a la lente, los cambios en el entorno de la muestra debido a la evaporación deben controlarse de cerca. [27]
En la actualidad, la mayoría de las técnicas de obtención de imágenes en vivo se basan en regímenes de alta iluminación o en el marcado fluorescente, lo que induce fototoxicidad y compromete la capacidad de mantener las células vivas e inalteradas a lo largo del tiempo. Dado que nuestro conocimiento de la biología se basa en la observación, es fundamental minimizar las perturbaciones inducidas por la técnica de obtención de imágenes.
El auge de la microscopía confocal está estrechamente relacionado con la accesibilidad de los láseres de alta potencia, que pueden alcanzar altas intensidades de excitación de luz. Sin embargo, la salida de alta potencia puede dañar los fluoróforos sensibles , por lo que los láseres suelen funcionar significativamente por debajo de su potencia de salida total. [30] La sobreexposición a la luz puede provocar fotodaño debido al fotoblanqueo o fototoxicidad . Los efectos del fotoblanqueo pueden reducir significativamente la calidad de las imágenes fluorescentes y, en los últimos años, ha habido una demanda significativa de fluoróforos comerciales de mayor duración. Una solución, la serie Alexa Fluor , muestra poco o ningún desvanecimiento incluso a altas intensidades de láser. [31]
En condiciones fisiológicas, muchas células y tipos de tejidos están expuestos a niveles bajos de luz. [32] Como resultado, es importante minimizar la exposición de células vivas a altas dosis de longitudes de onda de luz ultravioleta (UV), infrarrojas (IR) o excitadoras de fluorescencia, que pueden dañar el ADN , aumentar las temperaturas celulares y causar fotoblanqueo respectivamente. [33] Los fotones de alta energía absorbidos por los fluoróforos y la muestra se emiten en longitudes de onda más largas proporcionales al desplazamiento de Stokes . [34] Sin embargo, los orgánulos celulares pueden dañarse cuando la energía del fotón produce cambios químicos y moleculares en lugar de ser reemitida. [35] Se cree que el principal culpable de la toxicidad inducida por la luz que experimentan las células vivas es el resultado de los radicales libres producidos por la excitación de moléculas fluorescentes. [32] Estos radicales libres son altamente reactivos y causan la destrucción de componentes celulares, lo que puede resultar en un comportamiento no fisiológico.
Un método para minimizar el daño solar consiste en reducir la concentración de oxígeno en la muestra para evitar la formación de especies reactivas de oxígeno . [36] Sin embargo, este método no siempre es posible en la obtención de imágenes de células vivas y puede requerir una intervención adicional. Otro método para reducir los efectos de los radicales libres en la muestra es el uso de reactivos antidesvanecimiento. Desafortunadamente, la mayoría de los reactivos antidesvanecimiento comerciales no se pueden utilizar en la obtención de imágenes de células vivas debido a su toxicidad. [37] En cambio, se pueden utilizar eliminadores de radicales libres naturales, como la vitamina C o la vitamina E, sin alterar sustancialmente el comportamiento fisiológico en escalas de tiempo más cortas. [38] Recientemente se ha desarrollado y comercializado la obtención de imágenes de células vivas sin fototoxicidad. La microscopía holotomográfica evita la fototoxicidad gracias a su láser de baja potencia (láser clase 1: 0,2 mW/mm2 ) . [4] [5] [39]