La antropología molecular , también conocida como antropología genética , es el estudio de cómo la biología molecular ha contribuido a la comprensión de la evolución humana. [1] Este campo de la antropología examina los vínculos evolutivos entre las poblaciones humanas antiguas y modernas, así como entre las especies contemporáneas. Generalmente, las comparaciones se realizan entre secuencias, ya sean secuencias de ADN o de proteínas ; sin embargo, los primeros estudios utilizaron serología comparativa .
Al examinar las secuencias de ADN en diferentes poblaciones, los científicos pueden determinar la proximidad de las relaciones entre poblaciones (o dentro de las poblaciones). Ciertas similitudes en la composición genética permiten a los antropólogos moleculares determinar si diferentes grupos de personas pertenecen o no al mismo haplogrupo y, por lo tanto, si comparten un origen geográfico común . Esto es importante porque permite a los antropólogos rastrear patrones de migración y asentamiento , lo que brinda información útil sobre cómo se han formado y progresado las poblaciones contemporáneas a lo largo del tiempo. [2]
La antropología molecular ha sido extremadamente útil para establecer el árbol evolutivo de los humanos y otros primates , incluidas especies estrechamente relacionadas como los chimpancés y los gorilas. Si bien es evidente que existen muchas similitudes morfológicas entre los humanos y los chimpancés, por ejemplo, ciertos estudios también han concluido que existe aproximadamente un 98 por ciento de similitud entre el ADN de ambas especies. [ cita requerida ] Sin embargo, estudios más recientes han modificado la similitud del 98 por ciento a una similitud del 94 por ciento, lo que demuestra que la brecha genética entre los humanos y los chimpancés es mayor de lo que se pensaba originalmente. [3] Esta información es útil para buscar ancestros comunes y llegar a una mejor comprensión de cómo evolucionaron los humanos.
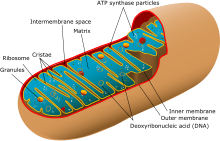
En los seres humanos existen dos grupos de ligamiento continuo que son transmitidos por un solo sexo. El primero es el cromosoma Y , que se transmite de padre a hijo. Las hembras anatómicas portan un cromosoma Y sólo en raras ocasiones, como resultado de un defecto genético. El otro grupo de ligamiento es el ADN mitocondrial (ADNmt). El ADNmt casi siempre sólo se transmite a la siguiente generación por las hembras, pero en circunstancias muy excepcionales el ADNmt puede transmitirse a través de los machos. [ aclaración necesaria ] La porción no recombinante del cromosoma Y y el ADNmt, en circunstancias normales, no experimentan una recombinación productiva. Parte del cromosoma Y puede experimentar una recombinación con el cromosoma X y, dentro de la historia de los simios , el límite ha cambiado. Tales cambios recombinantes en la región no recombinante del Y son extremadamente raros. [ cita requerida ]

El ADN mitocondrial se convirtió en un área de investigación en filogenética a fines de la década de 1970. A diferencia del ADN genómico, ofrecía ventajas en el sentido de que no sufría recombinación. El proceso de recombinación, si es lo suficientemente frecuente, corrompe la capacidad de crear árboles parsimoniosos debido a tramos de sustituciones de aminoácidos (SNP). [ aclaración necesaria ] Cuando se busca entre especies distantemente relacionadas, la recombinación es un problema menor ya que la recombinación entre ramas de ancestros comunes se previene después de que ocurre la especiación verdadera. Cuando se examinan especies estrechamente relacionadas, o la ramificación dentro de las especies, la recombinación crea una gran cantidad de "SNP irrelevantes" para el análisis cladístico. El ADNmt, a través del proceso de división de orgánulos, se volvió clonal con el tiempo; muy poco, o a menudo nada, de ese ADNmt paterno se transmite. Si bien la recombinación puede ocurrir en el ADNmt, existe poco riesgo de que se transmita a la siguiente generación. Como resultado, los ADNmt se convierten en copias clonales entre sí, excepto cuando surge una nueva mutación. Como resultado, el ADNmt no tiene los inconvenientes de los loci autosómicos cuando se estudia en grupos de cruzamiento. Otra ventaja del ADNmt es que las regiones hipervariables evolucionan muy rápidamente; esto demuestra que ciertas regiones del ADN mitocondrial se acercan a la neutralidad. Esto permitió el uso del ADN mitocondrial para determinar que la edad relativa de la población humana era pequeña, habiendo pasado por una constricción reciente hace unos 150.000 años (ver #Causas de errores).
El ADN mitocondrial también se ha utilizado para verificar la proximidad de los chimpancés a los humanos en relación con los gorilas , y para verificar la relación de estas tres especies con los orangutanes .
Más recientemente, [ ¿ cuándo? ] el genoma del ADNmt se ha utilizado para estimar patrones de ramificación en pueblos de todo el mundo, como cuándo se estableció el nuevo mundo y cómo. El problema con estos estudios ha sido que dependen en gran medida de las mutaciones en la región codificante. Los investigadores han descubierto cada vez más que a medida que los humanos se mudaron de las regiones sudorientales de África, se acumularon más mutaciones en la región codificante de lo esperado, y se cree que en su paso al nuevo mundo algunos grupos [ cita requerida ] pasaron de los trópicos asiáticos a Siberia a una antigua región terrestre llamada Beringia y migraron rápidamente a Sudamérica. Muchos de los ADNmt tienen muchas más mutaciones y en sitios de codificación raramente mutados en relación con las expectativas de mutaciones neutrales.
El ADN mitocondrial ofrece otra ventaja sobre el ADN autosómico. Generalmente hay de 2 a 4 copias de cada cromosoma en cada célula (1 a 2 de cada cromosoma parental). En el caso del ADN mitocondrial, puede haber de docenas a cientos en cada célula. Esto aumenta la cantidad de cada loci de ADN mitocondrial en al menos una magnitud. En el caso del ADN antiguo, en el que el ADN está muy degradado, la cantidad de copias de ADN es útil para extender y unir fragmentos cortos, y disminuye la cantidad de hueso extraído de restos fósiles/antiguos de gran valor. A diferencia del cromosoma Y, tanto los restos masculinos como los femeninos contienen ADN mitocondrial en cantidades aproximadamente iguales.


El cromosoma Y se encuentra en el núcleo de las células normales ( ADN nuclear ). A diferencia del ADNmt, tiene mutaciones en la porción no recombinante (NRY) del cromosoma muy espaciadas, tan separadas que encontrar las mutaciones en los nuevos cromosomas Y es un trabajo intensivo en comparación con el ADNmt. Muchos estudios se basan en repeticiones en tándem; sin embargo, las repeticiones en tándem pueden expandirse y retraerse rápidamente y en algunos patrones predecibles. El cromosoma Y solo rastrea las líneas masculinas y no se encuentra en las mujeres, mientras que el ADNmt se puede rastrear en los hombres aunque no transmitan el ADNmt. Además, se ha estimado que las poblaciones masculinas efectivas en el período prehistórico eran típicamente dos hembras por macho, y estudios recientes muestran que la hegemonía cultural juega un papel importante en el paso del Y. Esto ha creado discordancia entre hombres y mujeres para el Tiempo hasta el Ancestro Común Más Reciente (TMRCA). Las estimaciones para el TMRCA del Y varían de 1/4 a menos de la mitad del TMRCA del ADNmt. No está claro si esto se debe a las altas proporciones de machos a hembras en el pasado, junto con las repetidas migraciones desde África, como resultado de un cambio en la tasa de mutación, o como algunos han propuesto, que las hembras de la LCA entre chimpancés y humanos continuaron transmitiendo ADN millones después de que los machos dejaron de hacerlo. En la actualidad, la mejor evidencia sugiere que en la migración, la proporción de machos a hembras en los humanos puede haber disminuido, lo que provocó un recorte de la diversidad Y en múltiples ocasiones dentro y fuera de África.

Para la filogenética molecular de corto alcance y el cronometraje molecular, el cromosoma Y es muy eficaz y crea una segunda perspectiva. Un argumento que surgió fue que los maoríes , según el ADNmt, parecen haber migrado desde el este de China o Taiwán, y, según el cromosoma Y, desde la región de Papúa Nueva Guinea. Cuando se utilizaron los haplotipos HLA para evaluar las dos hipótesis, se descubrió que ambas eran correctas: los maoríes eran una población mestiza. Dichas mezclas parecen ser comunes en la población humana y, por lo tanto, el uso de un solo loci haploide puede brindar una perspectiva sesgada.
El cromosoma X también es una forma de ADN nuclear. Dado que se encuentra como una copia en los machos y dos cromosomas no idénticos en las hembras, tiene una ploidía de 1,5. Sin embargo, en los seres humanos la ploidía efectiva es algo mayor, ~1,7, ya que las hembras en la población reproductora han tendido a superar en número a los machos en una proporción de 2:1 durante una gran parte de la prehistoria humana. Al igual que el ADNmt, el ADN ligado al cromosoma X tiende a enfatizar mucho más la historia de la población femenina que la masculina. Se han realizado varios estudios de loci en el cromosoma X, en total se han examinado 20 sitios. Estos incluyen PDHA1, PDHA1, Xq21.3, Xq13.3, Zfx, Fix, Il2rg, Plp, Gk, Ids, Alas2, Rrm2p4, AmeIX, Tnfsf5, Licam y Msn. El tiempo hasta el ancestro común más reciente (TMRCA) varía desde fijo hasta ~1,8 millones de años, con una mediana de alrededor de 700.000 años. Estos estudios se corresponden aproximadamente con la distribución de fijación esperada de los alelos, dado el desequilibrio de ligamiento entre sitios adyacentes. Para algunos alelos, el punto de origen es difícil de alcanzar, para otros, el punto de origen apunta hacia el África subsahariana. Hay algunas distinciones dentro del SSA que sugieren una región más pequeña, pero no hay un tamaño de muestra y una cobertura suficientes para definir un lugar del ancestro común más reciente. El TMRCA es coherente con el cuello de botella implícito en el ADNmt y lo extiende, con confianza, a unos 500.000 años.
Se ha secuenciado el ADN mitocondrial neandertal de Krings y la similitud de secuencias indica un origen igualmente reciente a partir de una pequeña población de la rama neandertal de los homínidos tardíos . También se ha secuenciado el gen MCR1, pero los resultados son controvertidos; un estudio afirma que los problemas de contaminación no se pueden resolver a partir de las similitudes con los neandertales humanos. Sin embargo, lo más importante es que no se ha obtenido ninguna secuencia de ADN del Homo erectus , el Homo floresiensis ni ninguno de los otros homínidos tardíos. Algunas de las secuencias antiguas obtenidas tienen errores muy probables y se han realizado controles adecuados para evitar la contaminación.

La filogenética molecular se basa en la cuantificación de sustituciones y luego en la comparación de secuencias con otras especies. Hay varios puntos en el proceso que generan errores. El primer y mayor desafío es encontrar "anclas" que permitan a la investigación calibrar el sistema. En este ejemplo, hay 10 mutaciones entre chimpancés y humanos, pero el investigador no tiene fósiles conocidos que sean ancestrales de ambos pero no ancestrales de la siguiente especie en el árbol, el gorila . Sin embargo, hay fósiles que se cree que son ancestrales de orangutanes y humanos, de hace unos 14 millones de años. Para que el investigador pueda usar la comparación entre el orangután y el humano y obtenga una diferencia de 24. Usando esto puede estimar (24/(14*2, el "2" es para la longitud de la rama al humano (14ma) y la rama al orangután (14ma) desde su último ancestro común (LCA). La tasa de mutación en 0.857 para un tramo de secuencia. Las tasas de mutación se dan, sin embargo, como tasa por sitio de nucleótido (nt), por lo que si la secuencia tuviera, digamos, 100 nt de longitud, esa tasa sería 0.00857/nt por millón de años. Diez mutaciones*100nt/(0.00857*2) = 5.8 millones de años.
Existen varios problemas que no se ven en lo anterior. Primero, las mutaciones ocurren como eventos aleatorios. Segundo, la probabilidad de que cualquier sitio en el genoma varíe es diferente del siguiente sitio, un muy buen ejemplo son los codones para aminoácidos, los primeros dos nt en un codón pueden mutar a 1 cada mil millones de años, pero el tercer nt puede mutar 1 cada millón de años. A menos que los científicos estudien la secuencia de una gran cantidad de animales, particularmente aquellos cercanos a la rama que se está examinando, generalmente no saben cuál es la tasa de mutación para un sitio dado. Las mutaciones ocurren en las posiciones 1 y 2 de los codones, pero en la mayoría de los casos estas mutaciones están bajo selección negativa y, por lo tanto, se eliminan de la población en pequeños períodos de tiempo. Al definir la tasa de evolución en el ancla, uno tiene el problema que crea la mutación aleatoria. Por ejemplo, una tasa de .005 o .010 también puede explicar 24 mutaciones de acuerdo con la distribución de probabilidad binomial . Algunas de las mutaciones que ocurrieron entre los dos se han revertido, ocultando una tasa inicialmente más alta. La selección puede influir en esto, una mutación rara puede ser selectiva en un punto X en el tiempo, pero más tarde el clima puede cambiar o la especie migra y ya no es selectiva, y se ejerce presión sobre nuevas mutaciones que reviertan el cambio, y a veces puede ocurrir la reversión de un nt, cuanto mayor sea la distancia entre dos especies, más probable es que esto ocurra. Además, a partir de esa especie ancestral, ambas especies pueden mutar aleatoriamente un sitio al mismo nucleótido. Muchas veces esto se puede resolver obteniendo muestras de ADN de especies en las ramas, creando un árbol parsimonioso en el que se puede deducir el orden de mutación, creando un diagrama de longitud de rama. Este diagrama producirá entonces una estimación más precisa de las mutaciones entre dos especies. Estadísticamente, se puede asignar varianza en función del problema de aleatoriedad, mutaciones retroactivas y mutaciones paralelas (homoplasias) para crear un rango de error.
Sin embargo, existe otro problema en la calibración que ha desafiado el análisis estadístico. Existe una designación de verdadero/falso de un fósil a un ancestro menos común. En realidad, las probabilidades de tener el ancestro menos común de dos especies existentes como ancla son bajas; a menudo, ese fósil ya se encuentra en una rama (subestimando la edad), se encuentra en una tercera rama (subestimando la edad) o, en el caso de estar dentro de la especie LCA, puede haber sido millones de años más antiguo que la rama. Hasta la fecha, la única forma de evaluar esta variación es aplicar la filogenética molecular a las especies que se afirma que son puntos de ramificación. Sin embargo, esto solo identifica los puntos de anclaje "externos". Y dado que es más probable que los fósiles más abundantes sean más jóvenes que el punto de ramificación, el fósil externo puede ser simplemente un representante raro y más antiguo. Estas incógnitas crean una incertidumbre que es difícil de cuantificar y, a menudo, no se intenta.
En trabajos recientes se ha podido estimar, de forma aproximada, la varianza. La tendencia general, a medida que se descubren nuevos fósiles, es que los fósiles más antiguos subestiman la edad del punto de ramificación. Además de esto, la datación de fósiles ha tenido un historial de errores y ha habido muchas dataciones revisadas. La edad asignada por los investigadores a algunos puntos de ramificación importantes casi se ha duplicado en los últimos 30 años. Un excelente ejemplo de esto es el debate sobre LM3 (lago Mungo 3) en Australia. Originalmente se fechó en alrededor de 30 mil mediante datación por carbono, pero la datación por carbono tiene problemas, sin embargo, para muestras de más de 20 mil de edad, y graves problemas para muestras de alrededor de 30 mil de edad. Otro estudio examinó el fósil y estimó que la edad era de 62 mil.
En el momento en que se tiene una estimación de la tasa de mutación, dado lo anterior, deben existir dos fuentes de varianza que deben multiplicarse de forma cruzada para generar una varianza general. Esto se hace con poca frecuencia en la literatura.
El tiempo hasta el ancestro común más reciente ( TMRCA ) combina los errores en la calibración con los errores en la determinación de la edad de una rama local.

Con el recién descubierto ADN como material genético, a principios de la década de 1960 la secuenciación de proteínas estaba empezando a despegar. [4] La secuenciación de proteínas comenzó en el citocromo C y la hemoglobina. Gerhard Braunitzer secuenció la hemoglobina y la mioglobina , en total se realizaron más de cientos de secuencias de especies de amplia distribución. En 1967, AC Wilson comenzó a promover la idea de un "reloj molecular". En 1969, el reloj molecular se aplicó a la evolución antropoide y V. Sarich y AC Wilson descubrieron que la albúmina y la hemoglobina tienen tasas comparables de evolución, lo que indica que los chimpancés y los humanos se separaron hace unos 4 a 5 millones de años. [5] En 1970, Louis Leakey confrontó esta conclusión argumentando a favor de una calibración incorrecta de los relojes moleculares. [6] En 1975, la secuenciación de proteínas y la serología comparativa combinadas se utilizaron para proponer que el pariente vivo más cercano de los humanos (como especie ) era el chimpancé. [7] En retrospectiva, el último ancestro común (LCA) de los humanos y los chimpancés parece ser más antiguo que la estimación de Sarich y Wilson , pero tampoco tan antiguo como Leakey afirmó. Sin embargo, Leakey estaba en lo cierto en la divergencia de los monos del viejo y el nuevo mundo, el valor que usaron Sarich y Wilson fue una subestimación significativa. Este error en la capacidad de predicción resalta un tema común. (Véase Causas de error)

En 1979, W. M. Brown y Wilson comenzaron a estudiar la evolución del ADN mitocondrial en animales y descubrieron que evolucionaban rápidamente. [8] La técnica que utilizaron fue el polimorfismo de longitud de fragmentos de restricción ( RFLP ), que era más asequible en ese momento en comparación con la secuenciación. En 1980, W. M. Brown, al observar la variación relativa entre humanos y otras especies, reconoció que había una constricción reciente (hace 180.000 años) en la población humana. [9] Un año después, Brown y Wilson estaban analizando fragmentos RFLP y determinaron que la población humana se expandió más recientemente que otras poblaciones de simios. [10] En 1984 se realizó la primera secuencia de ADN de un animal extinto. [11] Sibley y Ahlquist aplican la tecnología de hibridación ADN-ADN a la filogenia antropoide y ven que la división pan/humano es más cercana que la división gorila/pan o gorila/humano, una afirmación muy controvertida. [12] [13] Sin embargo, en 1987 pudieron respaldar su afirmación. [14] En 1987, Cann, Stoneking y Wilson sugieren, mediante análisis RFLP del ADN mitocondrial humano, que los humanos evolucionaron a partir de un constricto en África de una sola hembra en una pequeña población, ~10.000 individuos, hace 200.000 años. [15]

En 1987, la amplificación por PCR del ADNmt se utilizó por primera vez para determinar secuencias. [16] En 1991, Vigilante et al. publicaron el trabajo seminal sobre la filogenia del ADNmt que implicaba al África subsahariana como el lugar de los ancestros comunes más recientes de los humanos para todos los ADNmt. [17] La guerra entre el fuera de África y el multirregionalismo, que ya estaba hirviendo con las críticas de Allan Templeton, pronto se intensificó con la participación de paleoantropólogos como Milford Wolpoff. [18] [19] [20] En 1995, F. Ayala publicó su artículo crítico en Science "El mito sobre Eva", que se basó en la secuencia HLA-DR . [21] Sin embargo, en ese momento Ayala no estaba al tanto de la rápida evolución de los loci HLA a través del proceso recombinatorio. En 1996, Parham y Ohta publicaron sus hallazgos sobre la rápida evolución del HLA por recombinación de corta distancia ('conversión génica' o 'recombinación abortiva'), lo que debilitó la afirmación de Ayala (Parham había escrito una revisión un año antes, pero esto había pasado desapercibido). [22] [23] Una serie de artículos seguirían de ambos lados, muchos con métodos y muestreos altamente defectuosos. Uno de los más interesantes [ ¿según quién? ] fue Harris y Hey, 1998, que mostró que el TMCRA (tiempo hasta el ancestro común más reciente) para el gen PDHA1 era muy superior a 1 millón de años. Dada una ploidía en este locus de 1,5 (3 veces mayor que el ADNmt), el TMRCA era más del doble de lo esperado. Si bien esto cae dentro de la "curva de fijación" de 1,5 ploidía (con un promedio de 2 mujeres y 1 hombre), la edad sugerida de 1,8 millones de años está cerca de ser un valor p significativamente desviado para el tamaño de la población, lo que posiblemente indica que la población humana se redujo o se separó de otra población. [24] Curiosamente, el siguiente loci ligado al cromosoma X que examinaron, el Factor IX, mostró una TMRCA de menos de 300.000 años. [25]

La secuenciación de ADN antiguo se había llevado a cabo a una escala limitada hasta finales de la década de 1990, cuando el personal del Instituto Max Planck sorprendió al mundo de la antropología al secuenciar el ADN de un neandertal de unos 40.000 años de antigüedad . [26] [27] [28] El resultado de ese experimento es que las diferencias entre los humanos que vivían en Europa, muchos de los cuales se derivaron del haplogrupo H (CRS), los neandertales se ramificaron a partir de los humanos más de 300.000 años antes de que el haplogrupo H llegara a Europa. Si bien el ADNmt y otros estudios continuaron apoyando un origen africano reciente único, este nuevo estudio básicamente respondió a las críticas del lado neandertal.
Se han logrado avances significativos en la secuenciación genómica desde que Ingman y sus colegas publicaron sus hallazgos sobre el genoma mitocondrial. [29] Se han publicado varios artículos sobre el ADNmt genómico; existe una variabilidad considerable en la tasa de evolución, y la variación de la tasa y la selección son evidentes en muchos sitios. En 2007, Gonder et al. propusieron que una población central de humanos, con el mayor nivel de diversidad y el menor nivel de selección, alguna vez vivió en la región de Tanzania y partes cercanas del sur de África; desde que los humanos abandonaron esta parte de África, las mitocondrias han estado evolucionando selectivamente hacia nuevas regiones. [30]
Crítico en la historia de la antropología molecular:
Loci que se han utilizado en filogenética molecular:
Loci ligados al cromosoma X: PDHA1, Xq21.3, Xq13.3, Zfx, Fix, Il2rg, Plp, Gk, Ids, Alas2, Rrm2p4, AmeIX, Tnfsf5, Licam y Msn
Autosómico: Numerosos.