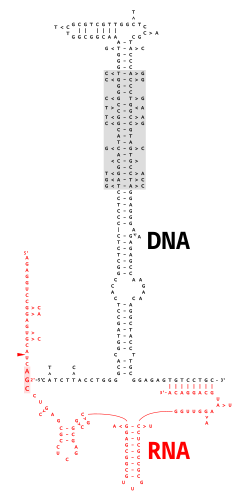
El ADN monocatenario multicopia (ADNms) es un tipo de ADN satélite extracromosómico que consta de una molécula de ADN monocatenario unida covalentemente mediante un enlace fosfodiéster 2'-5' a una guanosina interna de una molécula de ARN . La quimera de ADN/ARN resultante posee dos bucles unidos por una rama similar a las ramas que se encuentran en los intermediarios de empalme de ARN . La región codificante del ADNms, llamada " retrón ", también codifica un tipo de transcriptasa inversa , que es esencial para la síntesis de ADNms. [2]
Antes del descubrimiento del ADNms en las mixobacterias , [3] [4] un grupo de enjambres de bacterias que habitan en el suelo , se pensaba que las enzimas conocidas como transcriptasas inversas (RT) existían sólo en eucariotas y virus . El descubrimiento provocó un aumento de la investigación en la zona. Como resultado, se ha descubierto que el ADNms está ampliamente distribuido entre las bacterias, incluidas varias cepas de Escherichia coli y bacterias patógenas. [5] Investigaciones adicionales descubrieron similitudes entre la transcriptasa inversa codificada por el VIH y un marco de lectura abierto (ORF) que se encuentra en la región codificante del ADNms. Las pruebas confirmaron la presencia de actividad transcriptasa inversa en lisados crudos de cepas que contienen retrones. [6] Aunque se identificó tentativamente un dominio de RNasa H en el ORF de retron, más tarde se descubrió que la actividad de RNasa H requerida para la síntesis de ADNms en realidad la proporciona el huésped. [7]
El descubrimiento del ADNms ha dado lugar a preguntas más amplias sobre dónde se originó la transcriptasa inversa, ya que se han encontrado genes que codifican la transcriptasa inversa (no necesariamente asociados con el ADNms) en procariotas, eucariotas, virus e incluso arqueas . Después de que se descubrió un fragmento de ADN que codifica la producción de ADNms en E. coli , [8] se conjeturó que los bacteriófagos podrían haber sido responsables de la introducción del gen RT en E. coli . [9] Estos descubrimientos sugieren que la transcriptasa inversa jugó un papel en la evolución de los virus a partir de bacterias, con una hipótesis que afirma que, con la ayuda de la transcriptasa inversa, los virus pueden haber surgido como un gen de ADNms separable que adquirió una cubierta proteica. Dado que casi todos los genes RT funcionan en la replicación de retrovirus y/o en el movimiento de elementos transponibles , es razonable imaginar que los retrones podrían ser elementos genéticos móviles, pero ha habido poca evidencia que respalde tal hipótesis, salvo el hecho observado de que el ADNms está ampliamente aunque esporádicamente dispersa entre especies bacterianas de una manera que sugiere una transferencia tanto horizontal como vertical. [5] [10] [11] Dado que no se sabe si las secuencias de retrones per se representan elementos móviles, los retrones se definen funcionalmente por su capacidad para producir ADNms y al mismo tiempo evitan deliberadamente especulaciones sobre otras posibles actividades.
La función del ADNms sigue siendo desconocida a pesar de que hay muchas copias presentes dentro de las células. Las mutaciones knockout que no expresan ADNms son viables, por lo que la producción de ADNms no es esencial para la vida en condiciones de laboratorio. La sobreexpresión de ADNms es mutagénica, aparentemente como resultado de la valoración de las proteínas reparadoras por los pares de bases no coincidentes que son típicos de su estructura. [10] Se ha sugerido que el ADNms puede tener algún papel en la patogenicidad o la adaptación a condiciones estresantes. [12] La comparación de secuencias de ADNms de Myxococcus xanthus , Stigmatella aurantiaca , [1] y muchas otras bacterias [5] [12] revelan dominios conservados e hipervariables que recuerdan a las secuencias conservadas e hipervariables que se encuentran en las moléculas de alorreconocimiento. [13] Los principales ADNms de M. xanthus y S. aurantiaca , por ejemplo, comparten una homología de secuencia del 94 % excepto dentro de un dominio de 19 pares de bases que comparte una homología de secuencia de sólo el 42 %. [1] La presencia de tales dominios es significativa porque las mixobacterias exhiben comportamientos sociales cooperativos complejos que incluyen enjambres y formación de cuerpos fructíferos, mientras que E. coli y otras bacterias patógenas forman biopelículas que exhiben una mayor resistencia a los antibióticos y detergentes. La sostenibilidad de las asambleas sociales que requieren una importante inversión individual de energía depende generalmente de la evolución de los mecanismos de alorreconocimiento que permiten a los grupos distinguir el yo del no-yo. [14]

Se supone que la biosíntesis de ADNms sigue una vía única que no se encuentra en ningún otro lugar de la bioquímica de ADN/ARN. Debido a la similitud de la unión de ramas 2'-5' con las uniones de ramas encontradas en los intermediarios de empalme de ARN, al principio se podría haber esperado que la formación de ramas se realizara mediante ligadura mediada por espliceosoma o ribozima . Sorprendentemente, sin embargo, los experimentos en sistemas libres de células que utilizan retron transcriptasa inversa purificada indican que la síntesis de ADNc se ceba directamente a partir del grupo 2'-OH del residuo G interno específico del ARN cebador. [15] La RT reconoce estructuras de tallo-bucle específicas en el ARN precursor, lo que hace que la síntesis de ADNms por parte de la RT sea muy específica de su propio retrón. [16] La preparación de la síntesis de ADNms ofrece un desafío fascinante para nuestra comprensión de la síntesis de ADN. Las ADN polimerasas (que incluyen la RT) comparten características estructurales altamente conservadas, lo que significa que sus sitios catalíticos activos varían poco de una especie a otra, o incluso entre las ADN polimerasas que usan ADN como plantilla, versus las ADN polimerasas que usan ARN como plantilla. La región catalítica de la transcriptasa inversa eucariota comprende tres dominios denominados "dedos", "palma" y "pulgar" que sujetan la plantilla del cebador bicatenario en un agarre con la mano derecha con el 3'-OH del cebador enterrado en el sitio activo de la polimerasa, [17] un grupo de residuos ácidos y polares altamente conservados situados en la palma entre lo que serían los dedos índice y medio. En los RT eucariotas, el dominio RNasa H se encuentra en la muñeca debajo de la base del pulgar, pero los RT retron carecen de actividad RNasa H. La hendidura de unión del ácido nucleico, que se extiende desde el sitio activo de la polimerasa hasta el sitio activo de la RNasa H, tiene aproximadamente 60 Å de longitud en las RT eucariotas, lo que corresponde a casi dos vueltas helicoidales. [18] Cuando la RT eucariótica extiende un cebador convencional, la doble hélice de ADN/ARN en crecimiento gira en espiral a lo largo de la hendidura y, a medida que la doble hélice pasa el dominio de la RNasa H, el ARN molde se digiere para liberar la cadena naciente de ADNc. Sin embargo, en el caso de la extensión del cebador de ADNms, una larga cadena de ARN permanece unida al 3'-OH de la G cebadora. Aunque es posible modelar un complejo molde RT-cebador que haría que el 2'-OH fuera accesible para Durante la reacción de cebado, [16] una mayor extensión de la cadena de ADN presenta un problema: a medida que avanza la síntesis de ADN, la voluminosa cadena de ARN que se extiende desde el 3'-OH necesita de alguna manera descender en espiral por la hendidura de unión sin ser bloqueada por impedimento estérico . Para superar este problema, la transcriptasa inversa de ADNms claramente requeriría características especiales que no comparten otras RT. [10]