Síntesis de oligonucleótidos
[1] Los productos suelen ser aislados mediante HPLC para obtener los oligonucleótidos deseados en una alta pureza.
Los oligonucleótidos tienen muchas aplicaciones tanto en el campo de biología molecular como en medicina.
[21] Tras la eliminación del grupo acetilo 3’-O usando hidrólisis alcalina, se llevó a cabo una posterior elongación de la cadena.
Siguiendo esta metodología, se sintetizaron conjuntos de tri- y tetradeoxirribonucleótidos, los que fueron convertidos enzimáticamente en oligonucleótidos más largos, lo que permitió la elucidación del código genético.
El método parece ser un paso atrás desde la química más selectiva descrita anteriormente; sin embargo, en aquel entonces, no habían sido introducidos la mayor parte de grupos protectores de fosfato que hay hoy en día.
La falta de una estrategia de protección conveniente necesitaba tomar una retirada a una química más lenta y menos selectiva para lograr el objetivo último del estudio.
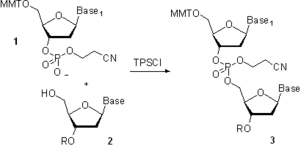
En la década de 1960, grupos dirigidos por R. Letsinger[22] y C. Reese[23] desarrollaron una solución basada en fosfotriéster.
Esto precluía la formación de oligonucleótidos ramificados en el fosfato internucleosídico.
Esto llevó al descubrimiento de la metodología del triéster fosfito.
El grupo liderado por M. Caruthers tuvo la ventaja de tener 1H-tetrasolidofosfitos menos agresivos y más selectivos y por lo tanto implementaron el método en una fase sólida.
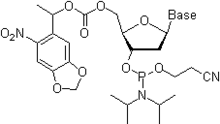
[28] Poco tiempo después, los trabajadores del mismo grupo mejoraron el método usando nucleósidos fosforamiditos más estables como bloques de construcción.
Para poder ser introducidos en una secuencia, un modificador no nucleosídico tiene que tener por lo menos 2 grupos hidroxilos, uno de ellos suele estar protegido por el grupo DMT, mientras que el otro contiene los restos del fosforamidito reactivo.
Los fosforamitos no nucleosídicos son usados para introducir los grupos deseados que no están disponibles en los nucleósidos naturales o que pueden ser introducidos de manera más fácil usando diseños químicos más simples.
Es importante recordar que llevar a cabo una detritilación por un largo periodo de tiempo o con soluciones de ácidos más fuertes de las recomendadas puede llevar a una depurinación del soporte sólido ligado al oligonucleótido y por lo tanto reduce el rendimiento del producto deseado.
[48][70][71][72] La reacción es muy sensible a la presencia de agua, especialmente cuando se usan soluciones diluidas de fosforamiditos, y normalmente se lleva a cabo por acetonitrilo anhídrido.
Generalmente, a gran escala, en la síntesis, el exceso es menor y la concentración de fosforamiditos es mayor.
La oxidación puede llevarse a cabo bajo condiciones anhídridas usando terc-butil hidroperoxido[75] o, más eficiente, (1S)-(+)-(10-camphorsulfonyl)-oxaziridino (CSO).
[80] Para que el material del soporte sólido sea adecuado para la síntesis de oligonucleótidos, los enlazadores no nucleosidicos o los succionatos de nucleosido deben de estar unidos por un enlace covalente a un grupo amino reactivo, como aminopropil CPG, LCAA CPG o aminometil MPPS.
El resto de los grupos aminos que no reaccionaron son cubiertos con anhídrido acético.
Esta propiedad determina el uso de OPS como oligonucleótidos antisentido in vitro y en vivo, aplicaciones donde la amplia exposición a las nucleasas es inevitable.
El formato de columna funciona mejor en la investigación y en aplicaciones a grande escala, cuando la síntesis no es compleja.
En 1980 surgió uno de los primeros sintetizadores a mediana escala, fue fabricado por la compañía Biosearch, en Novato, CA (8800).
La escala de 8800 estaba limitada al flujo requerido para fluidizar el soporte.
Esto era una tarea de diseño fácil y algunas estrategias semi automáticas funcionaban sin hacer ninguna modificación al equipo LC que ya existía.
La plataforma inicial llamada VLSS que significa sintetizadores a muy grande escala por sus siglas en inglés, utilizaba columnas de cromatografía líquida como reactores y podía sintetizar hasta 75 milimoles de material.
El diseño VLSS continuó perfeccionándose y continúa en el sintetizador Qmaster[112] que es una plataforma a pequeña escala que produce cantidades de miligramos a gramos de oligonucleótidos sintetizados.