Microcompartimiento bacteriano
Los microcompartimientos bacterianos (MCB o BMC por sus siglas en inglés) son orgánulos bacterianos que consisten en una cubierta proteica que encierra ciertas enzimas y otras proteínas.
Hasta la década de 1990, se pensaba que los carboxisomas eran una rareza confinada a ciertas bacterias autótrofas.
El término metabolosoma se usa para referirse a tales BMC catabólicas (en contraste con el carboxisoma autotrófico).
En 2014, una encuesta exhaustiva identificó 23 loci diferentes que codifican hasta 10 BMC funcionalmente distintos en 23 filos bacterianos.
Se han determinado las estructuras cristalinas de varias de estas proteínas, lo que demuestra que se ensamblan en hexámeros cíclicos, normalmente con un pequeño poro en el centro.
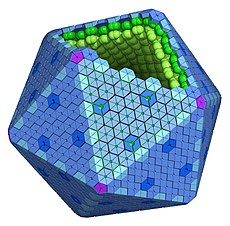
Se necesitan doce unidades pentagonales para cubrir los vértices de una capa icosaédrica.
De manera similar, para el PDU BMC, la cáscara debe ser permeable al propanodiol, propanol, propionilfosfato y potencialmente también vitamina B12, pero está claro que el propionaldehído se secuestra de alguna manera para prevenir daño celular.
Para metabolitos más grandes, es evidente un mecanismo de activación en algunas proteínas BMC-T.
El aldehído se fija inicialmente a la coenzima A por una aldehído deshidrogenasa dependiente de NAD +, pero estos dos cofactores deben reciclarse, ya que aparentemente no pueden cruzar la capa.
Parece que la mayoría, si no todos, los metabolosomas utilizan estas enzimas centrales.
El PDU BMC generalmente está codificado por un locus de 21 genes.
El 1,2-propanodiol se deshidrata a propionaldehído por la propanodiol deshidratasa, que requiere vitamina B12 como cofactor.
El propionaldehído causa mutaciones en el ADN y, como resultado, es tóxico para las células, lo que posiblemente explica por qué este compuesto está secuestrado dentro de un BMC, los productos finales del PDU BMC son propanol y propionil-fosfato, que luego se desfosforila a propionato, generando un ATP.
Algunas bacterias, codifican un único locus en el que están presentes genes para las BMC de PDU y EUT.
Se propone que la enzima de radical glicilo deshidrate el propanodiol a propionaldehído, que luego se procesa de una manera idéntica a la PDU canónica BMC.
Algunas micrografías indican que su ensamblaje ocurre como una coalescencia simultánea de enzimas y proteínas de la cáscara en oposición a la forma aparentemente escalonada observada para los beta-carboxisomas.
Además, muchas proteínas encapsuladas contienen extensiones terminales que son sorprendentemente similares al péptido C-terminal de CcmN que recluta proteínas de caparazón.
Se obtuvieron resultados similares para el GRM BMC, el cual ambos azúcares inducen los genes que codifican el BMC, así como los que codifican las enzimas disimilatorias de fucosa y ramnosa.
El descubrimiento del péptido de encapsulación en los extremos de algunas proteínas asociadas a BMC proporciona un medio para comenzar a diseñar BMC personalizadas fusionando proteínas extrañas a este péptido y co-expresándolo con proteínas de la cubierta.
Por ejemplo, al agregar este péptido a la piruvato descarboxilasa y la alcohol deshidrogenasa, los investigadores han diseñado un biorreactor de etanol.