
La variación del número de copias ( CNV ) es un fenómeno en el que se repiten secciones del genoma y el número de repeticiones en el genoma varía entre individuos. [1] La variación del número de copias es un tipo de variación estructural : concretamente, es un tipo de evento de duplicación o eliminación que afecta a un número considerable de pares de bases. [2] Aproximadamente dos tercios del genoma humano completo pueden estar compuestos de repeticiones [3] y entre el 4,8% y el 9,5% del genoma humano pueden clasificarse como variaciones en el número de copias. [4] En los mamíferos , las variaciones en el número de copias juegan un papel importante en la generación de la variación necesaria en la población, así como en el fenotipo de la enfermedad. [1]
Las variaciones en el número de copias generalmente se pueden clasificar en dos grupos principales: repeticiones cortas y repeticiones largas. Sin embargo, no existen límites claros entre los dos grupos y la clasificación depende de la naturaleza de los loci de interés. Las repeticiones cortas incluyen principalmente repeticiones de dinucleótidos (dos nucleótidos repetidos, por ejemplo, ACACAC...) y repeticiones de trinucleótidos. Las repeticiones largas incluyen repeticiones de genes completos. Esta clasificación basada en el tamaño de la repetición es el tipo de clasificación más obvio, ya que el tamaño es un factor importante al examinar los tipos de mecanismos que probablemente dieron lugar a las repeticiones, [5] de ahí los probables efectos de estas repeticiones en el fenotipo.
Uno de los ejemplos más conocidos de variación del número de copias cortas es la repetición de trinucleótidos de los pares de bases CAG en el gen de la Huntingtina responsable del trastorno neurológico de la enfermedad de Huntington . [6] Para este caso particular, una vez que el trinucleótido CAG se repite más de 36 veces en una expansión de repetición de trinucleótido , la enfermedad de Huntington probablemente se desarrollará en el individuo y probablemente será heredada por su descendencia. [6] El número de repeticiones del trinucleótido CAG está inversamente correlacionado con la edad de aparición de la enfermedad de Huntington. [7] A menudo se piensa que estos tipos de repeticiones cortas se deben a errores en la actividad de la polimerasa durante la replicación , incluido el deslizamiento de la polimerasa, el cambio de plantilla y el cambio de bifurcación, que se analizarán en detalle más adelante. El tamaño de repetición corto de estas variaciones en el número de copias se presta a errores en la polimerasa, ya que estas regiones repetidas son propensas a que la polimerasa las reconozca erróneamente y las regiones replicadas pueden replicarse nuevamente, lo que genera copias adicionales de la repetición. [8] Además, si estas repeticiones de trinucleótidos están en el mismo marco de lectura en la porción codificante de un gen, puede conducir a una cadena larga del mismo aminoácido , posiblemente creando agregados de proteínas en la célula, [7] y si Estas repeticiones cortas caen en la porción no codificante del gen, lo que puede afectar la expresión y regulación del gen . Por otro lado, es menos común identificar en el genoma un número variable de repeticiones de genes completos. Un ejemplo de repetición de un gen completo es el gen de la alfa-amilasa 1 ( AMY1 ) que codifica la alfa-amilasa y que tiene una variación significativa en el número de copias entre diferentes poblaciones con diferentes dietas. [9] Aunque el mecanismo específico que permite que el gen AMY1 aumente o disminuya su número de copias sigue siendo un tema de debate, algunas hipótesis sugieren que la unión de extremos no homólogos o la unión de extremos mediada por microhomología probablemente sea responsable de estos genes completos. repite. [9] Las repeticiones de genes completos tienen efectos inmediatos en la expresión de ese gen en particular, y el hecho de que la variación del número de copias del gen AMY1 se haya relacionado con la dieta es un ejemplo notable de adaptación evolutiva humana reciente. [9]Aunque estos son los grupos generales en los que se agrupan las variaciones del número de copias, el número exacto de pares de bases que afectan las variaciones del número de copias depende de los loci específicos de interés. Actualmente, utilizando datos de todas las variaciones del número de copias reportadas, el tamaño medio de la variante del número de copias es de alrededor de 118 kb y la mediana es de alrededor de 18 kb. [10]
En términos de la arquitectura estructural de las variaciones del número de copias, la investigación ha sugerido y definido regiones críticas en el genoma donde las variaciones del número de copias están cuatro veces más enriquecidas. [2] Estas regiones de puntos críticos se definieron como regiones que contienen repeticiones largas que son entre un 90 % y un 100 % similares, conocidas como duplicaciones segmentarias , ya sea en tándem o intercaladas y, lo más importante, estas regiones de puntos críticos tienen una mayor tasa de reordenamiento cromosómico . [2] Se pensaba que estos reordenamientos cromosómicos a gran escala daban lugar a variaciones normales y enfermedades genéticas , incluidas variaciones en el número de copias. [1] Además, estos puntos críticos de variación del número de copias son consistentes en muchas poblaciones de diferentes continentes, lo que implica que estos puntos críticos fueron adquiridos de forma independiente por todas las poblaciones y transmitidos de generación en generación, o se adquirieron en la evolución humana temprana antes de que las poblaciones se dividieran. esto último parece más probable. [1] Por último, los sesgos espaciales de la ubicación en la que las variaciones del número de copias están más densamente distribuidas no parecen ocurrir en el genoma. [1] Aunque originalmente se detectó mediante hibridación fluorescente in situ y análisis de microsatélites que las repeticiones del número de copias se localizan en regiones que son altamente repetitivas, como los telómeros , centrómeros y heterocromatina , [11] estudios recientes de todo el genoma han concluido lo contrario. [2] Es decir, las regiones subteloméricas y las regiones pericentroméricas son donde se encuentran la mayoría de los puntos críticos de reordenamiento cromosómico, y no hay un aumento considerable en las variaciones del número de copias en esa región. [2] Además, estas regiones de puntos críticos de reordenamiento cromosómico no tienen un número de genes disminuido, lo que nuevamente implica que existe un sesgo espacial mínimo en la ubicación genómica de las variaciones del número de copias. [2]
Inicialmente se pensó que la variación del número de copias ocupaba una porción extremadamente pequeña e insignificante del genoma a través de observaciones citogenéticas . [12] Las variaciones en el número de copias generalmente se asociaron solo con pequeñas repeticiones en tándem o trastornos genéticos específicos, [13] por lo tanto, las variaciones en el número de copias inicialmente solo se examinaron en términos de loci específicos. Sin embargo, los avances tecnológicos llevaron a un número cada vez mayor de formas altamente precisas de identificar y estudiar las variaciones del número de copias. Las variaciones en el número de copias se estudiaron originalmente mediante técnicas citogenéticas, que son técnicas que permiten observar la estructura física del cromosoma. [12] Una de estas técnicas es la hibridación fluorescente in situ (FISH), que implica la inserción de sondas fluorescentes que requieren un alto grado de complementariedad en el genoma para su unión. [10] La hibridación genómica comparativa también se usó comúnmente para detectar variaciones en el número de copias mediante visualización del fluoróforo y luego comparar la longitud de los cromosomas. [10] Un inconveniente importante de estas primeras técnicas es que la resolución genómica es relativamente baja y sólo se pueden detectar repeticiones grandes, como repeticiones de genes completos. [ cita necesaria ]
Los avances recientes en las tecnologías genómicas dieron lugar a muchos métodos importantes que tienen una resolución genómica extremadamente alta y, como resultado, se ha informado de un número cada vez mayor de variaciones en el número de copias en el genoma. [10] Inicialmente, estos avances implicaban el uso de una matriz de cromosomas artificiales bacterianos (BAC) con alrededor de 1 megabase de intervalos en todo el gen; [14] Los BAC también pueden detectar variaciones en el número de copias en puntos críticos de reordenamiento, lo que permite la detección de 119 nuevas variaciones en el número de copias. [2] La secuenciación genómica de alto rendimiento ha revolucionado el campo de la genómica humana y se han realizado estudios in silico para detectar variaciones en el número de copias en el genoma. [2] Las secuencias de referencia se han comparado con otras secuencias de interés utilizando fósmidos controlando estrictamente que los clones de fósmidos tengan 40 kb. [15] Las lecturas finales de secuenciación proporcionarían información adecuada para alinear la secuencia de referencia con la secuencia de interés, y cualquier desalineación es fácilmente perceptible, por lo que se concluye que son variaciones en el número de copias dentro de esa región del clon. [15] Este tipo de técnica de detección ofrece una alta resolución genómica y una ubicación precisa de la repetición en el genoma, y también puede detectar otros tipos de variación estructural como las inversiones. [10]
Además, otra forma de detectar la variación del número de copias es utilizar polimorfismos de un solo nucleótido (SNP). [10] Debido a la abundancia de datos de SNP humanos, la dirección de detección de la variación del número de copias ha cambiado para utilizar estos SNP. [16] Basándose en el hecho de que la recombinación humana es relativamente rara y que muchos eventos de recombinación ocurren en regiones específicas del genoma conocidas como puntos críticos de recombinación, el desequilibrio de ligamiento se puede utilizar para identificar variaciones en el número de copias. [16] Se han realizado esfuerzos para asociar variaciones en el número de copias con SNP de haplotipos específicos mediante el análisis del desequilibrio de ligamiento; utilizando estas asociaciones, se pueden reconocer variaciones en el número de copias en el genoma utilizando SNP como marcadores. Las técnicas de secuenciación de próxima generación , incluida la secuenciación de lectura corta y larga, se utilizan cada vez más en la actualidad y han comenzado a reemplazar las técnicas basadas en matrices para detectar variaciones en el número de copias. [17] [18] A diferencia de las técnicas basadas en matrices, los métodos de detección basados en secuenciación identifican fácilmente otras clases de variación estructural , como inversiones y translocaciones . [ cita necesaria ]
Hay dos tipos principales de mecanismos moleculares para la formación de variaciones en el número de copias: de base homóloga y de base no homóloga. [5] Aunque se han presentado muchas sugerencias, la mayoría de estas teorías son especulaciones y conjeturas. No existe evidencia concluyente que correlacione una variación específica del número de copias con un mecanismo específico.
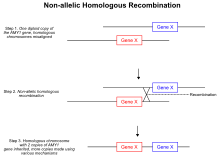
Una de las teorías mejor reconocidas que conduce a variaciones en el número de copias, así como a eliminaciones e inversiones, son las recombinaciones homólogas no alélicas . [19] Durante la recombinación meiótica , los cromosomas homólogos se emparejan y forman dos roturas bicatenarias terminadas que conducen a uniones Holliday . Sin embargo, en el mecanismo aberrante, durante la formación de las uniones Holliday, las roturas de la doble hebra se desalinean y el cruce aterriza en posiciones no alélicas en el mismo cromosoma. Cuando se resuelve la unión de Holliday, el evento de cruce desigual permite la transferencia de material genético entre los dos cromosomas homólogos y, como resultado, se repite una porción del ADN en ambos homólogos. [19] Dado que las regiones repetidas ya no se segregan de forma independiente , la región duplicada del cromosoma se hereda. Otro tipo de mecanismo basado en recombinación homóloga que puede conducir a una variación en el número de copias se conoce como replicación inducida por rotura. [20] Cuando se produce una rotura bicatenaria en el genoma inesperadamente, la célula activa vías que median la reparación de la rotura. [20] Los errores en la reparación de la rotura, similares a la recombinación homóloga no alélica, pueden conducir a un aumento en el número de copias de una región particular del genoma. Durante la reparación de una rotura de doble cadena, el extremo roto puede invadir su cromosoma homólogo en lugar de volver a unirse a la cadena original. [20] Como en el mecanismo de recombinación homóloga no alélica, una copia adicional de una región particular se transfiere a otro cromosoma, lo que lleva a un evento de duplicación. Además, se ha descubierto que las proteínas cohesina ayudan en el sistema de reparación de roturas de doble cadena al sujetar los dos extremos muy cerca, lo que evita la invasión intercromosómica de los extremos. [21] Si por algún motivo, como la activación del ARN ribosomal , la actividad de la cohesina se ve afectada, entonces puede haber un aumento local en los errores de reparación de roturas de doble cadena. [21]
La otra clase de posibles mecanismos que se supone que conducen a variaciones en el número de copias no tiene una base homóloga. Para distinguir entre este y los mecanismos basados en homólogos, es necesario comprender el concepto de homología. El emparejamiento homólogo de cromosomas implica el uso de hebras de ADN que son muy similares entre sí (~97%) y estas hebras deben ser más largas que cierta longitud para evitar emparejamientos cortos pero muy similares. [5] Los emparejamientos no homólogos, por otro lado, dependen solo de unos pocos pares de bases de similitud entre dos cadenas, por lo tanto, es posible que los materiales genéticos se intercambien o dupliquen en el proceso de reparaciones de doble cadena basadas en no homólogos. [5]
Un tipo de mecanismo basado en no homólogos es el mecanismo de unión de extremos no homólogos o mecanismo de unión de extremos de microhomología . [22] Estos mecanismos también participan en la reparación de roturas de doble cadena, pero no requieren homología o una microhomología limitada. [5] Cuando se reparan estas hebras, a menudo se agregan pequeñas eliminaciones o inserciones en la hebra reparada. Es posible que se inserten retrotransposones en el genoma a través de este sistema de reparación. [22] Si los retrotransposones se insertan en una posición no alélica del cromosoma, la recombinación meiótica puede impulsar la inserción para que se recombine en la misma cadena que una copia ya existente de la misma región. Otro mecanismo es el ciclo de ruptura-fusión-puente, que involucra cromátidas hermanas que han perdido su región telomérica debido a roturas de doble cadena. [23] Se propone que estas cromátidas hermanas se fusionen para formar un cromosoma dicéntrico y luego se segreguen en dos núcleos diferentes. [23] Debido a que separar el cromosoma dicéntrico provoca una rotura de doble cadena, las regiones finales pueden fusionarse con otras roturas de doble cadena y repetir el ciclo. [23] La fusión de dos cromátidas hermanas puede causar una duplicación invertida y cuando estos eventos se repiten a lo largo del ciclo, la región invertida se repetirá, lo que provocará un aumento en el número de copias. [23] El último mecanismo que puede conducir a variaciones en el número de copias es el deslizamiento de la polimerasa, que también se conoce como cambio de plantilla. [24] Durante la replicación normal del ADN, se requiere que la polimerasa de la cadena retrasada suelte y vuelva a sujetar la región de replicación continuamente. [24] Cuando ya existen repeticiones a pequeña escala en la secuencia de ADN, la polimerasa puede "confundirse" cuando se vuelve a sujetar para continuar con la replicación y, en lugar de sujetarse a los pares de bases correctos, puede desplazar algunos pares de bases y replicar una porción. de la región repetida nuevamente. [24] Tenga en cuenta que, aunque esto se ha observado experimentalmente y es un mecanismo ampliamente aceptado, las interacciones moleculares que llevaron a este error siguen siendo desconocidas. Además, debido a que este tipo de mecanismo requiere que la polimerasa salte alrededor de la cadena de ADN y es poco probable que la polimerasa pueda volver a sujetarse en otro locus a algunas kilobases de distancia, esto es más aplicable a repeticiones cortas como las repeticiones de dinucleótidos o trinucleótidos. [25]

La amilasa es una enzima de la saliva responsable de la descomposición del almidón en monosacáridos , y un tipo de amilasa está codificado por el gen de la alfa-amilasa ( AMY1 ). [9] El locus AMY1 , así como la enzima amilasa, es uno de los genes más estudiados y secuenciados del genoma humano. Sus homólogos también se encuentran en otros primates y, por lo tanto, es probable que el gen AMY1 de los primates sea ancestral del gen AMY1 humano y se haya adaptado temprano en la evolución de los primates. [9] AMY1 es uno de los genes mejor estudiados y tiene una amplia gama de números variables de copias en diferentes poblaciones humanas. [9] El gen AMY1 es también uno de los pocos genes estudiados que muestra evidencia convincente que correlaciona su función proteica con su número de copias. [9] Se sabe que el número de copias altera la transcripción y los niveles de traducción de un gen en particular; sin embargo, las investigaciones han demostrado que la relación entre los niveles de proteína y el número de copias es variable. [26] En los genes AMY1 de los estadounidenses de origen europeo se encuentra que la concentración de amilasa salival está estrechamente correlacionada con el número de copias del gen AMY1 . [9] Como resultado, se planteó la hipótesis de que el número de copias del gen AMY1 está estrechamente relacionado con su función proteica, que es digerir el almidón. [9]
Se ha descubierto que el número de copias del gen AMY1 está correlacionado con diferentes niveles de almidón en las dietas de diferentes poblaciones. [9] Ocho poblaciones de diferentes continentes se clasificaron en dietas altas en almidón y dietas bajas en almidón y su número de copias del gen AMY1 se visualizó utilizando FISH y qPCR de alta resolución . [9] Se descubrió que las poblaciones de dietas altas en almidón que consisten en poblaciones japonesas, hadza y europeas americanas tenían un número promedio de copias de AMY1 significativamente mayor (dos veces mayor) que las poblaciones de dietas bajas en almidón, incluidas Biaka, Mbuti, Datog y poblaciones yakutas. [9] Se planteó la hipótesis de que los niveles de almidón en la dieta habitual, el sustrato de AMY1, pueden afectar directamente el número de copias del gen AMY1 . [9] Dado que se concluyó que el número de copias de AMY1 está directamente correlacionado con la amilasa salival, [9] cuanto más almidón esté presente en la dieta diaria de la población, más favorable será evolutivamente tener múltiples copias del gen AMY1 . El gen AMY1 fue el primer gen que proporcionó pruebas sólidas de la evolución a nivel genético molecular . [26] Además, utilizando la hibridación genómica comparada , se compararon las variaciones en el número de copias de los genomas completos de la población japonesa con el de la población Yakut. [9] Se descubrió que la variación del número de copias del gen AMY1 era significativamente diferente de la variación del número de copias en otros genes o regiones del genoma, lo que sugiere que el gen AMY1 estaba bajo una fuerte presión selectiva que tenía poca o ninguna influencia en las otras variaciones en el número de copias. [9] Finalmente, se comparó la variabilidad de la longitud de 783 microsatélites entre las dos poblaciones con la variabilidad del número de copias del gen AMY1 . Se descubrió que el rango del número de copias del gen AMY1 era mayor que el de más del 97% de los microsatélites examinados. [9] Esto implica que la selección natural jugó un papel considerable en la configuración del número promedio de genes AMY1 en estas dos poblaciones. [9] Sin embargo, como solo se estudiaron seis poblaciones, es importante considerar la posibilidad de que pueda haber otros factores en su dieta o cultura que influyeron en el número de copias de AMY1 además del almidón.

Aunque no está claro cuándo comenzó a aumentar el número de copias del gen AMY1 , se sabe y confirma que el gen AMY1 existía en los primeros primates. Se descubrió que los chimpancés , los parientes evolutivos más cercanos a los humanos, tienen dos copias diploides del gen AMY1 que es idéntico en longitud al gen AMY1 humano, [9] que es significativamente menor que el de los humanos. Por otro lado, se descubrió que los bonobos , también un pariente cercano de los humanos modernos, tenían más de dos copias diploides del gen AMY1 . [9] No obstante, los genes AMY1 del bonobo fueron secuenciados y analizados, y se encontró que las secuencias codificantes de los genes AMY1 estaban alteradas, lo que puede conducir a la producción de amilasa salival disfuncional. [9] Se puede inferir de los resultados que el aumento en el número de copias AMY1 del bonobo probablemente no esté correlacionado con la cantidad de almidón en su dieta. Además, se planteó la hipótesis de que el aumento en el número de copias comenzó recientemente durante la evolución temprana de los homínidos , ya que ninguno de los grandes simios tenía más de dos copias del gen AMY1 que producía proteína funcional. [9] Además, se especuló que el aumento en el número de copias de AMY1 comenzó hace unos 20.000 años, cuando los humanos pasaron de un estilo de vida de cazadores-recolectores a sociedades agrícolas , que también fue cuando los humanos dependían en gran medida de tubérculos con alto contenido de almidón. [9] Esta hipótesis, aunque lógica, carece de evidencia experimental debido a las dificultades para recopilar información sobre el cambio de las dietas humanas, especialmente sobre tubérculos con alto contenido de almidón, ya que no pueden observarse ni probarse directamente. Los avances recientes en la secuenciación del ADN han permitido a los investigadores secuenciar ADN más antiguo, como el de los neandertales , con cierto grado de precisión. Quizás la secuenciación del ADN neandertal pueda proporcionar un marcador temporal de cuándo aumentó el número de copias del gen AMY1 y ofrecer información sobre la dieta humana y la evolución genética.
Actualmente se desconoce qué mecanismo dio lugar a la duplicación inicial del gen de la amilasa, y puede implicar que la inserción de las secuencias retrovirales se debió a una unión de extremos no homólogos, lo que provocó la duplicación del gen AMY1 . [27] Sin embargo, actualmente no hay evidencia que respalde esta teoría y, por lo tanto, esta hipótesis sigue siendo una conjetura. El origen reciente del gen AMY1 de copias múltiples implica que, dependiendo del entorno, el número de copias del gen AMY1 puede aumentar y disminuir muy rápidamente en relación con los genes que no interactúan tan directamente con el medio ambiente. [26] El gen AMY1 es un excelente ejemplo de cómo la dosis de genes afecta la supervivencia de un organismo en un entorno determinado. Las múltiples copias del gen AMY1 dan a quienes dependen más de dietas ricas en almidón una ventaja evolutiva, por lo que el alto número de copias del gen persiste en la población. [26]
Entre las neuronas del cerebro humano , son frecuentes las variaciones en el número de copias derivadas somáticamente. [28] Las variaciones en el número de copias muestran una amplia variabilidad (9 a 100% de las neuronas cerebrales en diferentes estudios). La mayoría de las alteraciones tienen un tamaño de entre 2 y 10 Mb y las eliminaciones superan con creces a las amplificaciones. [28] Las variaciones en el número de copias parecen ser mayores en las células cerebrales que en otros tipos de células. [28] Una fuente probable de variación en el número de copias es la reparación incorrecta del daño del ADN . [ cita necesaria ]
La duplicación genómica y la triplicación del gen parecen ser una causa rara de la enfermedad de Parkinson , aunque más común que las mutaciones puntuales. [29]
Las variantes del número de copias en el gen RCL1 están asociadas con una variedad de fenotipos neuropsiquiátricos en niños. [30]

Recientemente, se ha debatido conectar las variaciones en el número de copias con las familias de genes . Las familias de genes se definen como un conjunto de genes relacionados que cumplen funciones similares pero que tienen diferencias temporales o espaciales menores y estos genes probablemente derivan de un gen ancestral . [26] La razón principal por la que las variaciones en el número de copias están relacionadas con las familias de genes es que existe la posibilidad de que los genes de una familia se hayan derivado de un gen ancestral que se duplicó en diferentes copias. [26] Las mutaciones se acumulan a lo largo del tiempo en los genes y con la selección natural actuando sobre los genes, algunas mutaciones conducen a ventajas ambientales que permiten que esos genes se hereden y, finalmente, se separan familias de genes claras. Un ejemplo de una familia de genes que puede haberse creado debido a variaciones en el número de copias es la familia de genes de globina . La familia de genes de globina es una elaborada red de genes que consta de genes de globina alfa y beta , incluidos genes que se expresan tanto en embriones como en adultos, así como pseudogenes . [31] Estos genes de globina en la familia de las globinas están todos bien conservados y solo difieren en una pequeña porción del gen, lo que indica que se derivaron de un gen ancestral común, tal vez debido a la duplicación del gen de globina inicial. [31]
Las investigaciones han demostrado que las variaciones en el número de copias son significativamente más comunes en genes que codifican proteínas que interactúan directamente con el medio ambiente que en proteínas que participan en actividades celulares básicas. [32] Se sugirió que el efecto de la dosis genética que acompaña a la variación del número de copias puede provocar efectos perjudiciales si se alteran las funciones celulares esenciales, por lo que las proteínas implicadas en las vías celulares están sujetas a una fuerte selección purificadora . [32] Además, las proteínas funcionan juntas e interactúan con proteínas de otras vías, por lo que es importante ver los efectos de la selección natural en las vías biomoleculares en lugar de en proteínas individuales. Dicho esto, se descubrió que las proteínas en la periferia de la vía se enriquecen en las variaciones del número de copias, mientras que las proteínas en el centro de las vías se agotan en las variaciones del número de copias. [33] Se explicó que las proteínas en la periferia de la vía interactúan con menos proteínas y, por lo tanto, un cambio en la dosis de proteína afectado por un cambio en el número de copias puede tener un efecto menor en el resultado general de la vía celular. [33]
En los últimos años, los investigadores parecen haber cambiado su enfoque de la detección, localización y secuenciación de variaciones en el número de copias a análisis en profundidad del papel de estas variaciones en el número de copias en el genoma humano y en la naturaleza en general. Se necesitan pruebas para validar aún más la relación entre las variaciones del número de copias y las familias de genes, así como el papel que desempeña la selección natural en la configuración de estas relaciones y cambios. Además, los investigadores también pretenden dilucidar los mecanismos moleculares implicados en las variaciones del número de copias, ya que pueden revelar información esencial sobre las variaciones estructurales en general. Dando un paso atrás, el área de variación estructural en el genoma humano parece ser un tema de investigación en rápido crecimiento. Estos datos de investigación no sólo pueden proporcionar evidencia adicional de la evolución y la selección natural, sino que también pueden usarse para desarrollar tratamientos para una amplia gama de enfermedades genéticas. [ cita necesaria ]