
El centrómero une un par de cromátidas hermanas durante la división celular. Esta región estrecha del cromosoma conecta las cromátidas hermanas, creando un brazo corto (p) y un brazo largo (q) en las cromátidas. Durante la mitosis , las fibras del huso se unen al centrómero a través del cinetocoro .
La función física del centrómero es actuar como sitio de ensamblaje de los cinetocoros , una estructura multiproteica altamente compleja que es responsable de los eventos reales de la segregación cromosómica , es decir, la unión de los microtúbulos y la señalización a la maquinaria del ciclo celular cuando todos los cromosomas han adoptado las uniones correctas al huso , de modo que sea seguro que la división celular se complete y que las células entren en anafase .
En términos generales, existen dos tipos de centrómeros. Los "centrómeros puntuales" se unen a proteínas específicas que reconocen secuencias de ADN particulares con alta eficiencia. Cualquier fragmento de ADN que contenga la secuencia de ADN del centrómero puntual formará típicamente un centrómero si está presente en la especie apropiada. Los centrómeros puntuales mejor caracterizados son los de la levadura en ciernes, Saccharomyces cerevisiae . "Centrómeros regionales" es el término acuñado para describir la mayoría de los centrómeros, que normalmente se forman en regiones de secuencia de ADN preferida, pero que también pueden formarse en otras secuencias de ADN. La señal para la formación de un centrómero regional parece ser epigenética . La mayoría de los organismos, desde la levadura de fisión Schizosaccharomyces pombe hasta los humanos, tienen centrómeros regionales.
En cuanto a la estructura de los cromosomas mitóticos, los centrómeros representan una región estrecha del cromosoma (a menudo denominada constricción primaria) donde dos cromátidas hermanas idénticas están en contacto más cercano. Cuando las células entran en mitosis, las cromátidas hermanas (las dos copias de cada molécula de ADN cromosómico resultantes de la replicación del ADN en forma de cromatina) se unen a lo largo de su longitud mediante la acción del complejo de cohesión . Ahora se cree que este complejo se libera principalmente de los brazos cromosómicos durante la profase, de modo que cuando los cromosomas se alinean en el plano medio del huso mitótico (también conocido como placa de metafase), el último lugar donde se unen entre sí es en la cromatina dentro y alrededor del centrómero.
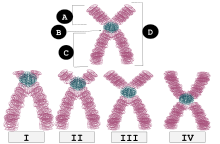
En los seres humanos, las posiciones del centrómero definen el cariotipo cromosómico , en el que cada cromosoma tiene dos brazos, p (el más corto de los dos) y q (el más largo). El brazo corto "p" recibe su nombre de la palabra francesa "petit", que significa "pequeño". [1] La posición del centrómero en relación con cualquier cromosoma lineal en particular se utiliza para clasificar los cromosomas como metacéntricos, submetacéntricos, acrocéntricos, telocéntricos u holocéntricos. [2] [3]
Metacéntrico significa que el centrómero está ubicado a mitad de camino entre los extremos de los cromosomas, lo que hace que los brazos tengan aproximadamente la misma longitud. Cuando los centrómeros son metacéntricos, los cromosomas parecen tener forma de "x".
Submetacéntrico significa que el centrómero está ubicado debajo de la mitad, con un brazo cromosómico más corto que el otro, lo que a menudo da como resultado una forma de L.
El centrómero de un cromosoma acrocéntrico está situado de forma que uno de los brazos del cromosoma es mucho más corto que el otro. El "acro-" de acrocéntrico se refiere a la palabra griega que significa "pico". El genoma humano tiene seis cromosomas acrocéntricos, incluidos cinco cromosomas autosómicos ( 13 , 14 , 15 , 21 , 22 ) y el cromosoma Y.
Los brazos p acrocéntricos cortos contienen poco material genético y pueden translocarse sin daño significativo, como en una translocación robertsoniana equilibrada . Además de algunos genes codificadores de proteínas, los brazos p acrocéntricos humanos también contienen regiones organizadoras del nucléolo (NOR), a partir de las cuales se transcribe el ARN ribosómico . Sin embargo, una proporción de brazos p acrocéntricos en líneas celulares y tejidos de donantes humanos normales no contienen NOR detectables. [4] El genoma del caballo doméstico incluye un cromosoma metacéntrico que es homólogo a dos cromosomas acrocéntricos en el caballo de Przewalski conespecífico pero no domesticado . Esto puede reflejar la fijación de una translocación robertsoniana equilibrada en caballos domésticos o, por el contrario, la fijación de la fisión de un cromosoma metacéntrico en dos cromosomas acrocéntricos en los caballos de Przewalski. Una situación similar existe entre los genomas humanos y de los grandes simios, con una reducción de dos cromosomas acrocéntricos en los grandes simios a un cromosoma metacéntrico en los humanos (véase aneuploidía y el cromosoma humano 2 ).
Muchas enfermedades que son resultado de translocaciones desequilibradas afectan con mayor frecuencia a los cromosomas acrocéntricos que a otros cromosomas no acrocéntricos. Los cromosomas acrocéntricos suelen estar ubicados dentro y alrededor del nucléolo . Como resultado, estos cromosomas tienden a estar menos densamente empaquetados que los cromosomas en la periferia nuclear. De manera consistente, las regiones cromosómicas que están menos densamente empaquetadas también son más propensas a las translocaciones cromosómicas en los cánceres.
Los cromosomas telocéntricos tienen un centrómero en un extremo del cromosoma y, por lo tanto, presentan solo un brazo a nivel citológico (microscópico). No están presentes en los humanos, pero pueden formarse a través de errores cromosómicos celulares. Los cromosomas telocéntricos se dan de forma natural en muchas especies, como el ratón doméstico , en el que todos los cromosomas, excepto el Y, son telocéntricos.
Los centrómeros de los cromosomas subtelocéntricos se encuentran entre la mitad y el final de los cromosomas, pero residen más cerca del final de los cromosomas.
Un cromosoma acéntrico es un fragmento de un cromosoma que carece de centrómero. Dado que los centrómeros son el punto de unión de las fibras del huso en la división celular, los fragmentos acéntricos no se distribuyen uniformemente entre las células hijas durante la división celular. Como resultado, una célula hija carecerá del fragmento acéntrico y podrían producirse consecuencias perjudiciales.
Los eventos de ruptura de cromosomas también pueden generar cromosomas acéntricos o fragmentos acéntricos.
Un cromosoma dicéntrico es un cromosoma anormal con dos centrómeros, que puede ser inestable a través de las divisiones celulares. Puede formarse a través de la translocación o fusión de dos segmentos cromosómicos, cada uno con un centrómero. Algunos reordenamientos producen cromosomas dicéntricos y fragmentos acéntricos que no pueden unirse a los husos en la mitosis. [5] La formación de cromosomas dicéntricos se ha atribuido a procesos genéticos, como la translocación robertsoniana [6] y la inversión paracéntrica. [7] Los cromosomas dicéntricos pueden tener una variedad de destinos, incluida la estabilidad mitótica. [8] En algunos casos, su estabilidad proviene de la inactivación de uno de los dos centrómeros para formar un cromosoma funcionalmente monocéntrico capaz de transmisión normal a las células hijas durante la división celular. [9]
Por ejemplo, el cromosoma humano 2 , que se cree que es el resultado de una translocación robertsoniana en algún punto de la evolución entre los grandes simios y el Homo , tiene un segundo centrómero vestigial cerca de la mitad de su brazo largo. [10]
El cromosoma monocéntrico es un cromosoma que tiene un solo centrómero en un cromosoma y forma una constricción estrecha.
Los centrómeros monocéntricos son la estructura más común en el ADN altamente repetitivo de plantas y animales. [11]
A diferencia de los cromosomas monocéntricos, los cromosomas holocéntricos no tienen una constricción primaria distintiva cuando se observan en la mitosis. En cambio, las fibras del huso se unen a lo largo de casi toda la longitud (griego: holo-) del cromosoma. En los cromosomas holocéntricos, las proteínas centroméricas, como CENPA (CenH3), se extienden por todo el cromosoma. [12] El nematodo, Caenorhabditis elegans , es un ejemplo bien conocido de un organismo con cromosomas holocéntricos, [13] pero este tipo de centrómero se puede encontrar en varias especies, plantas y animales, en eucariotas. Los holocentrómeros en realidad están compuestos de múltiples unidades de centrómero distribuidas que forman una estructura similar a una línea a lo largo de los cromosomas durante la mitosis. [14] Se implementan estrategias alternativas o no convencionales en la meiosis para lograr el apareamiento y la segregación de cromosomas homólogos necesarios para producir gametos o gametofitos viables para la reproducción sexual.
Existen diferentes tipos de holocentrómeros en diferentes especies, a saber, con o sin secuencias repetitivas de ADN centroméricas y con o sin CenH3 . La holocentricidad ha evolucionado al menos 13 veces de forma independiente en varias algas verdes, protozoos, invertebrados y diferentes familias de plantas. [15] A diferencia de las especies monocéntricas donde los fragmentos acéntricos generalmente se pierden durante la división celular, la rotura de cromosomas holocéntricos crea fragmentos con sitios de unión de fibras del huso normales. [16] Debido a esto, los organismos con cromosomas holocéntricos pueden desarrollar más rápidamente la variación del cariotipo, capaz de curar los cromosomas fragmentados mediante la posterior adición de tapas de telómeros en los sitios de rotura. [17]
Los cromosomas policéntricos tienen varios grupos de cinetocoros, es decir, centromas. El término se superpone parcialmente con "holocéntrico", pero "policéntrico" es claramente preferido cuando se habla de cromosomas monocéntricos formados defectuosamente. También hay cierta ambigüedad real, ya que no hay una línea clara que divida la transición de los cinetocoros que cubren todo el cromosoma a grupos distintos. En otras palabras, la diferencia entre "todo el cromosoma es un centroma" y "el cromosoma no tiene centroma" es confusa y el uso varía. Más allá de que "policentricidad" se usa más en relación con los defectos, no hay una preferencia clara en otros temas como el origen evolutivo o la distribución y la estructura detallada de los cinetocoros (por ejemplo, como se ve en el análisis del etiquetado o el ensamblaje del genoma ). [18] [19] [20] [21]
Incluso grupos claramente diferenciados de proteínas cinetocóricas no necesariamente producen más de una constricción: los cromosomas "metapolicéntricos" presentan una constricción alargada del cromosoma, que une un segmento más largo que sigue siendo visiblemente más corto que las cromátidas. [22] Los cromosomas metapolicéntricos pueden ser un paso en el surgimiento y la supresión del impulso centrómero, un tipo de impulso meiótico que interrumpe la paridad mediante centrómeros monocéntricos que desarrollan proteínas cinetocóricas adicionales para obtener una ventaja durante la meiosis. [23]

En función de las características micrográficas de tamaño, posición del centrómero y en ocasiones la presencia de un satélite cromosómico , los cromosomas humanos se clasifican en los siguientes grupos: [24]
Hay dos tipos de centrómeros. [25] En los centrómeros regionales, las secuencias de ADN contribuyen a la función, pero no la definen. Los centrómeros regionales contienen grandes cantidades de ADN y a menudo están empaquetados en heterocromatina . En la mayoría de los eucariotas , la secuencia de ADN del centrómero consiste en grandes conjuntos de ADN repetitivo (por ejemplo, ADN satélite ) donde la secuencia dentro de los elementos repetidos individuales es similar pero no idéntica. En los humanos, la unidad de repetición centromérica primaria se llama α-satélite (o alfoide), aunque se encuentran otros tipos de secuencias en esta región. [26] Se plantea la hipótesis de que los satélites del centrómero evolucionan mediante un proceso llamado expansión en capas. Evolucionan rápidamente entre especies, y los análisis en ratones salvajes muestran que el número de copias de satélites y la heterogeneidad se relacionan con los orígenes de la población y las subespecies. [27] Además, las secuencias satélite pueden verse afectadas por la endogamia. [27]
Los centrómeros puntuales son más pequeños y compactos. Las secuencias de ADN son necesarias y suficientes para especificar la identidad y la función del centrómero en organismos con centrómeros puntuales. En las levaduras en ciernes, la región del centrómero es relativamente pequeña (aproximadamente 125 pb de ADN) y contiene dos secuencias de ADN altamente conservadas que sirven como sitios de unión para las proteínas esenciales del cinetocoro . [26]
Dado que la secuencia de ADN centromérico no es el determinante clave de la identidad centromérica en los metazoos , se cree que la herencia epigenética juega un papel importante en la especificación del centrómero. [28] Los cromosomas hijos ensamblarán centrómeros en el mismo lugar que el cromosoma padre, independientemente de la secuencia. Se ha propuesto que la variante de histona H3 CENP-A (proteína A del centrómero) es la marca epigenética del centrómero. [29] Surge la pregunta de si debe haber todavía alguna forma original en la que se especifica el centrómero, incluso si posteriormente se propaga epigenéticamente. Si el centrómero se hereda epigenéticamente de una generación a la siguiente, el problema se retrotrae al origen de los primeros metazoos.
Por otra parte, gracias a comparaciones de los centrómeros en los cromosomas X, se han visto variaciones epigenéticas y estructurales en estas regiones. Además, un reciente ensamblaje del genoma humano ha detectado un posible mecanismo de cómo evolucionan las estructuras pericentroméricas y centroméricas, a través de un modelo de expansión en capas para secuencias αSat. Este modelo propone que diferentes repeticiones de secuencias αSat emergen periódicamente y se expanden dentro de un vector activo, desplazando secuencias antiguas y convirtiéndose en el sitio de ensamblaje del cinetocoro. Las αSat pueden originarse del mismo vector o de diferentes vectores. A medida que este proceso se repite en el tiempo, las capas que flanquean el centrómero activo se encogen y se deterioran. Este proceso plantea preguntas sobre la relación entre este proceso evolutivo dinámico y la posición del centrómero. [30]
El ADN centromérico se encuentra normalmente en un estado de heterocromatina , que es esencial para el reclutamiento del complejo de cohesión que media la cohesión de las cromátidas hermanas después de la replicación del ADN, así como para coordinar la separación de las cromátidas hermanas durante la anafase. En esta cromatina, la histona normal H3 se reemplaza por una variante específica del centrómero, CENP-A en humanos. [31] Se cree que la presencia de CENP-A es importante para el ensamblaje del cinetocoro en el centrómero. Se ha demostrado que CENP-C se localiza casi exclusivamente en estas regiones de la cromatina asociada a CENP-A. En las células humanas, se ha descubierto que las histonas están más enriquecidas con H4K20me 3 y H3K9me3 [32], que son modificaciones heterocromáticas conocidas. En Drosophila, las islas de retroelementos son los componentes principales de los centrómeros. [33]
En la levadura Schizosaccharomyces pombe (y probablemente en otros eucariotas), la formación de heterocromatina centromérica está conectada al ARNi . [34] En nematodos como Caenorhabditis elegans , algunas plantas y los órdenes de insectos Lepidoptera y Hemiptera, los cromosomas son "holocéntricos", lo que indica que no hay un sitio primario de unión de microtúbulos o una constricción primaria, y un cinetocoro "difuso" se ensambla a lo largo de toda la longitud del cromosoma.
En casos raros, los neocentrómeros pueden formarse en nuevos sitios en un cromosoma como resultado de un reposicionamiento del centrómero. Este fenómeno es más conocido a partir de estudios clínicos humanos y actualmente hay más de 90 neocentrómeros humanos conocidos identificados en 20 cromosomas diferentes. [35] [36] La formación de un neocentrómero debe estar acoplada con la inactivación del centrómero anterior, ya que los cromosomas con dos centrómeros funcionales ( cromosoma dicéntrico ) darán lugar a la rotura cromosómica durante la mitosis. En algunos casos inusuales, se ha observado que los neocentrómeros humanos se forman espontáneamente en cromosomas fragmentados. Algunas de estas nuevas posiciones eran originalmente eucromáticas y carecen por completo de ADN satélite alfa . Los neocentrómeros carecen de la estructura repetitiva observada en los centrómeros normales, lo que sugiere que la formación del centrómero está controlada principalmente epigenéticamente . [37] [38] Con el tiempo, un neocentrómero puede acumular elementos repetitivos y madurar en lo que se conoce como un nuevo centrómero evolutivo. Hay varios ejemplos bien conocidos en cromosomas de primates donde la posición del centrómero es diferente del centrómero humano del mismo cromosoma y se piensa que son nuevos centrómeros evolutivos. [37] Se ha sugerido que el reposicionamiento del centrómero y la formación de nuevos centrómeros evolutivos es un mecanismo de especiación . [39]
Las proteínas del centrómero también son el objetivo autoantigénico de algunos anticuerpos antinucleares , como los anticuerpos anticentrómero .
Se sabe que la regulación incorrecta del centrómero contribuye a la segregación incorrecta de los cromosomas, que está estrechamente relacionada con el cáncer y el aborto espontáneo. En particular, la sobreexpresión de muchos genes del centrómero se ha relacionado con fenotipos malignos del cáncer. La sobreexpresión de estos genes del centrómero puede aumentar la inestabilidad genómica en los cánceres. La inestabilidad genómica elevada, por un lado, se relaciona con fenotipos malignos; por otro lado, hace que las células tumorales sean más vulnerables a terapias adyuvantes específicas, como ciertas quimioterapias y radioterapias. [40] Recientemente se ha demostrado la inestabilidad del ADN repetitivo del centrómero en el cáncer y el envejecimiento. [41]
Cuando se producen roturas de ADN en los centrómeros en la fase G1 del ciclo celular , las células pueden reclutar la maquinaria de reparación recombinatoria homóloga al sitio dañado, incluso en ausencia de una cromátida hermana . [42] Parece que la reparación recombinatoria homóloga puede ocurrir en roturas centroméricas a lo largo del ciclo celular para prevenir la activación de vías de reparación de ADN mutagénicas inexactas y preservar la integridad centromérica. [42]
La palabra centrómero ( / ˈ s ɛ n t r ə ˌ m ɪər / [43] [44] ) utiliza formas combinadas de centro- y -mero , lo que produce "parte central", que describe la ubicación del centrómero en el centro del cromosoma.
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ){{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace )