La reparación por escisión de nucleótidos es un mecanismo de reparación del ADN . [2] El daño al ADN ocurre constantemente debido a productos químicos (por ejemplo, agentes intercalantes ), radiación y otros mutágenos . Existen tres vías de reparación por escisión para reparar el daño del ADN monocatenario: reparación por escisión de nucleótidos (NER), reparación por escisión de bases (BER) y reparación de errores de coincidencia de ADN (MMR). Si bien la vía BER puede reconocer lesiones específicas no voluminosas en el ADN, solo puede corregir bases dañadas que son eliminadas por glicosilasas específicas . De manera similar, la vía MMR solo se dirige a pares de bases Watson-Crick que no coinciden .
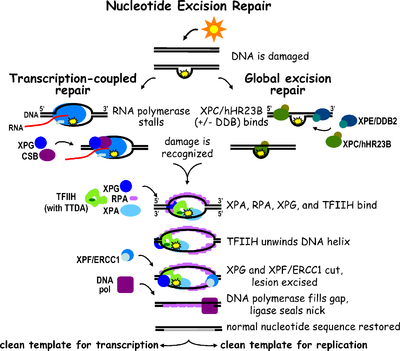
La reparación por escisión de nucleótidos (NER) es un mecanismo de escisión particularmente importante que elimina el daño del ADN inducido por la luz ultravioleta (UV). El daño del ADN por rayos UV da como resultado aductos de ADN voluminosos ; estos aductos son en su mayoría dímeros de timina y fotoproductos 6,4. El reconocimiento del daño conduce a la eliminación de un segmento corto de ADN monocatenario que contiene la lesión. El ADN monocatenario intacto permanece y la ADN polimerasa lo utiliza como plantilla para sintetizar una secuencia complementaria corta . La ligadura final para completar NER y formar un ADN bicatenario se lleva a cabo mediante ADN ligasa . NER se puede dividir en dos subvías: NER genómica global (GG-NER o GGR) y NER acoplada a transcripción (TC-NER o TCR). Las dos subvías difieren en cómo reconocen el daño del ADN, pero comparten el mismo proceso de incisión, reparación y ligadura de la lesión.
La importancia de NER se evidencia en las graves enfermedades humanas que resultan de mutaciones genéticas congénitas de las proteínas NER. El xeroderma pigmentoso y el síndrome de Cockayne son dos ejemplos de enfermedades asociadas a NER.
La reparación por escisión de nucleótidos es más compleja en eucariotas que en procariotas , pero el principio general es similar. Hay nueve proteínas principales involucradas en NER en células de mamíferos. Las deficiencias de determinadas proteínas provocan enfermedades; Los nombres de las proteínas están asociados con la enfermedad. XPA , XPB , XPC , XPD, XPE , XPF y XPG derivan del eroderma pigmentoso y CSA y CSB representan proteínas vinculadas al síndrome de Cockayne. Además, las proteínas ERCC1 , RPA , RAD23A , RAD23B y otras también participan en la reparación por escisión de nucleótidos. A continuación se encuentra una lista más completa de proteínas involucradas en NER.
La reparación por escisión de nucleótidos eucariotas se puede dividir en dos subvías: NER genómico global (GG-NER) y NER acoplado a transcripción (TC-NER). Tres conjuntos diferentes de proteínas participan en el reconocimiento del daño del ADN en cada subvía. Después del reconocimiento del daño, las tres subvías convergen para los pasos de incisión dual, reparación y ligadura.
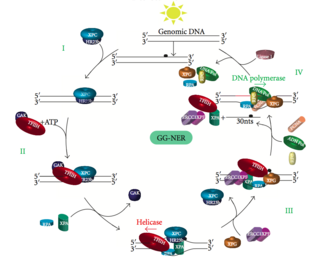
La NER genómica global repara el daño en cadenas de ADN transcritas y no transcritas en genes activos e inactivos en todo el genoma. Este proceso no depende de la transcripción. Esta vía emplea varias proteínas de "detección de daños", incluidos los complejos de unión de daños al ADN (DDB) y XPC-Rad23B que escanean constantemente el genoma y reconocen las distorsiones de la hélice: el complejo XPC -Rad23B es responsable del reconocimiento de la distorsión, mientras que DDB1 y DDB2 ( XPE ) también puede reconocer algunos tipos de daños causados por la luz ultravioleta. Además, XPA desempeña una función de reconocimiento de daños que aún no está bien definida. Tras la identificación de un sitio dañado, se reclutan proteínas de reparación posteriores en el ADN dañado para verificar la presencia de daño en el ADN, se extrae el ADN dañado que rodea la lesión y luego se rellena el parche de reparación.
Las mutaciones en la maquinaria GG-NER son responsables de múltiples trastornos genéticos que incluyen:
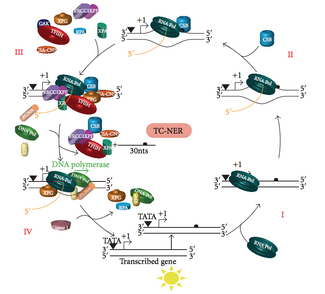
En un momento dado, la mayor parte del genoma de un organismo no se encuentra en proceso de transcripción; Existe una diferencia en la eficiencia de NER entre las regiones transcripcionalmente silenciosas y transcripcionalmente activas del genoma. Para muchos tipos de lesiones, NER repara las hebras transcritas de genes transcripcionalmente activos más rápido que las hebras no transcritas y el ADN transcripcionalmente silencioso.
TC-NER y GG-NER difieren sólo en los pasos iniciales del reconocimiento de daños en el ADN. La principal diferencia entre TC-NER y GG-NER es que TC-NER no requiere proteínas XPC o DDB para el reconocimiento de distorsiones en células de mamíferos. En cambio, TC-NER se inicia cuando la ARN polimerasa se detiene en una lesión en el ADN: la ARN polimerasa bloqueada sirve como una señal de reconocimiento de daños, que reemplaza la necesidad de las propiedades de reconocimiento de distorsión de los complejos XPC-RAD23B y DDB. Las proteínas CS (CSA y CSB) se unen a algunos tipos de daños en el ADN en lugar de XPC-Rad23B.
Otros mecanismos de reparación son posibles pero menos precisos y eficientes.
TC-NER se inicia cuando la ARN polimerasa se detiene en una lesión en el ADN, tras lo cual los complejos de proteínas ayudan a hacer retroceder la polimerasa. Las mutaciones en la maquinaria TC-NER son responsables de múltiples trastornos genéticos que incluyen:
El factor de transcripción II H (TFIIH) es la enzima clave implicada en la escisión dual. TFIIH y XPG se reclutan primero en el sitio del daño del ADN (XPG estabiliza TFIIH). Las subunidades TFIIH de XPD y XPB actúan como una helicasa 5'-3' y 3'-5', respectivamente: ayudan a desenrollar el ADN y generar una unión entre el ADN bicatenario y monocatenario alrededor de la burbuja de transcripción . Además de estabilizar TFIIH, XPG también tiene actividad endonucleasa ; corta el daño del ADN en el lado 3' mientras que la proteína heterodimérica XPF - ERCC1 corta en el lado 5'. La incisión dual conduce a la eliminación de un ssDNA con una brecha de una sola hebra de 25 a 30 nucleótidos. Los oligonucleótidos pequeños, extirpados y que contienen daños en el ADN (sedDNA), se liberan inicialmente del dúplex en complejo con TFIIH, pero luego se disocian de una manera dependiente de ATP y se unen a la proteína de replicación A (RPA). La inhibición de la síntesis y ligadura del ADN que llena los huecos da como resultado una acumulación de ADN sed unidos a RPA en la célula.
La proteína de replicación A (RPA) y XPA son las dos últimas proteínas asociadas con el principal complejo de reparación de NER. Estas dos proteínas están presentes antes de la unión de TFIIH, ya que participan en la verificación del daño del ADN. También pueden proteger el ADN monocatenario. Después de la verificación, se realiza la incisión del lado 5' y comienza la reparación del ADN antes de la incisión del lado 3'. Esto ayuda a reducir el ADN monocatenario expuesto durante el proceso de reparación.
El factor de replicación C ( RFC ) carga el antígeno nuclear de células en proliferación (PCNA) en la cadena de ADN. Esto permite que las ADN polimerasas implicadas en la reparación (δ, ε y/o κ) copie la cadena no dañada mediante translocación. La ADN ligasa I y la endonucleasa Flap 1 o el complejo Ligasa-III-XRCC1 sellan las muescas para completar NER.

El proceso de reparación por escisión de nucleótidos está controlado en Escherichia coli por el complejo enzimático endonucleasa UvrABC , que consta de cuatro proteínas Uvr: UvrA, UvrB, UvrC y ADN helicasa II (a veces también conocida como UvrD en este complejo). En primer lugar, un complejo UvrA-UvrB escanea el ADN y la subunidad UvrA reconoce distorsiones en la hélice causadas, por ejemplo, por dímeros de pirimidina . Cuando el complejo reconoce tal distorsión, la subunidad UvrA sale y entra una proteína UvrC y se une al monómero UvrB y, por lo tanto, forma un nuevo dímero UvrBC . UvrB escinde un enlace fosfodiéster 4 nucleótidos aguas abajo del daño del ADN, y UvrC escinde un enlace fosfodiéster 8 nucleótidos aguas arriba del daño del ADN y creó un segmento extirpado de 12 nucleótidos. Luego entra la ADN helicasa II (a veces llamada UvrD) y elimina el segmento extirpado rompiendo activamente los enlaces de hidrógeno entre las bases complementarias. A continuación, el hueco resultante se rellena con ADN polimerasa I y ADN ligasa. El proceso de escisión básico es muy similar en las células superiores, pero estas células suelen implicar muchas más proteínas; E. coli es un ejemplo sencillo. [5]
TC-NER también existe en bacterias y está mediado por la proteína TRCF (Mfd) . TRCF es una ATPasa SF2 que utiliza la hidrólisis de ATP para translocar el ADNds aguas arriba de la burbuja de transcripción y translocar hacia adelante la ARN polimerasa, iniciando así la disociación del complejo de elongación ternario de la ARN polimerasa. TRCF también recluta la maquinaria de reparación por escisión de nucleótidos Uvr(A)BC mediante interacción física directa con la subunidad UvrA.
Aunque los estudios históricos han mostrado resultados inconsistentes, la variación genética o la mutación de los genes de reparación por escisión de nucleótidos pueden afectar el riesgo de cáncer al afectar la eficacia de la reparación. Los polimorfismos de un solo nucleótido (SNP) y los SNP codificantes no sinónimos (nsSNP) están presentes en niveles muy bajos (>1%) en la población humana. [7] Si se localizan en genes NER o secuencias reguladoras, dichas mutaciones pueden afectar negativamente la capacidad de reparación del ADN, lo que resulta en una mayor probabilidad de desarrollo de cáncer. Si bien no se ha caracterizado el impacto funcional de todos los polimorfismos, algunos polimorfismos en los genes de reparación del ADN o sus secuencias reguladoras inducen cambios fenotípicos y están involucrados en el desarrollo del cáncer. [8] Un estudio de casos de cáncer de pulmón encontró una asociación modesta entre los polimorfismos SNP específicos de NER y el riesgo de cáncer de pulmón. [9] Los resultados indican que algunas variaciones polimórficas hereditarias en los genes NER pueden provocar una predisposición al cáncer de pulmón y, potencialmente, a otros estados cancerosos.
Dos genes importantes en la vía NER para los cuales el polimorfismo ha mostrado un impacto funcional y fenotípico son los genes XPD y XPC . [10] XPD, también conocido como ERCC2, sirve para abrir el ADN alrededor del sitio del daño durante NER, además de otras actividades transcripcionales. Los estudios han demostrado que los polimorfismos en el exón 10 (G>A)(Asp312Asn) y el exón 23 (A>T)(Lys751Gln) están relacionados con la predisposición genética a varios tipos de cáncer. [11] [12] El gen XPC es responsable de una proteína que reconoce el ADN durante la primera parte de la vía NER. Este gen puede tener polimorfismos en el intrón 9 y los SNP en el exón 15 que también se han correlacionado con el riesgo de cáncer. La investigación ha demostrado que un polimorfismo de inserción/deleción poli (AT) bialélico en el intrón 9 de XPC se asocia con un mayor riesgo de cáncer de piel, mama y próstata, [12] [13] [14] especialmente en las poblaciones del norte de la India.
El estudio de un cáncer hereditario, el xeroderma pigmentoso, ha ayudado a identificar varios genes que codifican proteínas en la vía NER, dos de los cuales son XPC y XPD. XP es causado por una deficiencia homocigótica en la reparación del daño del ADN por rayos UV (GG-NER), que aumenta 1000 veces el riesgo de cáncer de piel de los pacientes. En pacientes heterocigotos, el riesgo de cáncer es esporádico, pero puede predecirse basándose en la evaluación analítica de polimorfismos en genes de reparación del ADN relacionados con XP purificados a partir de linfocitos . [15] En un estudio sobre las tasas de recaída de cánceres colorrectales de alto riesgo en estadios II y III, el polimorfismo XPD (ERCC2) 2251A>C se correlacionó significativamente con la recaída temprana después del tratamiento quimioterapéutico. [16] Los estudios han indicado que los efectos de los genes NER polimórficos son aditivos, y cuanto mayor es la frecuencia de variantes, mayor es el riesgo de cáncer. [15] [16] [17]
En humanos y ratones, la mutación de la línea germinal en los genes empleados en NER causa características de envejecimiento prematuro. Estos genes y sus proteínas correspondientes incluyen ERCC1 ( ERCC1 ), ERCC2 (XPD), ERCC3 ( XPB ), ERCC4 (XPF), ERCC5 (XPG), ERCC6 (CSB) y ERCC8 (CSA).
Los ratones mutantes ERCC1 con deficiencia de reparación del ADN muestran características de envejecimiento acelerado y tienen una vida útil limitada. [18] El envejecimiento acelerado en el mutante involucra numerosos órganos.
Las mutaciones en el gen ERCC2 (XPD) pueden provocar varios síndromes, ya sea xeroderma pigmentoso (XP), tricotiodistrofia (TTD) o una combinación de XP y TTD (XPTTD), o una combinación de XP y síndrome de Cockayne (XPCS). [19] TTD y CS muestran características de envejecimiento prematuro. Estas características pueden incluir sordera neurosensorial , degeneración de la retina, hipometilación de la sustancia blanca, calcificación del sistema nervioso central, estatura reducida y caquexia (pérdida de tejido adiposo subcutáneo). [19] [20] Los fibroblastos XPCS y TTD de humanos y ratones mutantes ERCC2 (XPD) muestran evidencia de reparación defectuosa de los daños oxidativos del ADN que pueden ser la base de los síntomas progeroides segmentarios (envejecimiento prematuro) [21] (consulte la teoría del envejecimiento del daño al ADN ). .
Las mutaciones en el gen ERCC3 (XPB) pueden provocar, en humanos, xeroderma pigmentoso (XP) o XP combinado con el síndrome de Cockayne (XPCS). [22]
La deficiencia de ERCC4 (XPF) en humanos da como resultado una variedad de condiciones, incluido el envejecimiento acelerado. [23]
En los seres humanos, los defectos mutacionales en el gen ERCC5 (XPG) pueden causar la afección propensa al cáncer xeroderma pigmentoso (XP) por sí sola o en combinación con el grave trastorno del desarrollo neurológico, el síndrome de Cockayne (CS) o el síndrome cerebro-óculo-facio-facio letal infantil. síndrome esquelético. [24] Un modelo de ratón mutante ERCC5 (XPG) presenta características de envejecimiento prematuro que incluyen caquexia y osteoporosis con fenotipos degenerativos pronunciados tanto en el hígado como en el cerebro. [24] Estos ratones mutantes desarrollan un fenotipo degenerativo de envejecimiento prematuro multisistémico que parece fortalecer el vínculo entre el daño del ADN y el envejecimiento . [24] (ver Teoría del envejecimiento del daño al ADN ).
El síndrome de Cockayne (SC) surge de mutaciones de la línea germinal en cualquiera de los dos genes ERCC8 (CSA) o ERCC6 (CSB). Las mutaciones ERCC8 (CSA) generalmente dan lugar a una forma más moderada de CS que las mutaciones ERCC6 (CSB). [25] Las mutaciones en el gen CSA representan aproximadamente el 20% de los casos de CS. [26] Las personas con CSA y CSB se caracterizan por un crecimiento posnatal severo y retraso mental y un envejecimiento acelerado que conduce a una muerte prematura a la edad de 12 a 16 años. [27]
Según lo revisado por Gorbunova et al., [28] los estudios de NER en diferentes células y tejidos de individuos jóvenes y viejos con frecuencia han mostrado una disminución en la capacidad de NER con el aumento de la edad. Esta disminución puede deberse a niveles constitutivos reducidos de proteínas empleadas en la vía NER. [29]