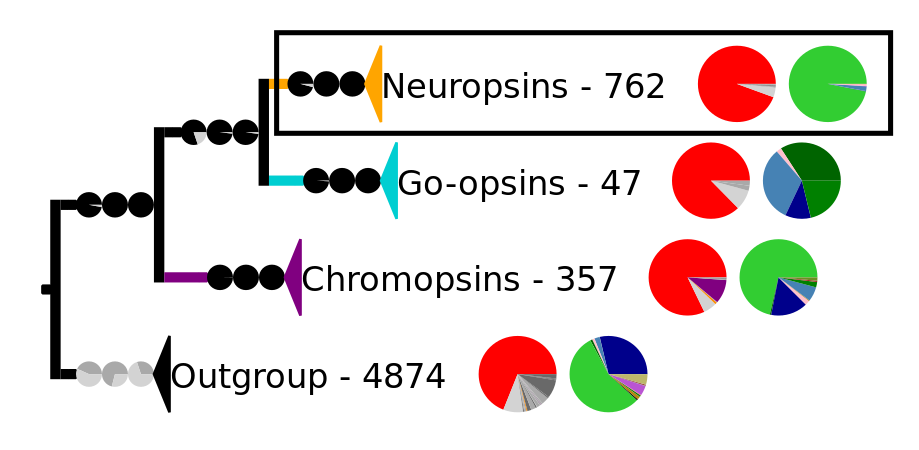Gen codificador de proteínas en la especie Homo sapiens.
La opsina-5 , también conocida como receptor 136 acoplado a proteína G o neuropsina es una proteína que en los humanos está codificada por el gen OPN5 . "La opsina-5 es un miembro de la subfamilia de opsinas de los receptores acoplados a proteína G" . [5] [6] [7] Es una proteína fotorreceptora sensible a la luz ultravioleta (UV) . El gen OPN5 se descubrió en genomas humanos y de ratón y su expresión de ARNm también se encontró en tejidos neurales. La neuropsina es biestable a 0 °C y activa una vía mediada por Gi de proteína G heterotrimérica sensible a los rayos UV en tejidos de mamíferos y aves. [8] [9]
Función
La neuropsina humana se expresa en el ojo, el cerebro, los testículos y la médula espinal. La neuropsina pertenece a la subfamilia de siete exones de genes de opsina de mamíferos que incluye la peropsina (RRH) y el receptor acoplado a proteína G de la retina (RGR). La neuropsina tiene diferentes isoformas creadas mediante empalme alternativo. [7]
Fotoquímica
Cuando se reconstituye con 11-cis-retinal , las neuropsinas humanas y de ratón se absorben al máximo a 380 nm. Cuando se iluminan, estas neuropsinas se convierten en fotoproductos que absorben el azul (470 nm), que son estables en la oscuridad. Los fotoproductos vuelven a la forma absorbente de rayos UV cuando se iluminan con luz naranja (> 520 nm). [8]
Distribución de especies
Las neuropsinas se conocen en equinodermos , [10] anélidos , artrópodos , braquiópodos , tardígrados, moluscos y la mayoría se conocen en cráneos . [11] Los craneados son el taxón que contiene a los mamíferos y con ellos a los humanos. Sin embargo, los ortólogos de neuropsina sólo se han verificado experimentalmente en un pequeño número de animales, entre ellos el ser humano , el ratón ( Mus musculus ), [5] el pollo ( Gallus gallus domesticus ), [9] [12] la codorniz japonesa ( Coturnix japonica ), [13] la estrella quebradiza europea Amphiura filiformis (emparentada con las estrellas de mar), [10] el oso de agua tardígrado ( Hypsibius dujardini ), [14] y el renacuajo de Xenopus laevis . [15]
Las búsquedas en bases de datos de secuencias genéticas disponibles públicamente han encontrado supuestos ortólogos de neuropsina en las dos ramas principales de Bilateria : protostomos y deuterostomos . Entre los protóstomos, se han encontrado supuestas neuropsinas en los moluscos lapa del búho ( Lottia gigantea ) (una especie de caracol de mar) y ostra del Pacífico ( Crassostrea gigas ), en la pulga de agua ( Daphnia pulex ) (un artrópodo ) y en el gusano anélido . Capitella teleta . [14]
Filogenia
Las neuropsinas son uno de los tres subgrupos de las tetraopsinas (también conocidas como RGR/Go o opsinas del grupo 4). Los otros grupos son las cromopsinas y las Go-opsinas. Las tetraopsinas son uno de los cinco grupos principales de opsinas animales , también conocidas como opsinas tipo 2). Los otros grupos son las opsinas ciliares (c-opsinas, cilopsinas), las opsinas rabdoméricas (r-opsinas, rabopsinas), las xenopsinas y las nesopsinas. Cuatro de estos subclados se encuentran en Bilateria (todos menos las nesopsinas). [11] [16] Sin embargo, los clados bilaterales constituyen un taxón parafilético sin las opsinas de los cnidarios . [11] [16] [17] [18]
La relación filogenética de las neuropsinas con las otras opsinas.
Reconstrucción filogenética de las opsinas. El grupo externo contiene otros
receptores acoplados a proteína G. El marco resalta las tetraopsinas, que se amplían en la siguiente imagen.
Reconstrucción filogenética de las tetraopsinas. El grupo externo contiene otros receptores acoplados a proteína G, incluidas las otras opsinas. El marco resalta las neuropsinas, que se amplían en la siguiente imagen.
En la filogenia anterior, cada clado contiene secuencias de opsinas y otros receptores acoplados a proteína G. El número de secuencias y dos gráficos circulares se muestran al lado del clado. El primer gráfico circular muestra el porcentaje de un determinado aminoácido en la posición de las secuencias correspondientes a la posición 296 en la rodopsina bovina. Los aminoácidos están codificados por colores. Los colores son rojo para la lisina (K), morado para el ácido glutámico (E), gris oscuro y medio para otros aminoácidos y gris claro para las secuencias que no tienen datos en esa posición. El segundo gráfico circular muestra la composición taxonómica de cada clado: el verde representa los cráneos , el verde oscuro los cefalocordados , el verde medio los equinodermos , el rosa pálido los anélidos , el azul oscuro los artrópodos , el azul claro los moluscos y el morado los cnidarios . Las ramas a los clados tienen gráficos circulares, que proporcionan valores de soporte para las ramas. Los valores son de derecha a izquierda SH-aLRT/aBayes/UFBoot. Las ramas se consideran apoyadas cuando SH-aLRT ≥ 80%, aBayes ≥ 0,95 y UFBoot ≥ 95%. Si un valor de soporte está por encima de su umbral, el gráfico circular es negro; de lo contrario, es gris. [11]
Referencias
- ^ abc GRCh38: Ensembl lanzamiento 89: ENSG00000124818 - Ensembl , mayo de 2017
- ^ abc GRCm38: Ensembl lanzamiento 89: ENSMUSG00000043972 - Ensembl , mayo de 2017
- ^ "Referencia humana de PubMed:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ "Referencia de PubMed del ratón:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ ab Tarttelin EE, Bellingham J, Hankins MW, Foster RG, Lucas RJ (noviembre de 2003). "Neuropsina (Opn5): una nueva opsina identificada en tejido neural de mamíferos". Cartas FEBS . 554 (3): 410–6. doi : 10.1016/S0014-5793(03)01212-2 . PMID 14623103. S2CID 9577067.
- ^ Fredriksson R, Höglund PJ, Gloriam DE, Lagerström MC, Schiöth HB (noviembre de 2003). "Siete receptores acoplados a proteína rodopsina G humana conservados evolutivamente que carecen de parientes cercanos". Cartas FEBS . 554 (3): 381–8. doi : 10.1016/S0014-5793(03)01196-7 . PMID 14623098. S2CID 11563502.
- ^ ab "Entrez Gene: OPN5 opsina 5".
- ^ ab Kojima D, Mori S, Torii M, Wada A, Morishita R, Fukada Y (2011). "Proteína fotorreceptora OPN5 sensible a los rayos UV en humanos y ratones". MÁS UNO . 6 (10): e26388. Código Bib : 2011PLoSO...626388K. doi : 10.1371/journal.pone.0026388 . PMC 3197025 . PMID 22043319.
- ^ ab Yamashita T, Ohuchi H, Tomonari S, Ikeda K, Sakai K, Shichida Y (diciembre de 2010). "Opn5 es un pigmento biestable sensible a los rayos UV que se acopla con el subtipo Gi de proteína G". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 107 (51): 22084–22089. Código Bib : 2010PNAS..10722084Y. doi : 10.1073/pnas.1012498107 . PMC 3009823 . PMID 21135214.
- ^ ab Delroisse J, Ullrich-Lüter E, Ortega-Martinez O, Dupont S, Arnone MI, Mallefet J, Flammang P (2014). "Alta diversidad de opsinas en una estrella frágil infaunal no visual". Genómica BMC . 15 (1): 1035. doi : 10.1186/1471-2164-15-1035 . PMC 4289182 . PMID 25429842.
- ^ abcd Gühmann M, Porter ML, Bok MJ (agosto de 2022). "Las gluopsinas: opsinas sin lisina de unión a la retina". Células . 11 (15): 2441. doi : 10.3390/celdas11152441 . PMC 9368030 . PMID 35954284.
 El material fue copiado y adaptado de esta fuente, que está disponible bajo una licencia internacional Creative Commons Attribution 4.0.
El material fue copiado y adaptado de esta fuente, que está disponible bajo una licencia internacional Creative Commons Attribution 4.0. - ^ Tomonari S, Migita K, Takagi A, Noji S, Ohuchi H (julio de 2008). "Patrones de expresión de los genes relacionados con la opsina 5 en la retina de pollo en desarrollo". Dinámica del desarrollo . 237 (7): 1910–22. doi : 10.1002/dvdy.21611 . PMID 18570255. S2CID 42113764.
- ^ Nakane Y, Ikegami K, Ono H, Yamamoto N, Yoshida S, Hirunagi K, Ebihara S, Kubo Y, Yoshimura T (agosto de 2010). "Una opsina de tejido neural de mamíferos (Opsina 5) es un fotorreceptor cerebral profundo en las aves". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 107 (34): 15264–8. Código bibliográfico : 2010PNAS..10715264N. doi : 10.1073/pnas.1006393107 . PMC 2930557 . PMID 20679218.
- ^ ab Hering L, Mayer G (septiembre de 2014). "El análisis del repertorio de opsina en el tardígrado Hypsibius dujardini proporciona información sobre la evolución de los genes de opsina en panarthropoda". Biología y evolución del genoma . 6 (9): 2380–91. doi : 10.1093/gbe/evu193. PMC 4202329 . PMID 25193307.
- ^ Currie SP, Doherty GH, Sillar KT (mayo de 2016). "La fotorrecepción profunda del cerebro vincula la detección de luminancia con la salida del motor en los renacuajos de ranas Xenopus". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 113 (21): 6053–6058. Código Bib : 2016PNAS..113.6053C. doi : 10.1073/pnas.1515516113 . PMC 4889350 . PMID 27166423.
- ^ ab Ramirez MD, Pairett AN, Pankey MS, Serb JM, Speiser DI, Swafford AJ, Oakley TH (26 de octubre de 2016). "El último ancestro común de la mayoría de los animales bilaterales poseía al menos 9 opsinas". Biología y evolución del genoma : evw248. doi : 10.1093/gbe/evw248 . PMC 5521729 . PMID 27797948.
- ^ Porter ML, Blasic JR, Bok MJ, Cameron EG, Pringle T, Cronin TW, Robinson PR (enero de 2012). "Arrojando nueva luz sobre la evolución de las opsinas". Actas. Ciencias Biologicas . 279 (1726): 3–14. doi :10.1098/rspb.2011.1819. PMC 3223661 . PMID 22012981.
- ^ Liegertová M, Pergner J, Kozmiková I, Fabian P, Pombinho AR, Strnad H, et al. (Julio de 2015). "El genoma de cubozoo ilumina la diversificación funcional de las opsinas y la evolución de los fotorreceptores". Informes científicos . 5 : 11885. Código Bib : 2015NatSR...511885L. doi :10.1038/srep11885. PMC 5155618 . PMID 26154478.
Otras lecturas
- Terakita A (2005). "Las opsinas". Biología del genoma . 6 (3): 213. doi : 10.1186/gb-2005-6-3-213 . PMC 1088937 . PMID 15774036.
- Vassilatis DK, Hohmann JG, Zeng H, Li F, Ranchalis JE, Mortrud MT, Brown A, Rodriguez SS, Weller JR, Wright AC, Bergmann JE, Gaitanaris GA (abril de 2003). "Los repertorios de receptores acoplados a proteína G de humanos y ratones". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 100 (8): 4903–8. Código Bib : 2003PNAS..100.4903V. doi : 10.1073/pnas.0230374100 . PMC 153653 . PMID 12679517.
Este artículo incorpora texto de la Biblioteca Nacional de Medicina de Estados Unidos , que se encuentra en el dominio público .