
El nacimiento de genes de novo es el proceso por el cual nuevos genes evolucionan a partir de ADN no codificante . [1] [3] Los genes de novo representan un subconjunto de genes nuevos y pueden codificar proteínas o, en cambio, actuar como genes de ARN. [4] Los procesos que gobiernan el nacimiento de genes de novo no se comprenden bien, aunque existen varios modelos que describen posibles mecanismos por los cuales puede ocurrir el nacimiento de genes de novo .
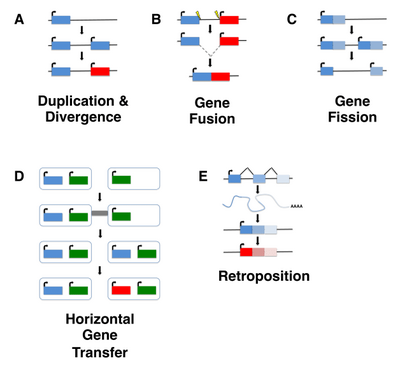
Aunque el nacimiento de genes de novo puede haber ocurrido en cualquier punto de la historia evolutiva de un organismo, los eventos de nacimiento de genes de novo antiguos son difíciles de detectar. La mayoría de los estudios de genes de novo hasta la fecha se han centrado en genes jóvenes, típicamente genes taxonómicamente restringidos (TRG) que están presentes en una sola especie o linaje, incluidos los llamados genes huérfanos , definidos como genes que carecen de cualquier homólogo identificable. Sin embargo, es importante señalar que no todos los genes huérfanos surgen de novo , y en su lugar pueden surgir a través de mecanismos bastante bien caracterizados, como la duplicación de genes (incluida la retroposición) o la transferencia horizontal de genes seguida de divergencia de secuencias, o por fisión/fusión de genes . [5] [6]
Aunque alguna vez se consideró que el nacimiento de genes de novo era un suceso altamente improbable, [7] ahora se han descrito varios ejemplos inequívocos, [8] y algunos investigadores especulan que el nacimiento de genes de novo podría desempeñar un papel importante en la innovación evolutiva, la especificación morfológica y la adaptación, [9] [10] probablemente promovido por su bajo nivel de pleiotropía .
Ya en la década de 1930, JBS Haldane y otros sugirieron que las copias de genes existentes pueden dar lugar a nuevos genes con funciones novedosas. [6] En 1970, Susumu Ohno publicó el texto seminal Evolution by Gene Duplication ( Evolución por duplicación de genes) . [11] Durante algún tiempo después, la opinión consensuada fue que prácticamente todos los genes se derivaban de genes ancestrales, [12] y François Jacob comentó en un ensayo de 1977 que "la probabilidad de que una proteína funcional aparezca de novo por asociación aleatoria de aminoácidos es prácticamente cero". [7]
Sin embargo, en el mismo año, Pierre-Paul Grassé acuñó el término " sobreimpresión " para describir la aparición de genes a través de la expresión de marcos de lectura abiertos alternativos (ORF) que se superponen a genes preexistentes. [13] Estos nuevos ORF pueden estar fuera de marco con el gen preexistente o ser antisentido con respecto a él. También pueden estar en marco con el ORF existente, creando una versión truncada del gen original, o representar extensiones 3' de un ORF existente en un ORF cercano. Los dos primeros tipos de sobreimpresión pueden considerarse un subtipo particular de nacimiento de genes de novo ; aunque se superponen con una región codificante previa del genoma, la secuencia primaria de aminoácidos de la nueva proteína es completamente nueva y se deriva de un marco que previamente no contenía un gen. Los primeros ejemplos de este fenómeno en bacteriófagos se informaron en una serie de estudios entre 1976 y 1978, [14] [15] [16] y desde entonces se han identificado numerosos otros ejemplos en virus, bacterias y varias especies eucariotas. [17] [18] [19] [20] [21] [22]
El fenómeno de la exonización también representa un caso especial de nacimiento de genes de novo , en el que, por ejemplo, secuencias intrónicas a menudo repetitivas adquieren sitios de empalme a través de la mutación, lo que da lugar a exones de novo . Esto se describió por primera vez en 1994 en el contexto de las secuencias Alu encontradas en las regiones codificantes de los ARNm de primates. [23] Curiosamente, dichos exones de novo se encuentran con frecuencia en variantes de empalme menores, lo que puede permitir la “prueba” evolutiva de secuencias nuevas al tiempo que se conserva la funcionalidad de la(s) variante(s) de empalme mayor. [24]
Aun así, algunos pensaban que la mayoría o todas las proteínas eucariotas se construían a partir de un grupo restringido de exones de "tipo inicial". [25] Utilizando los datos de secuencia disponibles en ese momento, una revisión de 1991 estimó que el número de exones eucariotas ancestrales únicos era < 60.000, [25] mientras que en 1992 se publicó un artículo que estimaba que la gran mayoría de las proteínas pertenecían a no más de 1.000 familias. [26] Sin embargo, casi al mismo tiempo, se publicó la secuencia del cromosoma III de la levadura en ciernes Saccharomyces cerevisiae , [27] lo que representó la primera vez que se había secuenciado un cromosoma completo de cualquier organismo eucariota. La secuenciación de todo el genoma nuclear de la levadura se completó a principios de 1996 mediante un esfuerzo internacional masivo y colaborativo. [28] En su revisión del proyecto del genoma de la levadura, Bernard Dujon señaló que la abundancia inesperada de genes que carecían de homólogos conocidos fue quizás el hallazgo más sorprendente de todo el proyecto. [28]
En 2006 y 2007, una serie de estudios proporcionaron posiblemente los primeros ejemplos documentados de nacimiento de genes de novo que no implicaron sobreimpresión. [29] [30] [31] Estos estudios se llevaron a cabo utilizando los transcriptomas de glándulas accesorias de Drosophila yakuba y Drosophila erecta e identificaron 20 genes putativos restringidos al linaje que parecían poco probables de haber resultado de la duplicación genética. [31] Levine y colegas identificaron y confirmaron cinco genes candidatos de novo específicos de Drosophila melanogaster y/o la estrechamente relacionada Drosophila simulans a través de un enfoque riguroso que combinó técnicas bioinformáticas y experimentales. [30]
Desde estos estudios iniciales, muchos grupos han identificado casos específicos de eventos de nacimiento de genes de novo en diversos organismos. [32] El primer gen de novo identificado en levadura, el gen BSC4, fue identificado en S. cerevisiae en 2008. Este gen muestra evidencia de selección purificadora, se expresa tanto a nivel de ARNm como de proteína, y cuando se elimina es sintéticamente letal con otros dos genes de levadura, todo lo cual indica un papel funcional para el producto del gen BSC4 . [33] Históricamente, un argumento en contra de la noción de nacimiento generalizado de genes de novo es la complejidad evolucionada del plegamiento de proteínas. Curiosamente, más tarde se demostró que Bsc4 adopta un estado parcialmente plegado que combina propiedades de plegamiento de proteínas nativas y no nativas. [34] En plantas, el primer gen de novo en ser caracterizado funcionalmente fue QQS , un gen de Arabidopsis thaliana identificado en 2009 que regula el metabolismo del carbono y el nitrógeno. [35] El primer gen de novo caracterizado funcionalmente identificado en ratones, un gen de ARN no codificante, también se describió en 2009. [36] En primates, un análisis informático de 2008 estimó que 15/270 genes huérfanos de primates se habían formado de novo . [37] Un informe de 2009 identificó los primeros tres genes humanos de novo , uno de los cuales es un objetivo terapéutico en la leucemia linfocítica crónica. [38] Desde entonces, una plétora de estudios a nivel del genoma han identificado grandes cantidades de genes huérfanos en muchos organismos, aunque el grado en el que surgieron de novo y el grado en el que pueden considerarse funcionales siguen siendo objeto de debate.
Existen dos enfoques principales para la identificación sistemática de genes nuevos: la filoestratigrafía genómica [39] y los métodos basados en la sintenia . [40] Ambos enfoques se utilizan ampliamente, de forma individual o de forma complementaria.
La filoestratigrafía genómica implica examinar cada gen en una especie focal o de referencia e inferir la presencia o ausencia de homólogos ancestrales mediante el uso de los algoritmos de alineamiento de secuencias BLAST [41] o herramientas relacionadas. A cada gen en la especie focal se le puede asignar una edad (también conocida como “nivel de conservación” o “filoestrato genómico”) que se basa en una filogenia predeterminada, con la edad correspondiente a la especie más distantemente relacionada en la que se detecta un homólogo. [39] Cuando un gen carece de cualquier homólogo detectable fuera de su propio genoma, o parientes cercanos, se dice que es un gen nuevo, taxonómicamente restringido o huérfano.
La filoestratigrafía está limitada por el conjunto de genomas estrechamente relacionados que están disponibles, y los resultados dependen de los criterios de búsqueda BLAST. [42] Además, a menudo es difícil determinar en función de la falta de similitud de secuencia observada si un gen nuevo ha surgido de novo o se ha desviado de un gen ancestral más allá del reconocimiento, por ejemplo, después de un evento de duplicación. Esto fue señalado por un estudio que simuló la evolución de genes de igual edad y encontró que los ortólogos distantes pueden ser indetectables para genes de rápida evolución. [43] Por otro lado, al tener en cuenta los cambios en la tasa de evolución en regiones jóvenes de genes, un enfoque filoestratigráfico fue más preciso para asignar edades de genes en datos simulados. [44] Estudios posteriores que utilizaron evolución simulada encontraron que la filoestratigrafía no pudo detectar un ortólogo en las especies más distantemente relacionadas para el 13,9% de los genes de D. melanogaster y el 11,4% de los genes de S. cerevisiae . [45] [46] Sin embargo, un nuevo análisis de estudios que utilizaron filoestratigrafía en levaduras, moscas de la fruta y humanos encontró que incluso al tener en cuenta dichas tasas de error y excluir los genes difíciles de estratificar de los análisis, las conclusiones cualitativas no se vieron afectadas. [47] El impacto del sesgo filoestratigráfico en los estudios que examinan varias características de los genes de novo sigue siendo objeto de debate.
Los enfoques basados en la sintenia utilizan el orden y el posicionamiento relativo de los genes (u otras características) para identificar los ancestros potenciales de los genes candidatos de novo . [10] [42] Las alineaciones sinténicas están ancladas por "marcadores" conservados. Los genes son el marcador más común para definir los bloques sinténicos, aunque también se utilizan k-meros y exones. [48] [40] La confirmación de que la región sinténica carece de potencial de codificación en especies de grupos externos permite afirmar un origen de novo con mayor confianza. [42] La evidencia más sólida posible para la aparición de novo es la inferencia de la(s) mutación(es) "habilitantes" específicas que crearon el potencial de codificación, típicamente a través del análisis de regiones de secuencia más pequeñas, denominadas regiones microsinténicas, de especies estrechamente relacionadas.
Un desafío en la aplicación de métodos basados en sintenia es que la sintenia puede ser difícil de detectar en escalas de tiempo más largas. Para abordar esto, se han creado varias técnicas de optimización, como el uso de exones agrupados independientemente de su orden específico para definir bloques sinténicos [40] o algoritmos que utilizan regiones genómicas bien conservadas para expandir bloques microsinténicos. [49] También existen dificultades asociadas con la aplicación de enfoques basados en sintenia a ensamblajes genómicos que están fragmentados [50] o en linajes con altas tasas de reordenamientos cromosómicos, como es común en los insectos. [51] Los enfoques basados en sintenia se pueden aplicar a estudios de todo el genoma de genes de novo [37] [38] [52] [53] [54] [55] [56] [57] y representan un área prometedora de desarrollo algorítmico para la datación de nacimiento de genes. Algunos han utilizado enfoques basados en sintenia en combinación con búsquedas de similitud en un intento de desarrollar procesos estandarizados y estrictos [58] que puedan aplicarse a cualquier grupo de genomas en un intento de abordar discrepancias en las diversas listas de genes de novo que se han generado.
Incluso cuando se ha establecido el origen evolutivo de una secuencia codificante particular, todavía no hay consenso sobre lo que constituye un evento genuino de nacimiento de un gen de novo . Una razón para esto es la falta de acuerdo sobre si la totalidad de la secuencia debe ser o no de origen no génico. Para los genes de novo que codifican proteínas , se ha propuesto que los genes de novo se dividan en subtipos en función de la proporción del ORF en cuestión que se derivó de una secuencia no codificante previa. [42] Además, para que se produzca el nacimiento de un gen de novo , la secuencia en cuestión debe ser un gen, lo que ha llevado a cuestionar lo que constituye un gen, con algunos modelos que establecen una dicotomía estricta entre secuencias génicas y no génicas, y otros que proponen un continuo más fluido. [59]
Todas las definiciones de genes están vinculadas a la noción de función, ya que se acepta generalmente que un gen genuino debe codificar un producto funcional, ya sea ARN o proteína. Sin embargo, existen diferentes puntos de vista sobre lo que constituye una función, dependiendo de si una secuencia dada se evalúa utilizando enfoques genéticos, bioquímicos o evolutivos. [42] [60] [61] [62] La ambigüedad del concepto de "función" es especialmente problemática para el campo del nacimiento de genes de novo , donde los objetos de estudio a menudo evolucionan rápidamente. [62] Para abordar estos desafíos, el Modelo de Función de Pittsburgh deconstruye "función" en cinco significados para describir las diferentes propiedades que adquiere un locus que experimenta un nacimiento de genes de novo : expresión, capacidades, interacciones, implicaciones fisiológicas e implicaciones evolutivas. [62]
En general, se acepta que un gen de novo genuino se expresa en al menos algún contexto, [5] lo que permite que opere la selección, y muchos estudios utilizan evidencia de expresión como criterio de inclusión para definir genes de novo . La expresión de secuencias a nivel de ARNm se puede confirmar individualmente a través de técnicas como PCR cuantitativa , o globalmente a través de secuenciación de ARN (RNA-seq) . De manera similar, la expresión a nivel de proteína se puede determinar con alta confianza para proteínas individuales utilizando técnicas como espectrometría de masas o transferencia Western , mientras que el perfil de ribosomas (Ribo-seq) proporciona un estudio global de la traducción en una muestra dada. Idealmente, para confirmar que un gen surgió de novo , también se demostraría una falta de expresión de la región sinténica de especies del grupo externo. [63]
Los enfoques genéticos para detectar un fenotipo específico o un cambio en la aptitud tras la interrupción de una secuencia particular son útiles para inferir una función. [61] También se pueden emplear otros enfoques experimentales, incluidas las pruebas de detección de interacciones proteína-proteína y/o genéticas, para confirmar un efecto biológico para un ORF de novo particular.
Se pueden emplear enfoques evolutivos para inferir la existencia de una función molecular a partir de firmas de selección derivadas computacionalmente. En el caso de los TRG, una firma común de selección es la relación de sustituciones no sinónimas a sinónimas ( relación dN/dS ), calculada a partir de diferentes especies del mismo taxón. De manera similar, en el caso de genes específicos de especies, se pueden utilizar datos de polimorfismo para calcular una relación pN/pS a partir de diferentes cepas o poblaciones de la especie focal. Dado que los genes de novo jóvenes y específicos de especies carecen de una conservación profunda por definición, detectar desviaciones estadísticamente significativas de 1 puede ser difícil sin un número irrealmente grande de cepas/poblaciones secuenciadas. Un ejemplo de esto se puede ver en Mus musculus , donde tres genes de novo muy jóvenes carecen de firmas de selección a pesar de roles fisiológicos bien demostrados. [64] Por esta razón, los enfoques pN/pS a menudo se aplican a grupos de genes candidatos, lo que permite a los investigadores inferir que al menos algunos de ellos están conservados evolutivamente, sin poder especificar cuáles. En cambio, se han empleado otras firmas de selección, como el grado de divergencia de nucleótidos dentro de las regiones sinténicas, la conservación de los límites de ORF o, para los genes codificadores de proteínas, una puntuación de codificación basada en frecuencias de hexámeros de nucleótidos. [65] [66]
Las estimaciones de frecuencia y número de genes de novo en varios linajes varían ampliamente y dependen en gran medida de la metodología. Los estudios pueden identificar genes de novo solo mediante métodos basados en filoestratigrafía/BLAST, o pueden emplear una combinación de técnicas computacionales, y pueden o no evaluar evidencia experimental de expresión y/o papel biológico. [10] Además, los análisis a escala del genoma pueden considerar todos o la mayoría de los ORFs en el genoma, [59] o pueden, en cambio, limitar su análisis a genes previamente anotados.
El linaje de D. melanogaster es ilustrativo de estos diferentes enfoques. Un estudio temprano que utilizó una combinación de búsquedas BLAST realizadas en secuencias de ADNc junto con búsquedas manuales e información de sintenia identificó 72 nuevos genes específicos de D. melanogaster y 59 nuevos genes específicos de tres de las cuatro especies en el complejo de especies de D. melanogaster . Este informe encontró que solo 2/72 (~2,8%) de los nuevos genes específicos de D. melanogaster y 7/59 (~11,9%) de los nuevos genes específicos del complejo de especies se derivaron de novo , [56] y el resto surgió a través de duplicación/retroposición. De manera similar, un análisis de 195 genes jóvenes (<35 millones de años) de D. melanogaster identificados a partir de alineamientos sinténicos encontró que solo 16 habían surgido de novo . [54] Por el contrario, un análisis centrado en datos transcriptómicos de los testículos de seis cepas de D. melanogaster identificó 106 genes fijos y 142 segregantes de novo . [55] Para muchos de estos, se identificaron ORFs ancestrales pero no se expresaron. Un estudio más reciente encontró que hasta el 39 % de los genes huérfanos en el clado de Drosophila pueden haber surgido de novo , ya que se superponen con regiones no codificantes del genoma. [67] Destacando las diferencias entre las comparaciones inter e intra-especies, un estudio en poblaciones naturales de Saccharomyces paradoxus encontró que el número de polipéptidos de novo identificados era más del doble cuando se consideraba la diversidad intra-especies. [68] En primates, un estudio temprano identificó 270 genes huérfanos (únicos de humanos, chimpancés y macacos), de los cuales se pensaba que 15 se habían originado de novo . [37] Informes posteriores identificaron muchos más genes de novo solo en humanos que están respaldados por evidencia transcripcional y proteómica. [57] [69] Los estudios realizados en otros linajes/organismos también han llegado a conclusiones diferentes con respecto al número de genes de novo presentes en cada organismo, así como a los conjuntos específicos de genes identificados. En la siguiente tabla se describe una muestra de estos estudios a gran escala.
En términos generales, sigue habiendo debate sobre si la duplicación y divergencia o el nacimiento de genes de novo representan el mecanismo dominante para la aparición de nuevos genes, [54] [56] [59] [70] [71] [72] en parte porque es probable que los genes de novo surjan y se pierdan con mayor frecuencia que otros genes jóvenes. En un estudio sobre el origen de los genes huérfanos en tres linajes eucariotas diferentes, los autores descubrieron que, en promedio, solo alrededor del 30 % de los genes huérfanos se pueden explicar por la divergencia de secuencias. [72]
Es importante distinguir entre la frecuencia de nacimiento de genes de novo y el número de genes de novo en un linaje dado. Si el nacimiento de genes de novo es frecuente, se podría esperar que los genomas tendieran a crecer en su contenido genético con el tiempo; sin embargo, el contenido genético de los genomas suele ser relativamente estable. [10] Esto implica que un proceso frecuente de muerte genética debe equilibrar el nacimiento de genes de novo y, de hecho, los genes de novo se distinguen por su rápida renovación en relación con los genes establecidos. En apoyo de esta noción, los genes de Drosophila recientemente surgidos tienen muchas más probabilidades de perderse, principalmente a través de la pseudogenización , y los huérfanos más jóvenes se pierden a la tasa más alta; [73] esto a pesar del hecho de que se ha demostrado que algunos genes huérfanos de Drosophila se vuelven esenciales rápidamente. [54] Se observó una tendencia similar de pérdida frecuente entre familias de genes jóvenes en el género de nematodos Pristionchus . [74] De manera similar, un análisis de cinco transcriptomas de mamíferos encontró que la mayoría de los ORFs en ratones eran muy antiguos o específicos de la especie, lo que implica el nacimiento y muerte frecuentes de transcripciones de novo . [71] Una tendencia comparable podría mostrarse mediante análisis adicionales de seis transcriptomas de primates. [69] En poblaciones silvestres de S. paradoxus , los ORFs de novo emergen y se pierden a tasas similares. [68] Sin embargo, sigue habiendo una correlación positiva entre el número de genes específicos de la especie en un genoma y la distancia evolutiva desde su ancestro más reciente. [75] [67] También se encontró una rápida ganancia y pérdida de genes de novo a nivel de población al analizar nueve poblaciones naturales de espinosos de tres espinas. [76] Además del nacimiento y muerte de genes de novo a nivel del ORF, los procesos mutacionales y de otro tipo también someten a los genomas a una constante "renovación transcripcional". Un estudio en ratones descubrió que, si bien todas las regiones del genoma ancestral se transcribieron en algún momento en al menos un descendiente, la porción del genoma bajo transcripción activa en una cepa o subespecie dada está sujeta a cambios rápidos. [77] La renovación transcripcional de los genes de ARN no codificantes es particularmente rápida en comparación con los genes codificantes. [78]
Los genes de novo que han surgido recientemente difieren de los genes establecidos en varias formas. En una amplia gama de especies, se ha informado que los genes jóvenes y/o taxonómicamente restringidos tienen una longitud más corta que los genes establecidos, tienen una carga más positiva, evolucionan más rápido, [88] y se expresan menos. [37] [59] [ 73] [74 ] [ 89] [90] [91] [92] [93] [94] [95] [96] [71 ] [69 ] [67] [76] [ citas excesivas ] Aunque estas tendencias podrían ser el resultado de un sesgo de detección de homología, un nuevo análisis de varios estudios que tuvieron en cuenta este sesgo encontró que las conclusiones cualitativas alcanzadas no se vieron afectadas. [47] Otra característica incluye la tendencia de los genes jóvenes a tener sus aminoácidos hidrofóbicos más agrupados cerca unos de otros a lo largo de la secuencia primaria. [97] [98]
También se ha descubierto que la expresión de genes jóvenes es más específica de tejido o condición que la de genes establecidos. [29] [31] [37] [55] [57] [59] [94] [99] [100] [101] [67] [76] En particular, se observó una expresión relativamente alta de genes de novo en tejidos reproductivos masculinos en Drosophila , espinoso, ratones y humanos, y en el cerebro humano. [57] [102] [67] [76] En animales con sistemas inmunes adaptativos, una mayor expresión en el cerebro y los testículos puede ser una función de la naturaleza inmunológicamente privilegiada de estos tejidos. Un análisis en ratones encontró una expresión específica de transcripciones intergénicas en el timo y el bazo (además del cerebro y los testículos). Se ha propuesto que en vertebrados las transcripciones de novo primero deben expresarse en tejidos que carecen de células inmunes antes de que puedan expresarse en tejidos que tienen vigilancia inmunológica. [101]
En el caso de la evolución de secuencias, los estudios de análisis dN/dS a menudo indican que los genes de novo evolucionan a un ritmo mayor en comparación con otros genes. [103] [88] En el caso de la evolución de la expresión y la evolución estructural, los estudios cuantitativos en diferentes edades evolutivas o ramas filoestratigráficas son muy pocos.
También es interesante comparar las características de los genes de novo recientemente surgidos con el conjunto de ORFs no génicos de los que surgen. El modelado teórico ha demostrado que tales diferencias son el producto tanto de la selección de características que aumentan la probabilidad de funcionalización como de fuerzas evolutivas neutrales que influyen en el recambio alélico. [104] Los experimentos en S. cerevisiae mostraron que los dominios transmembrana predichos estaban fuertemente asociados con efectos beneficiosos de aptitud cuando se sobreexpresaban ORFs jóvenes, pero no cuando se sobreexpresaban ORFs establecidos (más viejos). [105] Los experimentos en E. coli mostraron que los péptidos aleatorios tendían a tener efectos más benignos cuando se enriquecían con aminoácidos que eran pequeños y que promovían el desorden estructural intrínseco. [106]
Las características de los genes de novo pueden depender de la especie o el linaje que se esté examinando. Esto parece ser en parte resultado de la variación del contenido de GC en los genomas y de que los genes jóvenes tienen más similitud con las secuencias no génicas del genoma en el que surgieron que los genes establecidos. [107] Las características de la proteína resultante, como el porcentaje de residuos transmembrana y la frecuencia relativa de varias características estructurales secundarias predichas , muestran una fuerte dependencia de GC en los genes huérfanos, mientras que en los genes más antiguos estas características están influenciadas solo débilmente por el contenido de GC. [107]
La relación entre la edad del gen y la cantidad de desorden estructural intrínseco (ISD) predicho en las proteínas codificadas ha sido objeto de un debate considerable. Se ha afirmado que el ISD también es una característica dependiente del linaje, ejemplificada por el hecho de que en organismos con un contenido de GC relativamente alto, que van desde D. melanogaster hasta el parásito Leishmania major , los genes jóvenes tienen un alto ISD, [108] [109] mientras que en un genoma de bajo GC como la levadura en ciernes, varios estudios han demostrado que los genes jóvenes tienen bajo ISD. [59] [89] [96] [107] Sin embargo, un estudio que excluyó genes jóvenes con evidencia dudosa de funcionalidad, definidos en términos binarios como estar bajo selección para retención de genes, encontró que los genes de levadura jóvenes restantes tienen alto ISD, lo que sugiere que el resultado de la levadura puede deberse a la contaminación del conjunto de genes jóvenes con ORFs que no cumplen con esta definición y, por lo tanto, es más probable que tengan propiedades que reflejen el contenido de GC y otras características no génicas del genoma. [110] Más allá de los huérfanos más jóvenes, este estudio encontró que la ISD tiende a disminuir con el aumento de la edad del gen, y que esto se debe principalmente a la composición de aminoácidos en lugar del contenido de GC. [110] Dentro de escalas de tiempo más cortas, el uso de genes de novo que tienen la mayor validación sugiere que los genes más jóvenes están más desordenados en Lachancea , pero menos desordenados en Saccharomyces . [96] El desorden estructural intrínseco y la propensión a la agregación no mostraron diferencias significativas con la edad en algunos estudios de mamíferos [71] y primates, [69] pero sí en otros estudios de mamíferos. [110] Un estudio grande de la base de datos completa del dominio de proteína Pfam mostró un enriquecimiento del dominio de proteína más joven para aminoácidos promotores de desorden en animales, pero un enriquecimiento sobre la base de la disponibilidad de aminoácidos en plantas. [98]
Un examen de genes de novo en A. thaliana encontró que ambos están hipermetilados y generalmente desprovistos de modificaciones de histonas . [53] De acuerdo con el modelo proto-gen o la contaminación con no genes, los niveles de metilación de los genes de novo fueron intermedios entre los genes establecidos y las regiones intergénicas. Los patrones de metilación de estos genes de novo se heredan de forma estable, y los niveles de metilación fueron más altos, y más similares a los genes establecidos, en genes de novo con capacidad verificada de codificación de proteínas. [53] En el hongo patógeno Magnaporthe oryzae , los genes menos conservados tienden a tener patrones de metilación asociados con bajos niveles de transcripción. [111] Un estudio en levaduras también encontró que los genes de novo se enriquecen en puntos calientes de recombinación , que tienden a ser regiones libres de nucleosomas. [96]
En Pristionchus pacificus , los genes huérfanos con expresión confirmada muestran estados de cromatina que difieren de los de los genes establecidos expresados de manera similar. [95] Los sitios de inicio de genes huérfanos tienen firmas epigenéticas que son características de los potenciadores, en contraste con los genes conservados que exhiben promotores clásicos. [95] Muchos genes huérfanos no expresados están decorados con modificaciones represivas de histonas, mientras que la falta de tales modificaciones facilita la transcripción de un subconjunto expresado de huérfanos, lo que respalda la noción de que la cromatina abierta promueve la formación de genes nuevos. [95]
Las proteínas de novo suelen presentar estructuras secundarias y tridimensionales menos definidas, a menudo sin plegamiento rígido pero con extensas regiones desordenadas. [103] [110] Aún faltan análisis cuantitativos sobre la evolución de los elementos estructurales secundarios y las estructuras terciarias a lo largo del tiempo. Como la estructura suele estar más conservada que la secuencia, la comparación de estructuras entre ortólogos podría proporcionar información más profunda sobre la aparición y evolución de genes de novo y ayudar a confirmar que estos genes son verdaderos genes de novo . [112] Sin embargo, hasta ahora solo se han caracterizado estructural y funcionalmente muy pocas proteínas de novo , especialmente debido a problemas con la purificación de proteínas y la estabilidad posterior. Se han logrado avances utilizando diferentes etiquetas de purificación, tipos de células y chaperonas. [113]
La 'glicoproteína anticongelante' (AFGP) en los bacalaos del Ártico evita que su sangre se congele en aguas árticas. [84] [83] Se ha demostrado que Bsc4, una proteína de novo corta no esencial en la levadura, [33] está construida principalmente por láminas β y tiene un núcleo hidrofóbico. [34] Está asociada a la reparación del ADN en condiciones deficientes de nutrientes. [114] La proteína de novo Goddard de Drosophila se ha caracterizado por primera vez en 2017. Las moscas macho de Knockdown Drosophila melanogaster no pudieron producir esperma. [80] Recientemente, se pudo demostrar que esta falta se debía a un fallo en la individualización de las espermátidas alargadas. Mediante el uso de predicciones filogenómicas y estructurales computacionales, análisis estructurales experimentales y ensayos biológicos celulares, se propuso que la mitad de la estructura de Goddard está desordenada y la otra mitad está compuesta por aminoácidos alfa-helicoidales. Estos análisis también indicaron que los ortólogos de Goddard muestran resultados similares. Por lo tanto, la estructura de Goddard parece haberse conservado principalmente desde su aparición. [81]
Con el desarrollo de tecnologías como RNA-seq y Ribo-seq, ahora se sabe que los genomas eucariotas se transcriben de manera generalizada [115] [116] [117] [118] y se traducen. [119] Muchos ORFs que no están anotados o están anotados como ARN largos no codificantes (lncRNA), se traducen en algún nivel, ya sea de manera específica de una condición o de un tejido. [59] [119] [120] [121] [122] [123] Aunque poco frecuentes, estos eventos de traducción exponen la secuencia no génica a la selección. Esta expresión generalizada forma la base de varios modelos que describen el nacimiento de genes de novo .
Se ha especulado que el paisaje epigenético de los genes de novo en las primeras etapas de formación puede ser particularmente variable entre poblaciones y entre ellas, lo que resulta en una expresión génica variable que permite que los genes jóvenes exploren el "paisaje de expresión". [124] El gen QQS en A. thaliana es un ejemplo de este fenómeno; su expresión está regulada negativamente por la metilación del ADN que, si bien es hereditaria durante varias generaciones, varía ampliamente en sus niveles tanto entre accesiones naturales como dentro de poblaciones silvestres. [124] La epigenética también es en gran medida responsable del entorno transcripcional permisivo en los testículos, particularmente a través de la incorporación a los nucleosomas de variantes de histonas no canónicas que son reemplazadas por protaminas similares a las histonas durante la espermatogénesis. [125]
El análisis de la diversidad potencial de plegamiento muestra que se predice que la mayoría de las secuencias de aminoácidos codificadas por los ORFs intergénicos de S. cerevisiae son plegables. [126] Más importante aún, estas secuencias de aminoácidos con potencial de plegamiento pueden servir como bloques de construcción elementales para genes de novo o integrarse en genes preexistentes. [126]
Para que se produzca el nacimiento de un gen codificador de proteínas de novo , una secuencia no génica debe transcribirse y adquirir un ORF antes de traducirse. Estos eventos podrían ocurrir en cualquier orden, y hay evidencia que respalda tanto un modelo de "ORF primero" como un modelo de "transcripción primero". [5] [127] Un análisis de genes de novo que se segregan en D. melanogaster encontró que las secuencias que se transcriben tenían un potencial de codificación similar a las secuencias ortólogas de líneas que carecen de evidencia de transcripción. [55] Este hallazgo respalda la noción de que pueden existir muchos ORF antes de ser transcritos. El gen de la glucoproteína anticongelante AFGP , que surgió de novo en los bacalaos del Ártico, proporciona un ejemplo más definitivo en el que se demostró que la aparición de novo del ORF precede a la región promotora. [83] Además, los ORF supuestamente no génicos lo suficientemente largos como para codificar péptidos funcionales son numerosos en los genomas eucariotas, y se espera que ocurran con alta frecuencia por casualidad. [55] [59] Mediante el rastreo de la historia evolutiva de las secuencias ORF y la activación de la transcripción de genes humanos de novo , un estudio mostró que algunos ORF estaban listos para conferir importancia biológica desde su nacimiento. [127] Al mismo tiempo, la transcripción de genomas eucariotas es mucho más extensa de lo que se pensaba anteriormente, y hay ejemplos documentados de regiones genómicas que se transcribieron antes de la aparición de un ORF que se convirtió en un gen de novo . [79] La proporción de genes de novo que codifican proteínas es desconocida, pero la aparición de la “transcripción primero” ha llevado a algunos a postular que los genes de novo que codifican proteínas pueden existir primero como intermediarios de genes de ARN. El caso de los ARN bifuncionales, que se traducen y funcionan como genes de ARN, muestra que tal mecanismo es plausible. [128]
Los dos eventos pueden ocurrir simultáneamente cuando el reordenamiento cromosómico es el evento que precipita el nacimiento del gen. [129]
Se han descrito varios modelos teóricos y posibles mecanismos de nacimiento de genes de novo . Los modelos generalmente no son mutuamente excluyentes, y es posible que múltiples mecanismos puedan dar lugar a genes de novo . [42] Un ejemplo es el gen de la proteína anticongelante tipo III, que se origina a partir de un antiguo gen de la sintasa del ácido siálico ( SAS ), en un pez zoárcido antártico.
Un estudio de caso temprano de nacimiento de genes de novo , que identificó cinco genes de novo en D. melanogaster , notó la expresión preferencial de estos genes en los testículos, [30] y se identificaron varios genes de novo adicionales utilizando datos transcriptómicos derivados de los testículos y las glándulas accesorias masculinas de D. yakuba y D. erecta . [29] [31] Esto concuerda con otros estudios que mostraron que hay una rápida evolución de los genes relacionados con la reproducción en una variedad de linajes, [130] [131] [132] lo que sugiere que la selección sexual puede desempeñar un papel clave en la evolución adaptativa y el nacimiento de genes de novo . Un análisis posterior a gran escala de seis cepas de D. melanogaster identificó 248 genes de novo expresados en los testículos , de los cuales ~57% no estaban fijados. [55] Un estudio reciente sobre doce especies de Drosophila identificó además una mayor proporción de genes de novo con expresión sesgada en los testículos en comparación con el proteoma anotado. [67] Se ha sugerido que la gran cantidad de genes de novo con expresión específica masculina identificados en Drosophila probablemente se debe al hecho de que dichos genes se retienen preferentemente en relación con otros genes de novo , por razones que no están del todo claras. [73] Curiosamente, se demostró que dos supuestos genes de novo en Drosophila ( Goddard y Saturn ) eran necesarios para la fertilidad masculina normal. [80] [81] Una pantalla genética de más de 40 supuestos genes de novo con expresión enriquecida en testículos en Drosophila melanogaster reveló que uno de los genes de novo, atlas , era necesario para la condensación adecuada de la cromatina durante las etapas finales de la espermatogénesis en el macho. atlas evolucionó a partir de la fusión de un gen codificador de proteínas que surgió en la base del género Drosophila y un ARN no codificante conservado. [133] El análisis comparativo de los transcriptomas de los testículos y las glándulas accesorias, un tejido somático de los machos que es importante para la fertilidad, de D. melanogaster sugiere que los genes de novo hacen una mayor contribución a la complejidad transcriptómica de los testículos en comparación con las glándulas accesorias. [134] Secuenciación de ARN de una sola célula de D. melanogasterLos testículos revelaron que el patrón de expresión de genes de novo estaba sesgado hacia la espermatogénesis temprana. [135]
En los seres humanos, un estudio que identificó 60 genes de novo específicos de los humanos descubrió que su expresión promedio, medida por RNA-seq, era más alta en los testículos. [57] Otro estudio que examinó los genes específicos de los mamíferos de manera más general también encontró una expresión enriquecida en los testículos. [136] Se cree que la transcripción en los testículos de los mamíferos es particularmente promiscua, debido en parte a la expresión elevada de la maquinaria de transcripción [137] [138] y un entorno de cromatina abierto. [139] Junto con la naturaleza inmunológicamente privilegiada de los testículos, se cree que esta transcripción promiscua crea las condiciones ideales para la expresión de secuencias no génicas requeridas para el nacimiento de genes de novo . La expresión específica de los testículos parece ser una característica general de todos los genes nuevos, ya que un análisis de Drosophila y especies de vertebrados encontró que los genes jóvenes mostraban una expresión sesgada hacia los testículos independientemente de su mecanismo de origen. [99]
El modelo de preadaptación del nacimiento de genes de novo utiliza modelos matemáticos para mostrar que cuando las secuencias que normalmente están ocultas se exponen a una selección débil o protegida, el conjunto resultante de secuencias "crípticas" (es decir, protogenes) se puede purgar de variantes "evidentemente perjudiciales", como las que tienden a conducir a la agregación de proteínas, y así enriquecerse en adaptaciones potenciales en relación con un conjunto de secuencias completamente no expresadas y no purgadas. [140] Esta revelación y purga de secuencias no genéticas deletéreas crípticas es un subproducto de la transcripción y traducción generalizadas de secuencias intergénicas, y se espera que facilite el nacimiento de genes codificadores de proteínas de novo funcionales. [122] Esto se debe a que al eliminar las variantes más perjudiciales, lo que queda es, mediante un proceso de eliminación, más propenso a ser adaptativo de lo esperado a partir de secuencias aleatorias. Utilizando la definición evolutiva de función (es decir, que un gen está por definición bajo selección purificadora contra la pérdida), el modelo de preadaptación supone que “el nacimiento de un gen es una transición repentina a la funcionalidad” [110] que ocurre tan pronto como un ORF adquiere un efecto beneficioso neto. Para evitar ser perjudiciales, se espera que los genes recién nacidos muestren versiones exageradas de las características génicas asociadas con la evitación del daño. Esto contrasta con el modelo de proto-gen, que espera que los genes recién nacidos tengan características intermedias entre los genes antiguos y los no genes. [110]
Las matemáticas del modelo de preadaptación suponen que la distribución de los efectos de la aptitud es bimodal, con nuevas secuencias de mutaciones que tienden a romper algo o a modificarlo, pero rara vez en el medio. [140] [141] Siguiendo esta lógica, las poblaciones pueden desarrollar soluciones locales, en las que la selección opera en cada locus individual y se mantiene una tasa de error relativamente alta, o una solución global con una tasa de error baja que permite la acumulación de secuencias crípticas deletéreas. [140] Se cree que el nacimiento de genes de novo se favorece en poblaciones que desarrollan soluciones locales, ya que la tasa de error relativamente alta dará como resultado un conjunto de variación críptica que se "preadapta" a través de la purga de secuencias deletéreas. Las soluciones locales son más probables en poblaciones con un tamaño de población efectivo alto .
En apoyo del modelo de preadaptación, un análisis de ISD en ratones y levaduras encontró que los genes jóvenes tienen un ISD más alto que los genes viejos, mientras que las secuencias no génicas aleatorias tienden a mostrar los niveles más bajos de ISD. [110] Aunque la tendencia observada puede haber resultado en parte de un subconjunto de genes jóvenes derivados por sobreimpresión, [142] también se observa un ISD más alto en genes jóvenes entre pares de genes virales superpuestos. [143] Con respecto a otras características estructurales predichas, como el contenido de la cadena β y la propensión a la agregación, los péptidos codificados por protogenes son similares a las secuencias no génicas y categóricamente distintos de los genes canónicos. [144]
Este modelo de proto-gen concuerda con el modelo de preadaptación sobre la importancia de la expresión generalizada, y se refiere al conjunto de secuencias expresadas de forma generalizada que no cumplen todas las definiciones de un gen como “proto-genes”. [59] En contraste con el modelo de preadaptación, el modelo de proto-gen sugiere que los genes recién nacidos tienen características intermedias entre los genes antiguos y los no genes. [110] Específicamente, este modelo prevé un proceso más gradual bajo selección desde el estado no génico al génico, rechazando la clasificación binaria de gen y no gen.
En una extensión del modelo de protogenes, se ha propuesto que a medida que los protogenes se vuelven más similares a los genes, su potencial para el cambio adaptativo da paso a efectos seleccionados; por lo tanto, el impacto previsto de las mutaciones en la aptitud depende del estado evolutivo del ORF. [105] Esta noción está respaldada por el hecho de que la sobreexpresión de ORFs establecidos en S. cerevisiae tiende a ser menos beneficiosa (y más dañina) que la sobreexpresión de ORFs emergentes. [105]
Varias características de los ORF se correlacionan con la edad de los ORF, determinada mediante análisis filoestratigráficos, y los ORF jóvenes tienen propiedades intermedias entre los ORF antiguos y los no genes; esto se ha tomado como evidencia a favor del modelo protogenético, en el que el estado protogenético es un continuo. [59] Esta evidencia ha sido criticada, porque también se esperan las mismas tendencias aparentes bajo un modelo en el que la identidad como gen es binaria. Bajo este modelo, cuando cada grupo de edad contiene una proporción diferente de genes frente a no genes, la paradoja de Simpson puede generar correlaciones en la dirección equivocada. [110]
El modelo de “crecimiento lento y muda” describe un mecanismo potencial de nacimiento de genes de novo , particular para los genes codificadores de proteínas. En este escenario, los ORFs codificadores de proteínas existentes se expanden en sus extremos, especialmente sus extremos 3', lo que lleva a la creación de nuevos dominios N- y C-terminales. [145] [146] [147] [148] [149] Los nuevos dominios C-terminales pueden evolucionar primero bajo selección débil a través de la expresión ocasional a través de la traducción de lectura continua, como en el modelo de preadaptación, y luego expresarse de manera constitutiva a través de una mutación que interrumpe el codón de terminación. [140] [146] Los genes que experimentan una alta lectura continua de la traducción tienden a tener C-terminales intrínsecamente desordenados. [150] Además, los genes existentes a menudo están cerca de secuencias repetitivas que codifican dominios desordenados. Estos nuevos dominios desordenados pueden conferir inicialmente cierta capacidad de unión no específica que se refina gradualmente por selección. Las secuencias que codifican estos nuevos dominios pueden ocasionalmente separarse de su ORF original, lo que conduce o contribuye a la creación de un gen de novo . [146] Curiosamente, un análisis de 32 genomas de insectos encontró que los nuevos dominios (es decir, aquellos exclusivos de los insectos) tienden a evolucionar de manera bastante neutral, con solo unos pocos sitios bajo selección positiva, mientras que sus proteínas hospedantes permanecen bajo selección purificadora, lo que sugiere que los nuevos dominios funcionales emergen gradualmente y algo estocásticamente. [151]
El modelo evolutivo de escape del conflicto adaptativo (EAC) propone una forma posible de fijar la nueva duplicación de genes: el conflicto debido a una función contrastante dentro de un solo gen impulsa la fijación de una nueva duplicación. [152] [153]
El modelo de "barrera de pleiotropía" sugiere que los genes recientemente evolucionados, incluidos los genes de novo y los genes relacionados con la duplicación, podrían facilitar la innovación evolutiva o la evolución de funciones específicas debido a su bajo (o nulo) efecto pleiotrópico , cuando se enfrentan a una nueva fuerza selectiva, según observaciones de datos de genes y enfermedades humanas.
Además de su importancia para el campo de la biología evolutiva, el nacimiento de genes de novo tiene implicaciones para la salud humana. Se ha especulado que los genes nuevos, incluidos los genes de novo , pueden desempeñar un papel descomunal en los rasgos específicos de las especies; [6] [10] [32] [154] sin embargo, muchos genes específicos de las especies carecen de anotación funcional. [136] No obstante, hay evidencia que sugiere que los genes de novo específicos de los humanos están involucrados en enfermedades como el cáncer. NYCM , un gen de novo exclusivo de los humanos y los chimpancés, regula la patogénesis de los neuroblastomas en modelos de ratón, [155] y el PART1 específico de los primates , un gen lncRNA, ha sido identificado como un supresor de tumores y un oncogén en diferentes contextos. [37] [156] [157] Varios otros genes de novo específicos de humanos o primates , incluidos PBOV1 , [158] GR6 , [159] [160] MYEOV , [161] ELFN1-AS1 , [162] y CLLU1 , [38] también están vinculados al cáncer. Algunos incluso han sugerido considerar los genes evolutivamente nuevos expresados específicamente en tumores como su propia clase de elementos genéticos, señalando que muchos de estos genes están bajo selección positiva y pueden ser neofuncionalizados en el contexto de los tumores. [162]
La expresión específica de muchos genes de novo en el cerebro humano [57] también plantea la intrigante posibilidad de que los genes de novo influyan en los rasgos cognitivos humanos. Un ejemplo de ello es FLJ33706 , un gen de novo que se identificó en GWAS y análisis de ligamiento para la adicción a la nicotina y muestra una expresión elevada en los cerebros de pacientes con Alzheimer. [163] En términos generales, la expresión de genes jóvenes específicos de primates se enriquece en el cerebro humano fetal en relación con la expresión de genes igualmente jóvenes en el cerebro del ratón. [164] La mayoría de estos genes jóvenes, varios de los cuales se originaron de novo , se expresan en el neocórtex, que se cree que es responsable de muchos aspectos de la cognición específica humana. Muchos de estos genes jóvenes muestran firmas de selección positiva, y las anotaciones funcionales indican que están involucrados en diversos procesos moleculares, pero están enriquecidos con factores de transcripción. [164]
Además de su papel en los procesos cancerosos, los genes humanos de origen de novo se han visto implicados en el mantenimiento de la pluripotencia [165] y en la función inmunológica. [37] [136] [166] La expresión preferencial de genes de novo en los testículos también sugiere un papel en la reproducción. Dado que la función de muchos genes humanos de novo sigue sin caracterizarse, parece probable que siga aumentando la apreciación de su contribución a la salud y el desarrollo humanos.
Nota: Para los fines de esta tabla, los genes se definen como genes huérfanos (cuando son específicos de una especie) o TRG (cuando se limitan a un grupo de especies estrechamente relacionadas) cuando no se ha investigado el mecanismo de origen, y como genes de novo cuando se ha inferido el origen de novo , independientemente del método de inferencia. La designación de genes de novo como “candidatos” o “protogenes” refleja el lenguaje utilizado por los autores de los respectivos estudios.
![]() Este artículo fue adaptado de la siguiente fuente bajo una licencia CC BY 4.0 (2019) (informes de los revisores): Stephen Branden Van Oss; Anne-Ruxandra Carvunis (23 de mayo de 2019). "Nacimiento de genes de novo". PLOS Genetics . 15 (5): e1008160. doi : 10.1371/JOURNAL.PGEN.1008160 . ISSN 1553-7390. PMC 6542195 . PMID 31120894. Wikidata Q86320144.
Este artículo fue adaptado de la siguiente fuente bajo una licencia CC BY 4.0 (2019) (informes de los revisores): Stephen Branden Van Oss; Anne-Ruxandra Carvunis (23 de mayo de 2019). "Nacimiento de genes de novo". PLOS Genetics . 15 (5): e1008160. doi : 10.1371/JOURNAL.PGEN.1008160 . ISSN 1553-7390. PMC 6542195 . PMID 31120894. Wikidata Q86320144.