
Los sitios CpG o sitios CG son regiones del ADN donde un nucleótido de citosina es seguido por un nucleótido de guanina en la secuencia lineal de bases a lo largo de su dirección 5' → 3' . Los sitios CpG ocurren con alta frecuencia en regiones genómicas llamadas islas CpG.
Las citosinas en los dinucleótidos CpG se pueden metilar para formar 5-metilcitosinas . Las enzimas que añaden un grupo metilo se denominan metiltransferasas de ADN . En los mamíferos, entre el 70% y el 80% de las citosinas CpG están metiladas. [1] La metilación de la citosina dentro de un gen puede cambiar su expresión, un mecanismo que forma parte de un campo científico más amplio que estudia la regulación genética y que se denomina epigenética . Las citosinas metiladas a menudo mutan a timinas .
En los seres humanos, aproximadamente el 70% de los promotores ubicados cerca del sitio de inicio de la transcripción de un gen (promotores proximales) contienen una isla CpG. [2] [3]
CpG es la abreviatura de 5'—C—fosfato—G—3' , es decir, citosina y guanina separadas por un solo grupo fosfato ; el fosfato une dos nucleósidos cualesquiera en el ADN. La notación CpG se utiliza para distinguir esta secuencia lineal monocatenaria del apareamiento de bases CG de citosina y guanina para secuencias bicatenarias. Por lo tanto, la notación CpG debe interpretarse como que la citosina es la 5 prima respecto a la base guanina. CpG no debe confundirse con GpC , lo que significa que una guanina va seguida de una citosina en la dirección 5' → 3' de una secuencia monocatenaria.
Desde hace mucho tiempo se ha observado que los dinucleótidos CpG aparecen con una frecuencia mucho menor en la secuencia de genomas de vertebrados de lo que se esperaría debido al azar. Por ejemplo, en el genoma humano, que tiene un contenido de GC del 42 % , [4] se esperaría que apareciera un par de nucleótidos consistente en citosina seguida de guanina . La frecuencia de dinucleótidos CpG en los genomas humanos es inferior a una quinta parte de la frecuencia esperada. [5]
Esta subrepresentación es una consecuencia de la alta tasa de mutación de los sitios CpG metilados: la desaminación espontánea de una citosina metilada da como resultado una timina y las bases desapareadas G:T resultantes a menudo se resuelven incorrectamente en A:T; mientras que la desaminación de la citosina no metilada da como resultado un uracilo , que como base extraña es rápidamente reemplazada por una citosina por el mecanismo de reparación por escisión de bases . La tasa de transición de C a T en los sitios CpG metilados es aproximadamente 10 veces mayor que en los sitios no metilados. [6] [7] [8] [9]
Los dinucleótidos CpG se encuentran frecuentemente en islas CpG (ver la definición de islas CpG, más abajo). Hay 28.890 islas CpG en el genoma humano (50.267 si se incluyen las islas CpG en secuencias repetidas). [10] Esto concuerda con las 28.519 islas CpG encontradas por Venter et al. [11] ya que la secuencia del genoma de Venter et al. no incluía los interiores de elementos repetitivos altamente similares y las regiones repetidas extremadamente densas cerca de los centrómeros. [12] Dado que las islas CpG contienen múltiples secuencias de dinucleótidos CpG, parece haber más de 20 millones de dinucleótidos CpG en el genoma humano.

Las islas CpG (o islas CG) son regiones con una alta frecuencia de sitios CpG. Aunque las definiciones objetivas de las islas CpG son limitadas, la definición formal habitual es una región con al menos 200 pb , un porcentaje de GC mayor del 50% y una relación CpG observada-esperada mayor del 60%. La "relación CpG observada-esperada" se puede derivar donde lo observado se calcula como: y lo esperado como [13] o . [14]
Muchos genes en los genomas de mamíferos tienen islas CpG asociadas con el inicio del gen [15] ( regiones promotoras ). Debido a esto, la presencia de una isla CpG se utiliza para ayudar en la predicción y anotación de genes.
En los genomas de los mamíferos, las islas CpG tienen una longitud de entre 300 y 3000 pares de bases y se han encontrado en aproximadamente el 40 % de los promotores de genes de mamíferos o cerca de ellos. [16] Más del 60 % de los genes humanos y casi todos los genes de mantenimiento tienen sus promotores integrados en islas CpG. [17] Dada la frecuencia de las secuencias de dos nucleótidos de GC, la cantidad de dinucleótidos CpG es mucho menor de lo que se esperaría. [14]
Un estudio de 2002 revisó las reglas de predicción de islas CpG para excluir otras secuencias genómicas ricas en GC, como las repeticiones Alu . Basándose en una búsqueda exhaustiva de las secuencias completas de los cromosomas humanos 21 y 22, se encontró que las regiones de ADN mayores de 500 pb tenían más probabilidades de ser las islas CpG "verdaderas" asociadas con las regiones 5' de los genes si tenían un contenido de GC mayor del 55% y una relación CpG observada-esperada del 65%. [18]
Las islas CpG se caracterizan por un contenido de dinucleótidos CpG de al menos el 60% de lo que sería estadísticamente esperado (~4–6%), mientras que el resto del genoma tiene una frecuencia de CpG mucho menor (~1%), un fenómeno llamado supresión de CG . A diferencia de los sitios CpG en la región codificante de un gen, en la mayoría de los casos los sitios CpG en las islas CpG de los promotores no están metilados si los genes se expresan. Esta observación llevó a la especulación de que la metilación de los sitios CpG en el promotor de un gen puede inhibir la expresión génica. La metilación, junto con la modificación de histonas , es fundamental para la impronta . [19] La mayoría de las diferencias de metilación entre tejidos, o entre muestras normales y cancerosas, ocurren a poca distancia de las islas CpG (en las "costas de las islas CpG") en lugar de en las islas mismas. [20]
Las islas CpG suelen aparecer en el sitio de inicio de la transcripción de los genes o cerca de él, en particular en los genes de mantenimiento , en los vertebrados. [14] La base AC (citosina) seguida inmediatamente de una base G (guanina) (un CpG) es poco común en el ADN de vertebrados porque las citosinas en tal disposición tienden a estar metiladas. Esta metilación ayuda a distinguir la cadena de ADN recién sintetizada de la cadena original, lo que ayuda en las etapas finales de la corrección del ADN después de la duplicación. Sin embargo, con el tiempo, las citosinas metiladas tienden a convertirse en timinas debido a la desaminación espontánea . Hay una enzima especial en los humanos ( Timina-ADN glicosilasa o TDG) que reemplaza específicamente las T de los desajustes T/G. Sin embargo, debido a la rareza de los CpG, se teoriza que no son suficientemente eficaces para prevenir una posible mutación rápida de los dinucleótidos. La existencia de islas CpG suele explicarse por la existencia de fuerzas selectivas para un contenido relativamente alto de CpG, o niveles bajos de metilación en esa zona genómica, tal vez en relación con la regulación de la expresión génica. Un estudio de 2011 demostró que la mayoría de las islas CpG son resultado de fuerzas no selectivas. [21]

En los seres humanos, aproximadamente el 70% de los promotores ubicados cerca del sitio de inicio de la transcripción de un gen (promotores proximales) contienen una isla CpG. [2] [3]
Los elementos promotores distales también contienen frecuentemente islas CpG. Un ejemplo es el gen de reparación de ADN ERCC1 , donde el elemento que contiene la isla CpG se encuentra aproximadamente 5400 nucleótidos antes del sitio de inicio de la transcripción del gen ERCC1 . [22] Las islas CpG también aparecen con frecuencia en los promotores de ARN no codificantes funcionales, como los microARN . [23]
En los seres humanos, la metilación del ADN se produce en la posición 5 del anillo de pirimidina de los residuos de citosina dentro de los sitios CpG para formar 5-metilcitosinas . La presencia de múltiples sitios CpG metilados en las islas CpG de los promotores provoca el silenciamiento estable de los genes. [24] El silenciamiento de un gen puede iniciarse por otros mecanismos, pero a menudo esto es seguido por la metilación de los sitios CpG en la isla CpG del promotor para provocar el silenciamiento estable del gen. [24]
En los cánceres, la pérdida de expresión de los genes ocurre aproximadamente 10 veces más frecuentemente por hipermetilación de las islas CpG promotoras que por mutaciones. Por ejemplo, en un cáncer colorrectal, generalmente hay alrededor de 3 a 6 mutaciones impulsoras y de 33 a 66 mutaciones de tipo autoestopista o pasajero. [25] En contraste, en un estudio de tumores de colon en comparación con la mucosa colónica adyacente de apariencia normal, 1734 islas CpG estaban altamente metiladas en los tumores, mientras que estas islas CpG no estaban metiladas en la mucosa adyacente. [26] La mitad de las islas CpG estaban en promotores de genes codificadores de proteínas anotados, [26] lo que sugiere que alrededor de 867 genes en un tumor de colon han perdido expresión debido a la metilación de las islas CpG. Un estudio independiente encontró un promedio de 1.549 regiones metiladas de forma diferencial (hipermetiladas o hipometiladas) en los genomas de seis cánceres de colon (en comparación con la mucosa adyacente), de las cuales 629 se encontraban en regiones promotoras conocidas de genes. [27] Un tercer estudio encontró más de 2.000 genes metilados de forma diferencial entre cánceres de colon y mucosa adyacente. Utilizando el análisis de enriquecimiento de conjuntos de genes , 569 de 938 conjuntos de genes estaban hipermetilados y 369 estaban hipometilados en los cánceres. [28] La hipometilación de las islas CpG en los promotores da como resultado la sobreexpresión de los genes o conjuntos de genes afectados.
Un estudio de 2012 [29] enumeró 147 genes específicos con promotores hipermetilados asociados al cáncer de colon, junto con la frecuencia con la que se encontraron estas hipermetilaciones en los cánceres de colon. Al menos 10 de esos genes tenían promotores hipermetilados en casi el 100% de los cánceres de colon. También indicaron 11 microARN cuyos promotores estaban hipermetilados en cánceres de colon en frecuencias entre el 50% y el 100% de los cánceres. Los microARN (miARN) son pequeños ARN endógenos que se emparejan con secuencias en ARN mensajeros para dirigir la represión postranscripcional. En promedio, cada microARN reprime varios cientos de genes diana. [30] Por lo tanto, los microARN con promotores hipermetilados pueden estar permitiendo la sobreexpresión de cientos a miles de genes en un cáncer.
La información anterior muestra que, en los cánceres, la hiper/hipometilación del promotor CpG de los genes y de los microARN causa pérdida de expresión (o a veces aumento de la expresión) de muchos más genes que la mutación.
Los genes de reparación del ADN se reprimen con frecuencia en los cánceres debido a la hipermetilación de las islas CpG dentro de sus promotores. En los carcinomas de células escamosas de cabeza y cuello, al menos 15 genes de reparación del ADN tienen promotores frecuentemente hipermetilados; estos genes son XRCC1 , MLH3 , PMS1 , RAD51B, XRCC3 , RAD54B, BRCA1 , SHFM1 , GEN1, FANCE , FAAP20, SPRTN , SETMAR , HUS1 y PER1 . [31] Aproximadamente diecisiete tipos de cáncer son frecuentemente deficientes en uno o más genes de reparación del ADN debido a la hipermetilación de sus promotores. [32] Como ejemplo, la hipermetilación del promotor del gen de reparación del ADN MGMT ocurre en el 93% de los cánceres de vejiga, el 88% de los cánceres de estómago, el 74% de los cánceres de tiroides, el 40%-90% de los cánceres colorrectales y el 50% de los cánceres cerebrales. La hipermetilación del promotor de LIG4 ocurre en el 82% de los cánceres colorrectales. La hipermetilación del promotor de NEIL1 ocurre en el 62% de los cánceres de cabeza y cuello y en el 42% de los cánceres de pulmón de células no pequeñas . La hipermetilación del promotor de ATM ocurre en el 47% de los cánceres de pulmón de células no pequeñas . La hipermetilación del promotor de MLH1 ocurre en el 48% de los carcinomas de células escamosas de cáncer de pulmón de células no pequeñas . La hipermetilación del promotor de FANCB ocurre en el 46% de los cánceres de cabeza y cuello .
Por otra parte, los promotores de dos genes, PARP1 y FEN1 , estaban hipometilados y estos genes estaban sobreexpresados en numerosos cánceres. PARP1 y FEN1 son genes esenciales en la vía de reparación del ADN propensa a errores y mutagénica, la unión de extremos mediada por microhomología . Si esta vía está sobreexpresada, el exceso de mutaciones que causa puede conducir al cáncer. PARP1 está sobreexpresada en leucemias activadas por tirosina quinasa, [33] en neuroblastoma, [34] en tumores testiculares y otros tumores de células germinales, [35] y en sarcoma de Ewing, [36] FEN1 está sobreexpresada en la mayoría de cánceres de mama, [37] próstata, [38] estómago, [39] [40] neuroblastomas, [41] páncreas, [42] y pulmón. [43]
El daño del ADN parece ser la principal causa subyacente del cáncer. [44] [45] Si la reparación precisa del ADN es deficiente, los daños del ADN tienden a acumularse. Este daño excesivo del ADN puede aumentar los errores mutacionales durante la replicación del ADN debido a la síntesis de translesión propensa a errores . El daño excesivo del ADN también puede aumentar las alteraciones epigenéticas debido a errores durante la reparación del ADN. [46] [47] Estas mutaciones y alteraciones epigenéticas pueden dar lugar al cáncer (ver neoplasias malignas ). Por lo tanto, la hiper/hipometilación de las islas CpG en los promotores de los genes de reparación del ADN es probablemente fundamental para la progresión al cáncer.
Dado que la edad tiene un fuerte efecto en los niveles de metilación del ADN en decenas de miles de sitios CpG, se puede definir un reloj biológico altamente preciso (denominado reloj epigenético o edad de metilación del ADN ) en humanos y chimpancés. [48]
Los sitios de dinucleótidos CpG no metilados pueden detectarse mediante el receptor tipo Toll 9 ( TLR 9 ) [49] en células dendríticas plasmocitoides , monocitos , células asesinas naturales (NK) y células B en humanos. Esto se utiliza para detectar infecciones virales intracelulares.
En los mamíferos, las metiltransferasas de ADN (que añaden grupos metilo a las bases de ADN) muestran una preferencia de secuencia por las citosinas dentro de los sitios CpG. [50] En el cerebro del ratón, el 4,2 % de todas las citosinas están metiladas, principalmente en el contexto de los sitios CpG, formando 5mCpG. [51] La mayoría de los sitios 5mCpG hipermetilados aumentan la represión de los genes asociados. [51]
Como analizaron Duke et al., la metilación del ADN neuronal (represión de la expresión de genes particulares) se altera con la actividad neuronal. La metilación del ADN neuronal es necesaria para la plasticidad sináptica ; se modifica con las experiencias; y la metilación y desmetilación activas del ADN son necesarias para la formación y el mantenimiento de la memoria. [52]
En 2016, Halder et al. [53] , utilizando ratones, y en 2017, Duke et al. [52], utilizando ratas, sometieron a los roedores a un condicionamiento de miedo contextual , lo que provocó la formación de una memoria a largo plazo especialmente fuerte. A las 24 horas después del condicionamiento, en la región cerebral del hipocampo de las ratas, la expresión de 1.048 genes se reguló a la baja (generalmente asociada con 5mCpG en los promotores de genes ) y la expresión de 564 genes se reguló al alza (a menudo asociada con la hipometilación de los sitios CpG en los promotores de genes). A las 24 horas después del entrenamiento, el 9,2% de los genes en el genoma de la rata de las neuronas del hipocampo estaban metilados de forma diferencial. Sin embargo, aunque el hipocampo es esencial para aprender nueva información, no almacena información por sí mismo. En los experimentos con ratones de Halder, se observaron 1.206 genes metilados de forma diferencial en el hipocampo una hora después del condicionamiento contextual del miedo, pero estas metilaciones alteradas se revirtieron y no se observaron después de cuatro semanas. En contraste con la ausencia de cambios de metilación de CpG a largo plazo en el hipocampo, se pudo detectar una metilación diferencial sustancial de CpG en las neuronas corticales durante el mantenimiento de la memoria. Hubo 1.223 genes metilados de forma diferencial en la corteza cingulada anterior de ratones cuatro semanas después del condicionamiento contextual del miedo.

En las células somáticas adultas, la metilación del ADN ocurre típicamente en el contexto de dinucleótidos CpG ( sitios CpG ), formando 5-metilcitosina -pG, o 5mCpG. Las especies reactivas de oxígeno (ROS) pueden atacar a la guanina en el sitio del dinucleótido, formando 8-hidroxi-2'-desoxiguanosina (8-OHdG), y dando como resultado un sitio de dinucleótido 5mCp-8-OHdG. La enzima de reparación por escisión de bases OGG1 se dirige a 8-OHdG y se une a la lesión sin escisión inmediata. OGG1, presente en un sitio 5mCp-8-OHdG, recluta a TET1 y TET1 oxida el 5mC adyacente al 8-OHdG. Esto inicia la desmetilación de 5mC. [54]

Como se revisó en 2018, [55] en las neuronas cerebrales, 5mC es oxidado por la familia de dioxigenasas de translocación diez-once (TET) ( TET1 , TET2 , TET3 ) para generar 5-hidroximetilcitosina (5hmC). En pasos sucesivos, las enzimas TET hidroxilan aún más 5hmC para generar 5-formilcitosina (5fC) y 5-carboxilcitosina (5caC). La timina-ADN glicosilasa (TDG) reconoce las bases intermedias 5fC y 5caC y escinde el enlace glucosídico dando como resultado un sitio apirimidínico ( sitio AP ). En una vía de desaminación oxidativa alternativa, 5hmC puede desaminarse oxidativamente por las desaminasas del complejo de edición de ARNm de la apolipoproteína B/citidina desaminasa inducida por actividad (AID/APOBEC) para formar 5-hidroximetiluracilo (5hmU) o 5mC puede convertirse en timina (Thy). 5hmU puede escindirse por TDG, la uracilo-ADN glicosilasa 1 monofuncional selectiva de cadena simple ( SMUG1 ), la ADN glicosilasa 1 similar a Nei ( NEIL1 ) o la proteína de unión a metil-CpG 4 ( MBD4 ). Los sitios AP y los desajustes T:G luego se reparan por enzimas de reparación por escisión de bases (BER) para producir citosina (Cyt).
Dos revisiones [56] [57] resumen la gran cantidad de evidencia sobre el papel crítico y esencial de las ROS en la formación de la memoria . La desmetilación del ADN de miles de sitios CpG durante la formación de la memoria depende de la iniciación por ROS. En 2016, Zhou et al., [54] demostraron que las ROS tienen un papel central en la desmetilación del ADN .
TET1 es una enzima clave involucrada en la desmetilación de 5mCpG. Sin embargo, TET1 solo puede actuar sobre 5mCpG si una ROS ha actuado primero sobre la guanina para formar 8-hidroxi-2'-desoxiguanosina (8-OHdG), lo que da como resultado un dinucleótido 5mCp-8-OHdG (ver la primera figura en esta sección). [54] Después de la formación de 5mCp-8-OHdG, la enzima de reparación por escisión de bases OGG1 se une a la lesión de 8-OHdG sin escisión inmediata. La adherencia de OGG1 al sitio 5mCp-8-OHdG recluta a TET1 , lo que permite que TET1 oxide el 5mC adyacente a 8-OHdG, como se muestra en la primera figura en esta sección. Esto inicia la vía de desmetilación que se muestra en la segunda figura en esta sección.
La expresión alterada de proteínas en las neuronas, controlada por la desmetilación dependiente de ROS de los sitios CpG en los promotores de genes dentro del ADN neuronal, es fundamental para la formación de la memoria. [58]
Se ha observado una disminución de CpG en el proceso de metilación del ADN de los elementos transponibles (ET), donde los ET no solo son responsables de la expansión del genoma, sino también de la pérdida de CpG en el ADN del huésped. Los ET pueden conocerse como "centros de metilación", por lo que durante el proceso de metilación, los ET se propagan hacia el ADN flanqueante una vez que se encuentran en el ADN del huésped. Esta propagación puede dar lugar posteriormente a una pérdida de CpG a lo largo del tiempo evolutivo. Los tiempos evolutivos más antiguos muestran una mayor pérdida de CpG en el ADN flanqueante, en comparación con los tiempos evolutivos más recientes. Por lo tanto, la metilación del ADN puede conducir eventualmente a una pérdida notable de sitios CpG en el ADN vecino. [59]
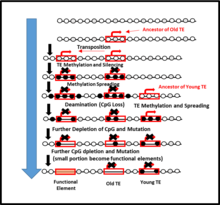
En general, existe una correlación inversa entre el tamaño del genoma y el número de islas CpG, ya que los genomas más grandes suelen tener un mayor número de elementos transponibles. La presión selectiva contra los TE se reduce sustancialmente si se suprime la expresión mediante metilación; otros TE pueden actuar como "centros de metilación" que facilitan la metilación del ADN flanqueante. Dado que la metilación reduce la presión selectiva sobre la secuencia de nucleótidos, la metilación a largo plazo de los sitios CpG aumenta la acumulación de transiciones espontáneas de citosina a timina, lo que da como resultado una pérdida de sitios Cp. [59]
Los elementos Alu son conocidos como el tipo más abundante de elementos transponibles. Algunos estudios han utilizado elementos Alu como una forma de estudiar los factores responsables de la expansión del genoma. Los elementos Alu son ricos en CpG en una mayor cantidad de secuencia, a diferencia de los LINE y ERV. Los Alus pueden funcionar como un centro de metilación, y la inserción en un ADN huésped puede producir metilación del ADN y provocar una propagación hacia el área de ADN flanqueante. Esta propagación es la razón por la que hay una pérdida considerable de CpG y expansión del genoma. [59] Sin embargo, este es un resultado que se analiza a lo largo del tiempo porque los elementos Alus más antiguos muestran una mayor pérdida de CpG en sitios de ADN vecinos en comparación con los más jóvenes.
{{cite book}}: |journal=ignorado ( ayuda ){{cite book}}: Mantenimiento de CS1: falta la ubicación del editor ( enlace ){{cite journal}}: Mantenimiento CS1: fecha y año ( enlace )