Un termómetro de ARN (o termosensor de ARN ) es una molécula de ARN no codificante sensible a la temperatura que regula la expresión genética . [1] Su característica única es que no necesita proteínas ni metabolitos para funcionar, sino que sólo reacciona a los cambios de temperatura. [2] Los termómetros de ARN a menudo regulan los genes necesarios durante una respuesta de choque térmico o de frío , pero se han implicado en otras funciones reguladoras, como la patogenicidad y la inanición . [1]
En general, los termómetros de ARN funcionan cambiando su estructura secundaria y terciaria [3] en respuesta a las fluctuaciones de temperatura. Esta transición estructural puede luego exponer u ocluir regiones importantes del ARN, como un sitio de unión a ribosomas , lo que luego afecta la tasa de traducción de un gen codificador de proteínas cercano .
Los termómetros de ARN, junto con los riboswitches , se utilizan como ejemplos en apoyo de la hipótesis del mundo del ARN . Esta teoría propone que el ARN fue alguna vez el único ácido nucleico presente en las células y fue reemplazado por el actual sistema ADN → ARN → proteína . [4]
Ejemplos de termómetros de ARN incluyen FourU , [5] el elemento regulador cis Hsp90 , [6] el elemento ROSE , [7] el termómetro Lig RNA , [8] y el termómetro Hsp17 . [9]
El primer elemento de ARN sensible a la temperatura se informó en 1989. [10] Antes de esta investigación, se descubrió que las mutaciones aguas arriba del sitio de inicio de la transcripción en un ARNm del fago cIII lambda (λ) afectaban el nivel de traducción de la proteína cIII. [11] Esta proteína participa en la selección de un ciclo de vida lítico o lisogénico en el fago λ, con altas concentraciones de cIII que promueven la lisogenia. [11] Un estudio más detallado de esta región de ARN aguas arriba identificó dos estructuras secundarias alternativas ; Un estudio experimental encontró que las estructuras eran intercambiables y dependían tanto de la concentración de iones de magnesio como de la temperatura. [10] [12] Ahora se cree que este termómetro de ARN fomenta la entrada a un ciclo lítico bajo estrés térmico para que el bacteriófago se replique rápidamente y escape de la célula huésped. [1]
El término "termómetro de ARN" no se acuñó hasta 1999, [13] cuando se aplicó al elemento de ARN rpoH identificado en Escherichia coli . [14] Más recientemente, se han empleado búsquedas bioinformáticas para descubrir varios candidatos novedosos a termómetros de ARN. [15] Sin embargo, las búsquedas tradicionales basadas en secuencias son ineficaces, ya que la estructura secundaria del elemento está mucho más conservada que la secuencia del ácido nucleico . [15]
Las reacciones biológicas y el organismo son sensibles a la temperatura para el funcionamiento celular. Los termómetros de ARN son una forma eficaz de responder a la temperatura porque permiten que las células monitoreen y detecten cambios para mantenerlas vivas y estables. Los mecanismos inducidos por ADN, ARN o proteínas evitan pequeños cambios porque al detectar cualquier cambio externo [16]
Las bacterias utilizan termómetros de ARN para entrar y sobrevivir en sus huéspedes montándose a ellos y provocando fluctuaciones en su temperatura. Las bacterias pueden responder rápidamente contra las condiciones de choque térmico y frío, ya que los termómetros de ARN controlan la expresión genética a un nivel traslacional. [dieciséis]
El primer termómetro de ARN descubierto en cloroplasto de Chlamydomonas reinhardtii , encontrado en la 5'-UTR del ARNm de psaA. Su función era diferente sobre todo porque se consideraba ausente, tiene una estructura secundaria tipo horquilla que protege la secuencia Shine-Dalgarno cuando la temperatura es baja, pero una vez que ocurre un cambio de temperatura, se funde y activa la producción de proteínas. [2] La investigación del termómetro de ARN de C. reinhardtii es la puerta de entrada a la observación del cloroplasto de los organismos fotosintéticos para la regulación genética y cómo puede usarse para la agricultura en algún momento en el futuro, ya que ayuda a las plantas a acostumbrarse a la temperatura externa. [2]
La mayoría de los termómetros de ARN conocidos están ubicados en la región no traducida (UTR) 5′ del ARN mensajero que codifica proteínas de choque térmico , aunque se ha sugerido que este hecho puede deberse, en parte, al sesgo de muestreo y a las dificultades inherentes a la detección de secuencias de ARN cortas y no conservadas. en datos genómicos . [17] [18]
Aunque se encuentra predominantemente en procariotas , se ha encontrado un potencial termómetro de ARN en mamíferos, incluidos los humanos . [19] El termosensor candidato de choque térmico ARN-1 (HSR1) activa el factor de transcripción de choque térmico 1 (HSF1) e induce proteínas protectoras cuando la temperatura celular excede los 37 °C ( temperatura corporal ), evitando así que las células se sobrecalienten. [19]
El primer termómetro de ARN descubierto en cloroplasto de Chlamydomonas reinhardtii, se encuentra en la 5'-UTR del ARNm de psaA. Su función era diferente sobre todo porque se consideraba ausente, tiene una estructura secundaria tipo horquilla que protege la secuencia Shine-Dalgarno cuando la temperatura es baja, pero una vez que ocurre un cambio de temperatura, se funde y activa la producción de proteínas. [2] La investigación del termómetro de ARN de C. reinhardtii es la puerta de entrada a la observación del cloroplasto de los organismos fotosintéticos para la regulación genética y cómo puede usarse para la agricultura en algún momento en el futuro, ya que ayuda a las plantas a acostumbrarse a la temperatura externa. [2]
Los elementos ROSE son una clase de termómetro de ARN bacteriano que regula la activación de genes que tienen pequeñas proteínas de choque térmico. Se derrite a un nivel moderado paralelo al aumento de la temperatura que rodea su entorno. Una vez que se derrite por completo a una temperatura alta de ~42 °C, se procede a la liberación de Shine-Dalgarno y el codón de inicio AUG. Los termómetros de ARN también se pueden encontrar en algunos simbiontes o patógenos de plantas; los simbiontes y patógenos utilizan los termómetros de ARN para regular la expresión genética de la planta. [3] Una bacteria simbiótica bien estudiada es la familia Rhizobiaceae. En la mayoría de las especies de rizobios, los elementos ROSE (de acción cis) eran visibles controlando los genes del choque térmico. [3]

Los termómetros de ARN son estructuralmente simples y pueden fabricarse a partir de secuencias cortas de ARN; el más pequeño tiene solo 44 nucleótidos y se encuentra en el ARNm de una proteína de choque térmico, hsp17, en la especie Synechocystis PCC 6803 . [9] Generalmente, estos elementos de ARN varían en longitud de 60 a 110 nucleótidos [21] y típicamente contienen una horquilla con una pequeña cantidad de pares de bases no coincidentes que reducen la estabilidad de la estructura, permitiendo así un despliegue más fácil en respuesta a un aumento de temperatura. . [17]
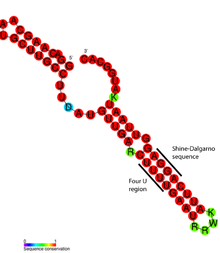
El análisis estructural detallado del termómetro de ARN ROSE reveló que las bases no coincidentes en realidad participan en un emparejamiento de bases no estándar que preserva la estructura helicoidal del ARN (ver figura). Los pares de bases inusuales constan de pares GG, UU y UC-U. Dado que estos pares de bases no canónicos son relativamente inestables, el aumento de temperatura provoca la fusión local de la estructura del ARN en esta región, exponiendo la secuencia de Shine-Dalgarno. [20]
Algunos termómetros de ARN son significativamente más complejos que una sola horquilla, como en el caso de una región encontrada en el ARNm de CspA que se cree que contiene un pseudonudo , así como múltiples horquillas. [22] [23]
Los termómetros de ARN sintético se han diseñado con una estructura sencilla de horquilla única. [24] Sin embargo, la estructura secundaria de estos termómetros de ARN cortos puede ser sensible a la mutación, ya que un solo cambio de base puede inactivar la horquilla in vivo . [25]

Los termómetros de ARN se encuentran en la UTR 5′ del ARN mensajero, aguas arriba de un gen codificador de proteínas. [1] Aquí pueden ocluir el sitio de unión al ribosoma (RBS) y evitar la traducción del ARNm en proteína. [17] A medida que aumenta la temperatura, la estructura en horquilla puede "derretirse" y exponer la secuencia RBS o Shine-Dalgarno para permitir la unión de la pequeña subunidad ribosómica ( 30S ), que luego ensambla otra maquinaria de traducción. [1] El codón de inicio , que normalmente se encuentra 8 nucleótidos aguas abajo de la secuencia de Shine-Dalgarno, [17] señala el comienzo de un gen codificador de proteínas que luego el ribosoma traduce a un producto peptídico . Además de este mecanismo de acción cis , se ha encontrado un único ejemplo de termómetro de ARN de acción trans en el ARNm de RpoS , donde se cree que está involucrado en la respuesta al hambre. [1]
Un ejemplo específico de motivo de termómetro de ARN es el termómetro FourU que se encuentra en Salmonella enterica . [5] Cuando se expone a temperaturas superiores a 45 °C, el bucle de tallo que se empareja con bases opuestas a la secuencia de Shine-Dalgarno se desempareja y permite que el ARNm entre en el ribosoma para que se produzca la traducción. [25] También se ha demostrado que la concentración de iones Mg 2+ afecta la estabilidad de FourU. [26] El termómetro de ARN mejor estudiado se encuentra en el gen rpoH de Escherichia coli . [27] Este termosensor regula positivamente las proteínas de choque térmico bajo altas temperaturas a través de σ 32 , un factor sigma de choque térmico especializado . [13]
Aunque normalmente se asocian con la expresión de proteínas inducida por el calor, los termómetros de ARN también pueden regular las proteínas de choque por frío. [22] Por ejemplo, la expresión de dos proteínas de 7 kDa está regulada por un termómetro de ARN en la bacteria termófila Thermus thermophilus [28] y se ha identificado un mecanismo similar en Enterobacteriales . [23]
Los patógenos pueden utilizar termómetros de ARN sensibles a temperaturas de 37 °C para activar genes específicos de infecciones. [17] Por ejemplo, la regulación positiva de prfA , que codifica un regulador transcripcional clave de genes de virulencia en Listeria monocytogenes , se demostró fusionando el ADN 5' de prfA con el gen de la proteína verde fluorescente ; Luego se transcribió la fusión del gen a partir del promotor T7 en E. coli y se observó fluorescencia a 37 °C pero no a 30 °C. [29]
La hipótesis del mundo del ARN afirma que el ARN fue alguna vez portador de información hereditaria y enzimáticamente activo , con diferentes secuencias que actuaban como biocatalizadores, reguladores y sensores. [30] La hipótesis propone entonces que la vida moderna basada en el ADN, el ARN y las proteínas evolucionó y la selección reemplazó la mayoría de las funciones del ARN con otras biomoléculas . [4]
Se cree que los termómetros de ARN y los riboswitches son evolutivamente antiguos debido a su distribución a gran escala en organismos emparentados lejanamente. [31] Se ha propuesto que, en el mundo del ARN, los termosensores de ARN habrían sido responsables de la regulación dependiente de la temperatura de otras moléculas de ARN. [4] [32] Los termómetros de ARN en organismos modernos pueden ser fósiles moleculares que podrían indicar una importancia anteriormente más extendida en un mundo de ARN. [4]