Flavivirus , rebautizado como Orthoflavivirus en 2023, [3] es un género de virus de ARN de cadena positiva de la familia Flaviviridae . El género incluye el virus del Nilo Occidental , el virus del dengue , el virus de la encefalitis transmitida por garrapatas , el virus de la fiebre amarilla , el virus del Zika y varios otros virus que pueden causar encefalitis , [4] así como flavivirus específicos de insectos (ISF) como el virus del agente de fusión celular (CFAV), el virus Palm Creek (PCV) y el virus del río Parramatta (PaRV). [5] Si bien los flavivirus de doble huésped pueden infectar tanto a vertebrados como a artrópodos, los flavivirus específicos de insectos están restringidos a sus artrópodos competentes. [6] Los medios por los cuales los flavivirus establecen una infección persistente en sus vectores competentes y causan enfermedades en humanos dependen de varias interacciones virus-huésped, incluida la intrincada interacción entre los antagonistas inmunes codificados por los flavivirus y las moléculas efectoras inmunes innatas antivirales del huésped. [7]
Los flavivirus reciben su nombre del virus de la fiebre amarilla; la palabra flavus significa "amarillo" en latín , y la fiebre amarilla a su vez recibe su nombre por su propensión a causar ictericia amarilla en las víctimas. [8]
Los flavivirus comparten varios aspectos comunes: tamaño común (40–65 nm), simetría ( nucleocápside envuelta , icosaédrica ), ácido nucleico ( ARN monocatenario de sentido positivo de alrededor de 10.000–11.000 bases) y apariencia bajo el microscopio electrónico . [ cita requerida ]
La mayoría de estos virus se transmiten principalmente por la picadura de un artrópodo infectado (mosquito o garrapata), y por lo tanto se clasifican como arbovirus . Las infecciones humanas con la mayoría de estos arbovirus son incidentales, ya que los humanos no pueden replicar el virus en títulos lo suficientemente altos como para volver a infectar a los artrópodos necesarios para continuar el ciclo de vida del virus; los humanos son entonces un huésped muerto . Las excepciones a esto son el virus de la fiebre amarilla , el virus del dengue y el virus del Zika . Estos tres virus aún requieren vectores de mosquitos, pero están lo suficientemente adaptados a los humanos como para no depender necesariamente de huéspedes animales (aunque también siguen teniendo importantes rutas de transmisión animal).
Otras vías de transmisión de los arbovirus son la manipulación de cadáveres de animales infectados, las transfusiones de sangre, las relaciones sexuales, el parto y el consumo de productos lácteos no pasteurizados . Sin embargo, la transmisión de vertebrados no humanos a humanos sin un artrópodo vector intermediario se produce en la mayoría de los casos con baja probabilidad. Por ejemplo, las primeras pruebas con fiebre amarilla demostraron que la enfermedad no es contagiosa .
Los virus no arbovirus conocidos de la familia flavivirus se reproducen en artrópodos o vertebrados, pero no en ambos, y un miembro extraño del género afecta a un nematodo . [9]

Los flavivirus tienen una envoltura esférica y una geometría icosaédrica con una simetría pseudo T=3. El diámetro de la partícula viral es de alrededor de 50 nm. [10]
Los flavivirus tienen genomas de ARN monocatenario de sentido positivo que no están segmentados y tienen una longitud de alrededor de 10 a 11 kbp. [10] En general, el genoma codifica tres proteínas estructurales (cápside, prM y envoltura) y siete proteínas no estructurales (NS1, NS2A, NS2B, NS3, NS4A, NS4B, NS5). [11] El ARN genómico está modificado en el extremo 5′ del ARN genómico de cadena positiva con una estructura cap-1 (me 7 -GpppA-me 2 ). [12]

Los flavivirus se replican en el citoplasma de las células hospedadoras. El genoma imita a la molécula de ARNm celular en todos los aspectos, excepto por la ausencia de la cola poliadenilada (poli-A) . Esta característica permite al virus explotar los aparatos celulares para sintetizar proteínas tanto estructurales como no estructurales durante la replicación . El ribosoma celular es crucial para la replicación del flavivirus, ya que traduce el ARN, de manera similar al ARNm celular, lo que da como resultado la síntesis de una única poliproteína . [11]
Las estructuras de la tapa del ARN celular se forman mediante la acción de una ARN trifosfatasa , con guanililtransferasa , N7- metiltransferasa y 2′-O metiltransferasa. El virus codifica estas actividades en sus proteínas no estructurales. La proteína NS3 codifica una ARN trifosfatasa dentro de su dominio helicasa . Utiliza el sitio de hidrólisis de ATP de la helicasa para eliminar el γ-fosfato del extremo 5′ del ARN. El dominio N-terminal de la proteína no estructural 5 (NS5) tiene las actividades de N7-metiltransferasa y guanililtransferasa necesarias para formar estructuras de tapa de ARN maduras. La afinidad de unión del ARN se reduce por la presencia de ATP o GTP y se mejora por la S-adenosil metionina . [12] Esta proteína también codifica una 2′-O metiltransferasa.

Una vez traducida , la poliproteína se escinde mediante una combinación de proteasas virales y del huésped para liberar productos polipeptídicos maduros. [13] Sin embargo, la modificación postraduccional celular depende de la presencia de una cola de poli-A; por lo tanto, este proceso no depende del huésped. En cambio, la poliproteína contiene una característica autocatalítica que libera automáticamente el primer péptido, una enzima específica del virus. Esta enzima puede entonces escindir la poliproteína restante en los productos individuales. Uno de los productos escindidos es una ARN polimerasa dependiente de ARN , responsable de la síntesis de una molécula de ARN de sentido negativo. En consecuencia, esta molécula actúa como plantilla para la síntesis de la progenie genómica de ARN. [ cita requerida ]
La replicación del ARN genómico de los flavivirus se produce en las membranas del retículo endoplasmático rugoso , en los compartimentos membranosos. Posteriormente, se ensamblan nuevas partículas virales. Esto ocurre durante el proceso de gemación , que también es responsable de la acumulación de la envoltura y la lisis celular . [ cita requerida ]
La quinasa 2 del receptor acoplado a proteína AG (también conocida como ADRBK1) parece ser importante en la entrada y replicación de varios virus en Flaviviridae . [14]
Los humanos, los mamíferos, los mosquitos y las garrapatas son los huéspedes naturales. Las vías de transmisión son la zoonosis y la picadura. [10]

El genoma de ARN de sentido positivo del Flavivirus contiene regiones no traducidas (UTR) 5' y 3'.
Los 5'UTR tienen una longitud de 95 a 101 nucleótidos en el virus del dengue . [15] Hay dos elementos estructurales conservados en el 5'UTR del flavivirus , un bucle de tallo grande (SLA) y un bucle de tallo corto (SLB). El SLA se pliega en una estructura en forma de Y con un bucle de tallo lateral y un bucle superior pequeño. [15] [16] Es probable que el SLA actúe como promotor y sea esencial para la síntesis de ARN viral. [17] [18] El SLB está involucrado en interacciones entre el 5'UTR y el 3'UTR que resultan en la ciclización del ARN viral, que es esencial para la replicación viral. [19]

Los 3'UTR tienen una longitud de entre 0,3 y 0,5 kb y contienen varias estructuras secundarias muy conservadas que se limitan a la familia de los flavivirus . La mayoría de los análisis se han realizado utilizando el virus del Nilo Occidental (VNO) para estudiar la función del 3'UTR. [ cita requerida ]
Actualmente se han identificado 8 estructuras secundarias dentro del 3'UTR del WNV y son (en el orden en que se encuentran con el 3'UTR) SL-I, SL-II, SL-III, SL-IV, DB1, DB2 y CRE. [20] [21] Algunas de estas estructuras secundarias se han caracterizado y son importantes para facilitar la replicación viral y proteger el 3'UTR de la digestión por endonucleasa 5' . La resistencia a la nucleasa protege el fragmento de ARN del 3' UTR corriente abajo de la degradación y es esencial para la citopaticidad y patogenicidad inducidas por el virus. [ cita requerida ]
Se ha sugerido que SL-II contribuye a la resistencia a las nucleasas. [21] Puede estar relacionado con otro bucle de horquilla identificado en el 5'UTR del genoma del virus de la encefalitis japonesa (JEV). [22] La horquilla del JEV está significativamente sobrerrepresentada tras la infección de la célula huésped y se ha sugerido que la estructura de la horquilla puede desempeñar un papel en la regulación de la síntesis de ARN. [ cita requerida ]
Esta estructura secundaria se encuentra dentro del extremo 3'UTR del genoma del flavivirus, antes de los elementos DB. Se desconoce la función de esta estructura conservada, pero se cree que contribuye a la resistencia a la ribonucleasa. [ cita requerida ]
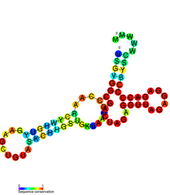
Estas dos estructuras secundarias conservadas también se conocen como elementos pseudo-repetidos. Se identificaron originalmente dentro del genoma del virus del dengue y se encuentran adyacentes entre sí dentro del 3'UTR. Parecen estar ampliamente conservadas en Flaviviradae. Estos elementos DB tienen una estructura secundaria que consta de tres hélices y desempeñan un papel en asegurar una traducción eficiente. La eliminación de DB1 tiene una reducción pequeña pero significativa en la traducción, pero la eliminación de DB2 tiene poco efecto. La eliminación de DB1 y DB2 redujo la eficiencia de la traducción del genoma viral al 25%. [20]
CRE es el elemento de replicación que actúa en cis, también conocido como elementos 3'SL del ARN, y se cree que es esencial en la replicación viral al facilitar la formación de un "complejo de replicación". [23] Aunque se ha presentado evidencia de la existencia de una estructura de pseudonudo en este ARN, no parece estar bien conservada en los flavivirus. [24] Se ha demostrado que las deleciones del UTR 3' de los flavivirus son letales para los clones infecciosos.
Posteriormente se encontró una estructura de horquilla conservada (cHP) en varios genomas de flavivirus y se cree que dirige la traducción de las proteínas de la cápside. Se encuentra justo aguas abajo del codón de inicio AUG . [25]

El ARN subgenómico del flavivirus (sfRNA) es una extensión del UTR 3' y se ha demostrado que desempeña un papel en la replicación y patogénesis del flavivirus . [26] El sfRNA se produce por degradación incompleta del ARN viral genómico por la exorribonucleasa 1 5'-3' de las células huésped (XRN1). [27] A medida que la XRN1 degrada el ARN viral, se detiene en los bucles de tallo formados por la estructura secundaria del UTR 5' y 3'. [28] Esta pausa da como resultado un fragmento no digerido del ARN genómico conocido como sfRNA. El sfRNA influye en el ciclo de vida del flavivirus de una manera dependiente de la concentración. La acumulación de sfRNA provoca (1) la antagonización de la respuesta inmune innata de la célula, disminuyendo así la defensa del huésped contra el virus [29] (2) la inhibición de la actividad de XRN1 y Dicer para modificar las vías de RNAi que destruyen el ARN viral [30] (3) la modificación del complejo de replicación viral para aumentar la reproducción viral. [31] En general, el sfRNA está implicado en múltiples vías que comprometen las defensas del huésped y promueven la infección por flavivirus. [ cita requerida ]

Los flavivirus pueden dividirse en dos clados: uno con virus transmitidos por vectores y otro sin vector conocido. [32] El clado de vectores, a su vez, puede subdividirse en un clado transmitido por mosquitos y un clado transmitido por garrapatas. Estos grupos pueden dividirse a su vez. [33]
El grupo de los mosquitos se puede dividir en dos ramas: una rama contiene virus neurotrópicos, a menudo asociados con enfermedades encefálicas en humanos o ganado. Esta rama tiende a ser transmitida por especies de Culex y tiene reservorios en aves. La segunda rama son los virus no neurotrópicos asociados con enfermedades hemorrágicas humanas. Estos tienden a tener especies de Aedes como vectores y huéspedes primates . [ cita requerida ]
Los virus transmitidos por garrapatas también forman dos grupos distintos: uno está asociado con las aves marinas y el otro – los virus del complejo de encefalitis transmitido por garrapatas – está asociado principalmente con los roedores . [ cita requerida ]
Los virus que carecen de un vector conocido se pueden dividir en tres grupos: uno estrechamente relacionado con los virus transmitidos por mosquitos, que está asociado con los murciélagos ; un segundo, genéticamente más distante, también está asociado con los murciélagos; y un tercer grupo está asociado con los roedores. [ cita requerida ]
Las relaciones evolutivas entre los elementos virales endogenizados de los flavivirus y los flavivirus contemporáneos utilizando métodos de máxima verosimilitud han identificado que los flavivirus transmitidos por artrópodos probablemente surgieron de una fuente arácnida. [34] Esto contradice trabajos anteriores con un número menor de virus existentes que mostraban que los virus transmitidos por garrapatas surgieron de un grupo transmitido por mosquitos. [35]
Se han encontrado varios genomas parciales y completos de flavivirus en invertebrados acuáticos como la araña marina Endeis spinosa [36] y varios crustáceos y cefalópodos. [37] Estas secuencias parecen estar relacionadas con las de los flavivirus específicos de insectos y también con los grupos de virus de murciélago Tamana. Si bien actualmente no está claro cómo encajan los flavivirus acuáticos en la evolución de este grupo de virus, existe cierta evidencia de que uno de estos virus, el flavivirus del tiburón Wenzhou, infecta tanto a un crustáceo ( Portunus trituberculatus ) como a un tiburón huésped del Pacífico ( Scoliodon macrorhynchos ), [38] [37] lo que indica un ciclo de vida de arbovirus acuático.

Se han realizado estimaciones de los tiempos de divergencia para varios de estos virus. [39] El origen de estos virus parece ser al menos hace 9400 a 14.000 años. Las cepas del dengue del Viejo Mundo y del Nuevo Mundo divergieron hace entre 150 y 450 años. Las cepas de encefalitis transmitida por garrapatas europeas y del Lejano Oriente divergieron hace unos 1087 (1610–649) años. Los virus de la encefalitis transmitida por garrapatas europeas y de la enfermedad de Loupe divergieron hace unos 572 (844–328) años. Esta última estimación es consistente con los registros históricos. El virus Kunjin divergió del virus del Nilo Occidental hace aproximadamente 277 (475–137) años. Este tiempo corresponde al asentamiento de Australia desde Europa. El grupo de la encefalitis japonesa parece haber evolucionado en África hace 2000–3000 años y luego se extendió inicialmente al sudeste asiático antes de migrar al resto de Asia.
Los estudios filogenéticos del virus del Nilo Occidental han demostrado que surgió como un virus distinto hace unos 1000 años. [40] Este virus inicial se desarrolló en dos linajes distintos, el linaje 1 y sus múltiples perfiles son la fuente de la transmisión epidémica en África y en todo el mundo. El linaje 2 se consideró una zoonosis de África . Sin embargo, en 2008, el linaje 2, previamente visto solo en caballos en África subsahariana y Madagascar, comenzó a aparecer en caballos en Europa, donde el primer brote conocido afectó a 18 animales en Hungría en 2008. [41] El virus del Nilo Occidental de linaje 1 se detectó en Sudáfrica en 2010 en una yegua y su feto abortado ; anteriormente, solo se había detectado el virus del Nilo Occidental de linaje 2 en caballos y humanos en Sudáfrica. [42] Un caso fatal de 2007 en una orca en Texas amplió el rango de hospedadores conocido del virus del Nilo Occidental para incluir a los cetáceos . [43]
El virus de la fiebre hemorrágica de Omsk parece haber evolucionado en los últimos 1000 años. [44] Los genomas virales se pueden dividir en 2 clados: A y B. El clado A tiene cinco genotipos y el clado B tiene uno. Estos clados se separaron hace unos 700 años. Esta separación parece haber ocurrido en la provincia de Kurgan. El clado A posteriormente se dividió en los clados C, D y E hace 230 años. Los clados C y E parecen haberse originado en las provincias de Novosibirsk y Omsk, respectivamente. La rata almizclera Ondatra zibethicus , que es muy susceptible a este virus, se introdujo en esta área en la década de 1930.
En el género Flavivirus hay 53 especies definidas: [45]

Especies y cepas ordenadas por vectores:
Grupo de virus transmitidos por garrapatas en mamíferos
Grupo de virus transmitidos por garrapatas de aves marinas
La vacuna 17D contra la fiebre amarilla , muy exitosa y presentada en 1937, produjo reducciones drásticas en la actividad epidémica. [ cita requerida ]
A mediados del siglo XX se introdujeron vacunas eficaces contra la encefalitis japonesa inactivada y la encefalitis transmitida por garrapatas . Los efectos adversos inaceptables han impulsado el cambio de una vacuna contra la encefalitis japonesa inactivada en el cerebro de ratón a vacunas contra la encefalitis japonesa de segunda generación, más seguras y eficaces. Estas pueden llegar a utilizarse ampliamente para prevenir eficazmente esta grave enfermedad en las enormes poblaciones de Asia (norte, sur y sudeste). [ cita requerida ]
Los virus del dengue producen muchos millones de infecciones al año debido a la transmisión por un mosquito vector mundial exitoso. Como el control de los mosquitos ha fracasado, varias vacunas contra el dengue se encuentran en distintas etapas de desarrollo. CYD-TDV, vendida bajo el nombre comercial Dengvaxia, es una vacuna quimérica tetravalente que empalma genes estructurales de los cuatro virus del dengue en una estructura principal de la fiebre amarilla 17D. [50] [51] Dengvaxia está aprobada en cinco países. [52]
Un enfoque alternativo para el desarrollo de vectores de vacunas de flavivirus se basa en el uso de virus que infectan a insectos. Los flavivirus específicos de insectos, como el virus Binjari, no pueden replicarse en células de vertebrados. Sin embargo, los virus recombinantes en los que los genes de la proteína estructural (prME) del virus Binjari se intercambian con los del virus del dengue, el virus Zika, el virus del Nilo Occidental, el virus de la fiebre amarilla o el virus de la encefalitis japonesa se replican de manera eficiente en células de insectos donde se producen altos títulos de partículas virales infecciosas. La inmunización de ratones con una vacuna Binjari que contiene las proteínas estructurales del virus Zika protegió a los ratones de la enfermedad después del desafío . Un enfoque similar emplea el virus alfa Eilat específico de insectos como plataforma de vacuna . ... Estas nuevas plataformas de vacunas generadas a partir de flavivirus y alfavirus específicos de insectos representan enfoques asequibles, eficientes y seguros para el desarrollo rápido de vacunas infecciosas atenuadas contra patógenos de estas dos familias de virus. [53]
{{cite journal}}: CS1 maint: multiple names: authors list (link) {{cite journal}}: CS1 maint: multiple names: authors list (link) {{cite journal}}: CS1 maint: multiple names: authors list (link){{cite journal}}: CS1 maint: multiple names: authors list (link)