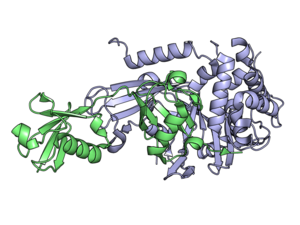

Las enzimas desubiquitinantes ( DUB ), también conocidas como peptidasas desubiquitinantes , isopeptidasas desubiquitinantes , deubiquitinasas , ubiquitina proteasas , ubiquitina hidrolasas o ubiquitina isopeptidasas , son un gran grupo de proteasas [1] que escinden la ubiquitina de las proteínas. [2] La ubiquitina se une a las proteínas para regular la degradación de proteínas a través del proteasoma y el lisosoma ; coordinar la localización celular de las proteínas; activar e inactivar proteínas; y modular las interacciones proteína-proteína . [3] [4] [5] Las DUB pueden revertir estos efectos al escindir el enlace peptídico o isopeptídico entre la ubiquitina y su proteína sustrato. En los humanos hay casi 100 genes DUB, que se pueden clasificar en dos clases principales: cisteína proteasas y metaloproteasas . Las proteasas de cisteína incluyen las proteasas específicas de ubiquitina (USP), las hidrolasas de ubiquitina C-terminal (UCH), las proteasas de dominio Machado-Josephin (MJD) y las proteasas de tumores ováricos (OTU). El grupo de metaloproteasas contiene solo las proteasas de dominio N-terminal+ (MPN+) (JAMM) de Jab1/Mov34/Mpr1 Pad1. [2]
En los humanos hay 102 genes DUB putativos, que pueden clasificarse en dos clases principales: cisteína proteasas y metaloproteasas , que consisten en 58 proteasas específicas de ubiquitina (USP), 4 hidrolasas C-terminales de ubiquitina (UCH), 5 proteasas del dominio Machado-Josephin (MJD), 14 proteasas tumorales ováricas (OTU) y 14 genes que contienen el dominio Jab1/Mov34/Mpr1 Pad1 N-terminal+ (MPN+) (JAMM). Se predice que 11 de estas proteínas no son funcionales, dejando 79 enzimas funcionales. [6] En la levadura, las USP se conocen como proteasas de procesamiento específicas de ubiquitina (UBP).
Hay seis superfamilias principales de proteasas de cisteína DUB: [7]
También existe un grupo putativo poco conocido de DUB llamado superfamilia de peptidasas de pliegue de papaína permutadas de virus dsADN y eucariotas (PPPDE), que, si se demuestra que son DUB genuinos, serían los séptimos en la clase de proteasas de cisteína. [11]
Las proteínas de la superfamilia del dominio Jab1/Mov34/Mpr1 Pad1 N-terminal+ (MPN+) (JAMM) se unen al zinc y, por lo tanto, son metaloproteasas. [7]
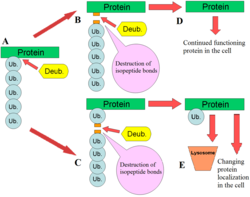
Las DUB desempeñan varias funciones en la vía de la ubiquitina. Una de las funciones mejor caracterizadas de las DUB es la eliminación de cadenas de monoubiquitina y poliubiquitina de las proteínas. Estas modificaciones son una modificación postraduccional (adición a una proteína después de que se ha creado) donde las proteínas ubiquitina individuales o cadenas de ubiquitina se agregan a las lisinas de una proteína sustrato. Estas modificaciones de ubiquitina se agregan a las proteínas mediante la maquinaria de ubiquitinación; enzimas activadoras de ubiquitina (E1), enzimas conjugadoras de ubiquitina (E2) y ligasas de ubiquitina (E3). El resultado es ubiquitina unida a residuos de lisina a través de un enlace isopeptídico . [12] Las proteínas se ven afectadas por estas modificaciones de varias maneras: regulan la degradación de proteínas a través del proteasoma y el lisosoma ; coordinan la localización celular de las proteínas; activan e inactivan proteínas; y modulan las interacciones proteína-proteína . [3] [4] [5] Los DUB desempeñan un papel antagónico en este eje al eliminar estas modificaciones, revirtiendo así el destino de las proteínas. [2] Además, un papel menos comprendido de los DUB es la escisión de proteínas similares a la ubiquitina, como SUMO y NEDD8 . Algunos DUB pueden tener la capacidad de escindir enlaces isopeptídicos entre estas proteínas y proteínas sustrato. [13]
Activan la ubiquitina mediante la proteólisis (descomposición) de las formas inactivas expresadas de ubiquitina. La ubiquitina está codificada en mamíferos por 4 genes diferentes: UBA52 , RPS27A , UBB y UBC . Un conjunto similar de genes se encuentra en otros eucariotas como la levadura. Los genes UBA52 y RPS27A producen ubiquitina que se fusiona a proteínas ribosómicas y los genes UBB y UBC producen poliubiquitina (una cadena de ubiquitina unida por sus extremos C y N ). [14] [15] Los DUB escinden la ubiquitina de estas proteínas, produciendo unidades individuales activas de ubiquitina. [2]
Las DUB también escinden proteínas de ubiquitina individuales que pueden haber tenido sus colas C-terminales unidas accidentalmente a pequeños nucleófilos celulares . [2] Estas ubiquitinamidas y ubiquitintioésteres pueden formarse durante reacciones de ubiquitinación estándar por la cascada E1-E2-E3. El glutatión y las poliaminas son dos nucleófilos que podrían atacar el enlace tioléster entre la ubiquitina y estas enzimas. La hidrolasa C-terminal de la ubiquitina es un ejemplo de DUB que hidroliza estos enlaces con amplia especificidad. [13] [16]
Las cadenas de poliubiquitina libre son escindidas por los DUB para producir monoubiquitina. Las cadenas pueden ser producidas por la maquinaria E1-E2-E3 en la célula libre de cualquier proteína sustrato. Otra fuente de poliubiquitina libre es el producto de la escisión de la ubiquitina con el sustrato. Si los DUB escinden la base de la cadena de poliubiquitina que está unida a una proteína, toda la cadena quedará libre y deberá ser reciclada por los DUB. [2]

Los DUB suelen contener un dominio catalítico rodeado de uno o más dominios accesorios, algunos de los cuales contribuyen al reconocimiento del objetivo. Estos dominios adicionales incluyen el dominio presente en las proteasas específicas de ubiquitina (DUSP); el dominio similar a la ubiquitina (UBL); el dominio de homología de meprina y TRAF (MATH); el dominio de la proteasa específica de ubiquitina de dedo de zinc (ZnF-UBP); el dominio mieloide, nervioso y DEAF1 de dedo de zinc (ZnF-MYND); el dominio asociado a la ubiquitina (UBA); el dominio CHORD-SGT1 (CS); el dominio de interacción y tráfico de microtúbulos (MIT); el dominio similar a la rodenasa; el dominio TBC/RABGAP; y el dominio B-box. [6] [17]
El dominio catalítico de las DUB es lo que las clasifica en grupos particulares; USP, OTU, MJD, UCH y MPN+/JAMM. Los primeros 4 grupos son cisteína proteasas , mientras que el último es una metaloproteasa de zinc . Las DUB de cisteína proteasa son similares a la papaína y, por lo tanto, tienen un mecanismo de acción similar. Utilizan díadas o tríadas catalíticas (ya sea de dos o tres aminoácidos ) para catalizar la hidrólisis de los enlaces amida entre la ubiquitina y el sustrato. Los residuos del sitio activo que contribuyen a la actividad catalítica de las DUB de cisteína proteasa son cisteína (díada/tríada), histidina (díada/tríada) y aspartato o asparagina (solo tríada). La histidina es polarizada por el aspartato o la asparagina en tríadas catalíticas o por otras formas en díadas. Este residuo polarizado reduce el pKa de la cisteína, lo que le permite realizar un ataque nucleofílico sobre el enlace isopeptídico entre el extremo C de la ubiquitina y el sustrato lisina . Las metaloproteasas coordinan los iones de zinc con residuos de histidina, aspartato y serina , que activan las moléculas de agua y les permiten atacar el enlace isopeptídico. [18] [19]
Los dominios similares a la ubiquitina (UBL) tienen una estructura (pliegue) similar a la ubiquitina, excepto que carecen de los residuos terminales de glicina. Se propone que 18 USP tienen dominios UBL. Solo otros 2 DUB tienen UBL fuera del grupo USP: OTU1 y VCPIP1 . USP4, USP7, USP11, USP15, USP32, USP40 y USP47 tienen múltiples dominios UBL. A veces, los dominios UBL están en tándem, como en USP7 donde están presentes 5 dominios UBL C-terminales en tándem . USP4, USP6, USP11, USP15, USP19, USP31, USP32 y USP43 tienen dominios UBL insertados en el dominio catalítico. Las funciones de los dominios UBL son diferentes entre USP, pero comúnmente regulan la actividad catalítica de USP. Pueden coordinar la localización en el proteasoma (USP14); regulan negativamente las USP al competir por el sitio catalítico de la USP (USP4) e inducen cambios conformacionales para aumentar la actividad catalítica (USP7). [17] [20] [21] Al igual que otros dominios UBL, la estructura de los dominios UBL de la USP muestra un pliegue de agarre β. [22] [23]
Se encuentran dominios DUSP en tándem simples o múltiples de aproximadamente 120 residuos en seis USP. La función del dominio DUSP es actualmente desconocida, pero puede desempeñar un papel en la interacción proteína-proteína , en particular en el reconocimiento del sustrato de los DUB. Esto se predice debido a la hendidura hidrofóbica presente en el dominio DUSP de USP15 y a que algunas interacciones de proteínas con USP que contienen DUSP no ocurren sin estos dominios. El dominio DUSP muestra un nuevo pliegue tipo trípode que comprende tres hélices y una lámina beta antiparalela hecha de tres hebras. Este pliegue se asemeja a las patas (hélices) y al asiento (lámina beta) del trípode. Dentro de la mayoría de los dominios DUSP en USP hay una secuencia conservada de aminoácidos conocida como motivo PGPI . Esta es una secuencia de cuatro aminoácidos; prolina , glicina , prolina e isoleucina , que se empaqueta contra el haz de tres hélices y está altamente ordenada. [6] [24]
Aún queda por dilucidar el alcance total del papel de las DUB en las enfermedades. Su participación en las enfermedades se predice debido a funciones conocidas en los procesos fisiológicos que intervienen en los estados patológicos, incluidos el cáncer y los trastornos neurológicos. [25]
La enzima USP28 se encuentra sobreexpresada en diferentes tipos de cáncer como el de colon o pulmón. Además, USP28 desubiquitina y estabiliza oncogenes importantes como c-Myc , Notch1 , c-jun o ΔNp63 . [26] [27] [28] En tumores escamosos, USP28 regula la resistencia a la quimioterapia regulando la reparación del ADN a través del eje de la vía ΔNp63 -anemia de Fanconia. [29]
Las enzimas desubiquitinantes UCH-L3 y YUH1 son capaces de hidrolizar la ubiquitina mutante UBB+1 a pesar del hecho de que la glicina en la posición 76 está mutada. [30]
Los niveles de UCH-L1 son altos en varios tipos de neoplasias malignas ( cáncer ). [31]
Las DUB desempeñan un papel activo en la modulación del ciclo celular. La proteasa de procesamiento específico de ubiquitina (USP) es una familia de enzimas desubiquitinantes que desempeñan un papel crucial en la regulación del ciclo celular. [32] Dos de estas enzimas son la USP17 y la USP44. La USP17 regula las vías responsables del progreso de las células a través del ciclo celular. [33] Sus objetivos incluyen reguladores de Ras, CDK2 y ciclina A. [34] La USP44 desempeña un papel importante en la iniciación de la anafase. [35] Una nueva investigación sobre el punto de control mitótico ha revelado un nuevo papel de la USP44 en la regulación de la progresión del ciclo celular. [35]
La vía ERK permite la transducción de señales mitogénicas externas en señales intracelulares que promueven la proliferación celular. Uno de los reguladores clave de esta vía es Ras, una GTPasa que, al activarse, se une a GTP para "activar" la cascada de señalización posterior. La enzima convertidora de Ras 1 (RCE1) escinde postraduccionalmente los 3 residuos en el extremo C de Ras, lo que permite que Ras se localice correctamente en la membrana plasmática. [36]
La USP17 actúa para desubiquitinar los dominios de ubiquitina K63 en RCE1. [34] Dicha estabilización de RCE1 permite la localización adecuada de Ras, promoviendo así la proliferación tras la activación de los receptores tempranos en la vía ERK. La hiperactividad de Ras puede provocar una desregulación del ciclo celular. [37] Por lo tanto, la regulación de Ras a través de la USP17 actúa como otro punto en la regulación de Ras.
Las quinasas dependientes de ciclina (CDK) son una familia de enzimas que fosforilan residuos de serina y treonina para impulsar la célula a través del ciclo celular. La activación de CDK2 es fundamental para la transición G1-S. Para que CDK2 se active, la ciclina A debe unirse al complejo de quinasa dependiente de ciclina (CDKC). El ciclo de división celular 25A (CDC25A) es una fosfatasa que elimina un grupo fosfato inhibidor de CDK2. [38] Mientras que la ubiquitinación marcaría CDC25A para degradación, bloqueando así la progresión a la fase S, USP17 desubiquitina CDC25A. [34] Un aumento en la estabilidad de CDC25A promueve la actividad de CDKC, impulsando así la célula a través de la transición G1-S.
La USP17 también regula la progresión del ciclo celular al actuar sobre SETD8 para regular negativamente la transcripción del inhibidor de la quinasa dependiente de ciclina 1 (CDKN1A), también conocido como p21. [34] La CDKN1A se une a la CDK2 y la inhibe utilizando su dominio de unión N-terminal, bloqueando así la progresión a través de la transición G1-S. La SETD8, una metiltransferasa, utiliza S-adenosil metionina para metilar el residuo Lys20 de la histona 4, lo que da como resultado la condensación de los cromosomas. [39] Esta compactación del ADN regula negativamente la transcripción de CDKN1A. La USP17 desubiquitina SETD8, reduciendo así su propensión a la degradación y aumentando su estabilidad intracelular. [34] La regulación negativa resultante en la transcripción de CDKN1A promueve la actividad de CDK2, lo que permite que la célula progrese a través de la transición G1-S. Véase el esquema del papel de las DUB en la regulación del ciclo celular. [34]

El punto de control del huso (también conocido como punto de control mitótico) garantiza la separación adecuada de los cromosomas. En términos generales, el punto de control mitótico promueve la fidelidad en la segregación cromosómica, lo que aumenta la probabilidad de que cada célula hija reciba solo un cromosoma duplicado. [40] Este mecanismo es crucial, ya que los errores en la separación cromosómica se han relacionado con el cáncer, los defectos de nacimiento y la resistencia a los antibióticos en los patógenos. [41] Una de las proteínas reguladoras centrales es el complejo promotor de anafase (APC/C). APC/C ubiquitina la securina. [42] La destrucción resultante de la separasa de liberación de seguridad, [40] que hidroliza la cohesión, la proteína que une las cromátidas hermanas.
Una nueva investigación de Stegmeier y colegas [35] publicada en la revista Nature demuestra un papel crucial para USP44 en la regulación del punto de control del huso. Usando un cribado de shRNA, se identificó que USP44 estabiliza la inhibición de APC/C [35] La unión de CDC20 a APC/C es necesaria para la ubiquitinación de la securina. [43] Una proteína llamada hMAD2 puede formar un trímero inactivo con APC y CDC20, formando el complejo hMAD2-CDC-APC. [43] Tras la ubiquitinación de CDC20 por UbcH10, hMAD2 se disocia y APC/C se activa. [44] Es importante señalar que la ubiquitinación de CDC20 no sirve para marcarlo para la degradación, sino que promueve la disociación de hMAD2 del complejo hMAD2-CDC-APC. La USP44, una proteasa que procesa específicamente la ubiquitina, puede estabilizar el complejo inactivo hMAD2-CDC-APC al contrarrestar la ubiquitinación de UbcH10. Esto bloquea la disociación de hMAD2 y permite la regulación adecuada de APC/C, manteniéndolo inactivo hasta que se adhiera correctamente al huso mitótico. Una vez que se produce la unión correcta, el comportamiento similar a un interruptor permite la activación de APC/C. [35] Esto da como resultado la ruptura de la cohesión, lo que permite la separación de las cromátidas hermanas.
Los daños en el ADN pueden resultar catastróficos para un organismo. Los mecanismos de mutación del ADN incluyen estrés oxidativo, errores de replicación del ADN, carcinógenos exógenos, radiación y mutación espontánea de bases. Cuando se produce un daño en el ADN, se detiene la progresión del ciclo celular para evitar la propagación de la mutación. El gen TP53 (también conocido como p53) es crucial para garantizar la conservación del genoma. [45] Las enzimas desubiquitinantes desempeñan un papel fundamental en el mantenimiento de la función del p53.
En las células sanas, p53 activa la ligasa de ubiquitina E3 MDM2, que a su vez ubiquitina p53. Esto crea un ciclo de retroalimentación negativa , por el cual la degradación de p53 permite que las células fluyan a través del ciclo celular. Cuando se daña el ADN, la proteasa de procesamiento específico de ubiquitina 7 (USP7) estabiliza p53 al escindir la ubiquitina. [46] Para que USP7 desubiquitine p53, debe localizarse en el núcleo. Sin embargo, no se ha encontrado ninguna secuencia de localización nuclear (NLS). [47] A pesar de que no se conoce ninguna NLS, un estudio mostró que, tras la eliminación del extremo N de USP7, no se produjo localización nuclear. [47] Es posible que otras proteínas faciliten la entrada nuclear de USP7.
Una vez estabilizado, el p53 puede ejercer su función de supresión tumoral. Las vías descendentes del p53 actúan para detener la progresión del ciclo celular en las fases G1 o G2 del ciclo celular [48] o promover la muerte celular, dependiendo de la gravedad del daño del ADN. [49] Ver esquema del papel de USP7 en la vía dependiente del p53. [48] o promover la muerte celular, dependiendo de la gravedad del daño del ADN. [49] Ver esquema del papel de USP7 en la vía dependiente del p53. [49]

{{cite book}}: |journal=ignorado ( ayuda )