El ARN mensajero de transferencia (abreviado tmRNA , también conocido como ARN 10Sa y por su nombre genético SsrA ) es una molécula de ARN bacteriano con propiedades duales similares a las del ARNt y del ARN mensajero . El tmRNA forma un complejo de ribonucleoproteína ( tmRNP ) junto con la proteína pequeña B (SmpB), el factor de elongación Tu ( EF-Tu ) y la proteína ribosómica S1. En la transtraducción , el tmRNA y sus proteínas asociadas se unen a los ribosomas bacterianos que se han estancado en medio de la biosíntesis de proteínas , por ejemplo, al llegar al final de un ARN mensajero que ha perdido su codón de parada. El tmRNA es notablemente versátil: recicla el ribosoma estancado, agrega una etiqueta inductora de proteólisis al polipéptido inacabado y facilita la degradación del ARN mensajero aberrante . [1] En la mayoría de las bacterias, estas funciones las llevan a cabo ARNtm de una sola pieza. En otras especies bacterianas, un gen ssrA permutado produce un ARNtm de dos piezas en el que dos cadenas de ARN independientes se unen mediante apareamiento de bases.
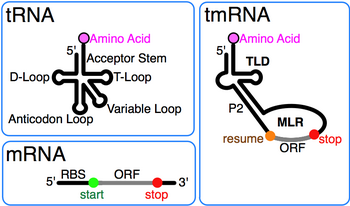
El ARNtm se denominó por primera vez ARN 10Sa en 1979, después de que una fracción electroforética mixta "10S" de ARN de Escherichia coli se resolviera aún más en ARNtm y ARN ARNasa P de tamaño similar (10Sb). [2] La presencia de pseudouridina en el ARN 10S mixto insinuó que el ARNtm tiene bases modificadas que también se encuentran en el ARNt . La similitud en el extremo 3' del ARNtm con el tallo-bucle T del ARNt se reconoció por primera vez al secuenciar ssrA de Mycobacterium tuberculosis . [3] La comparación de secuencias posterior reveló el dominio similar al ARNt (TLD) completo formado por los extremos 5' y 3' del ARNtm, incluido el tallo aceptor con elementos como los del ARNt de alanina que promueven su aminoacilación por la ligasa de ARNt de alanina . [4] También reveló diferencias con el ARNt : en el ARNtm falta el brazo anticodón y la región del brazo D es un bucle sin pares de bases.

La estructura secundaria completa del ARNtm de E. coli se dilucidó mediante análisis comparativo de secuencias y sondeo estructural . [5] [6] Los pares de bases Watson-Crick y GU se identificaron comparando las secuencias del ARNtm bacteriano utilizando métodos computacionales automatizados en combinación con procedimientos de alineación manual. [7] [8] La figura adjunta muestra el patrón de apareamiento de bases de este ARNtm prototípico, que está organizado en 12 hélices con soporte filogenético (también llamadas emparejamientos P1 a P12), algunas divididas en segmentos helicoidales.
Una característica destacada de cada ARNtm es el dominio conservado similar al ARNt (TLD), compuesto por las hélices 1, 12 y 2a (análogos del tallo aceptor del ARNt, el tallo T y el tallo variable, respectivamente), y que contiene los extremos 5' monofosfato y 3' CCA alanilable. La región similar al ARNm (MLR) es en el ARNtm estándar un gran bucle que contiene pseudonudos y una secuencia codificante (CDS) para el péptido etiqueta , marcado por el codón de reanudación y el codón de parada . El péptido etiqueta codificado (ANDENYALAA en E. coli ) varía entre bacterias, quizás dependiendo del conjunto de proteasas y adaptadores disponibles. [9]
Los tmRNA contienen típicamente cuatro pseudonudos , uno (pk1) aguas arriba del péptido etiqueta CDS, y los otros tres pseudonudos (pk2 a pk4) aguas abajo del CDS. Las regiones de pseudonudos, aunque generalmente se conservan, son evolutivamente plásticas. Por ejemplo, en los tmRNA (de una sola pieza) de las cianobacterias , pk4 se sustituye por dos pseudonudos más pequeños dispuestos en tándem. Esto sugiere que el plegamiento del tmRNA fuera del TLD puede ser importante, pero la región de pseudonudos carece de residuos conservados y los pseudonudos se encuentran entre las primeras estructuras que se pierden a medida que las secuencias ssrA divergen en los linajes de plástidos y endosimbiontes. El apareamiento de bases en la región de tres pseudonudos del tmRNA de E. coli se interrumpe durante la transtraducción. [7] [10]
Se ha informado de ssrA permutado circularmente en tres linajes principales: i) todas las alfaproteobacterias y las mitocondrias primitivas de los protistas jakobid, ii) dos grupos disjuntos de cianobacterias ( Gloeobacter y un clado que contiene Prochlorococcus y muchos Synechococcus ), y iii) algunos miembros de las betaproteobacterias ( Cupriavidus y algunos Rhodocyclales). [11] [12] Todos producen la misma forma general de dos piezas (piezas aceptoras y codificantes), equivalente a la forma estándar mellada corriente abajo del marco de lectura. Ninguno retiene más de dos pseudonudos en comparación con los cuatro (o más) del tmRNA estándar.
Las alfaproteobacterias tienen dos secuencias distintivas: el reemplazo de la secuencia típica de bucle T TΨCRANY por GGCRGUA y la secuencia AACAGAA en el bucle grande del pseudonudo 3´-terminal. En las mitocondrias, se ha perdido el MLR y una notable repermutación de ssrA mitocondrial da como resultado un pequeño producto de una sola pieza en Jakoba libera . [13]
Las cianobacterias proporcionan el caso más plausible para la evolución de un gen permutado a partir de un gen estándar, debido a las notables similitudes de secuencia entre los dos tipos de genes que aparecen en diferentes cepas de Synechococcus .
La mayoría de los ARNtm se transcriben como precursores más grandes que se procesan de forma muy similar al ARNt . La escisión en el extremo 5' la realiza la ribonucleasa P. [ 4] Varias exonucleasas pueden participar en el procesamiento del extremo 3' del ARNtm, aunque la ARNasa T y la ARNasa PH son las más eficaces. [14] [15] Dependiendo de la especie bacteriana, la 3'-CCA es codificada o añadida por la ARNt nucleotidiltransferasa .
Un procesamiento similar en los sitios internos del ARNtm precursor permutado explica su división física en dos partes. Los ARNtm de dos partes tienen dos extremos adicionales cuyo procesamiento debe tenerse en cuenta. En el caso de las alfaproteobacterias, un extremo 5' es el sitio de inicio de la transcripción sin procesar. [16] El extremo 3' más alejado puede ser, en algunos casos, el resultado de una terminación independiente de rho.


Las estructuras de alta resolución de las moléculas completas de ARNtm no están disponibles actualmente y pueden ser difíciles de obtener debido a la flexibilidad inherente del MLR. En 2007, se obtuvo la estructura cristalina del TLD de Thermus thermophilus unido a la proteína SmpB con una resolución de 3 Å. Esta estructura muestra que SmpB imita el tallo D y el anticodón de un ARNt canónico, mientras que la sección helicoidal 2a del ARNtm corresponde al brazo variable del ARNt. [18] Un estudio de microscopía crioelectrónica del ARNtm en una etapa temprana de la transtraducción muestra la relación espacial entre el ribosoma y el tmRNP (ARNtm unido a la proteína EF-Tu ). El TLD está ubicado cerca del centro asociado a la GTPasa en la subunidad ribosomal 50S; la hélice 5 y los pseudonudos pk2 a pk4 forman un arco alrededor del pico de la subunidad ribosomal 30S. [19]

La codificación por tmRNA fue descubierta en 1995 [20] cuando Simpson y colaboradores sobreexpresaron la citocina de ratón IL-6 en E. coli y encontraron múltiples péptidos derivados de citocinas truncados , cada uno marcado en los extremos carboxilo con la misma extensión de residuo de 11 aminoácidos (A)ANDENYALAA. Con la excepción de la alanina N-terminal , que proviene del extremo 3' del propio tmRNA, esta secuencia de marcado se rastreó hasta un marco de lectura abierto corto en el tmRNA de E. coli . Keiler, et al., reconocieron que el péptido de marcado confiere proteólisis y propusieron el modelo de trans -traducción para la acción del tmRNA. [21]
Aunque los detalles del mecanismo de trans -traducción están bajo investigación, en general se acepta que el ARNtm ocupa primero el sitio A vacío del ribosoma estancado . Posteriormente, el ribosoma se mueve desde el extremo 3' del ARN mensajero truncado hacia el codón de reanudación del MLR, seguido por una etapa propensa al deslizamiento desde donde la traducción continúa normalmente hasta que se encuentra el codón de parada del ARNtm en marco . La trans-traducción es esencial en algunas especies bacterianas, mientras que otras bacterias requieren ARNtm para sobrevivir cuando se someten a condiciones de crecimiento estresantes. [22] Se cree que el ARNtm puede ayudar a la célula con resistencia a los antibióticos al rescatar los ribosomas estancados por los antibióticos. [23] Dependiendo del organismo, el péptido etiqueta puede ser reconocido por una variedad de proteasas o adaptadores de proteasas. [9]

El ssrA es un objetivo para algunos ADN móviles y un pasajero para otros. Se ha descubierto que es interrumpido por tres tipos de elementos móviles. Mediante diferentes estrategias, ninguno de ellos altera la función génica: los intrones del grupo I se eliminan por autoempalme, los elementos palindrómicos rickettsiales (RPE) se insertan en sitios inocuos y las islas genómicas que codifican la integrasa dividen su ssrA objetivo pero restauran la porción escindida. [24] [25] [26] [27]
La ssrA no cromosómica se detectó por primera vez en un estudio genómico de micobacteriófagos (en el 10% de los fagos). [28] Se han encontrado otros elementos móviles, incluidos plásmidos e islas genómicas, que contienen ssrA . Un caso interesante es Rhodobacter sphaeroides ATCC 17025, cuyo gen tmRNA nativo está alterado por una isla genómica; a diferencia de todas las demás islas genómicas en genes tmRNA (o tRNA), esta isla ha inactivado el gen diana nativo sin restauración, pero lo compensa al llevar su propio gen tmRNA. Un pariente muy inusual de ssrA se encuentra en el micobacteriófago lítico DS6A, que codifica poco más que el TLD.
Una forma estructuralmente reducida y codificada por mitocondrias de tmRNA (mt-tmRNA) fue postulada por primera vez para el flagelado jakobid Reclinomonas americana . [11] Posteriormente, se confirmó la presencia de un gen mitocondrial ( ssrA ) que codifica para tmRNA, así como sitios de transcripción y procesamiento de ARN para todos los miembros de jakobids menos uno . [29] [13] La evidencia funcional, es decir, la aminoacilación de mt-tmRNA con alanina , está disponible para Jakoba libera . [13] Más recientemente, ssrA también fue identificado en genomas mitocondriales de oomicetos . [30] Al igual que en α-Proteobacteria (los ancestros de las mitocondrias ), los mt-tmRNA son moléculas de ARN de dos piezas permutadas circularmente, excepto en Jakoba libera donde el gen ha vuelto a codificar una conformación de tmRNA de una pieza. [13]

Los genes tmRNA mitocondriales fueron reconocidos inicialmente como secuencias cortas que se conservan entre los jakobids y que tienen el potencial de plegarse en una estructura secundaria distinta similar a la del ARNt. Con la disponibilidad de nueve secuencias completas de mtDNA de jakobids , [29] y una herramienta de búsqueda de covarianza significativamente mejorada (Infernal; [31] [32] [33] ), se ha desarrollado un modelo de covarianza basado en tmRNA mitocondriales de jakobids , que identificó genes ssrA mitocondriales también en oomicetos . En la actualidad, se han detectado un total de 34 mt-tmRNA de oomicetos en seis géneros: Albugo , Bremia , Phytophthora , Pseudoperonospora , Pythium y Saprolegnia . Un modelo de covarianza construido con secuencias de jakobids y oomicetos ahora está disponible en Rfam bajo el nombre 'mt-tmRNA'. [30]
El ARNtm bacteriano estándar consiste en un dominio similar al ARNt(Ala) (que permite la adición de una alanina no codificada a los ARNm que carecen de una codificación de terminación) y un dominio similar al ARNm que codifica una etiqueta de proteína que destina el polipéptido a la proteólisis. El dominio similar al ARNm se perdió en los ARNtmt. El análisis comparativo de secuencias indica características típicas de los ARNtmt. [30] La más conservada es la secuencia primaria del tallo aceptor de aminoacilo. Esta porción de la molécula tiene un residuo A invariable en la posición discriminadora y un par GU en la posición 3 (excepto en S. eculamonas ecuadoriensis , que tiene un par GC); esta posición es el sitio de reconocimiento para la alanil ARNt sintasa. P2 es una hélice de longitud variable (3 a 10 pares de bases) y corresponde al tallo anticodón de los ARNt, pero sin un bucle anticodón (ya que no es necesario para la función del ARNtm). P2 estabiliza la estructura similar al ARNt, pero cuatro nucleótidos invariantes en oomicetos y jakobids sugieren una función adicional, actualmente no identificada. P3 tiene cinco pares de bases y corresponde al brazo T de los ARNt, aunque con diferentes nucleótidos de consenso tanto en la región emparejada como en el bucle. La secuencia del bucle T se conserva en oomicetos y jakobids , con solo unas pocas desviaciones (p. ej., Saprolegnia ferax ). Finalmente, en lugar del tallo D similar al ARNt con un bucle D acortado de tres nucleótidos característico de los ARNtm bacterianos, las contrapartes mitocondriales tienen un bucle de 5 a 14 nt de longitud altamente variable. La secuencia intermedia (Int.) de los ARNtm de dos piezas es rica en A+U y de longitud irregular (4-34 nt). ). Para ver los modelos de estructura secundaria de los ARNtm de una y dos piezas, consulte la Figura 1.

Los datos de RNA-Seq de Phytophthora sojae muestran un nivel de expresión similar al de los ARNt mitocondriales vecinos , y cuatro sitios de procesamiento principales confirman los extremos predichos del ARNtm maduro. [30] Es probable que la molécula precursora del ARNtm sea procesada por la ARNasa P y una endonucleasa de procesamiento 3' del ARNt (ver Figura 2); se supone que esta última actividad conduce a la eliminación de la secuencia intermedia. Después de la adición de CCA en el nucleótido discriminador 3', el ARNtm puede cargarse mediante la alanil-ARNt sintetasa con alanina.