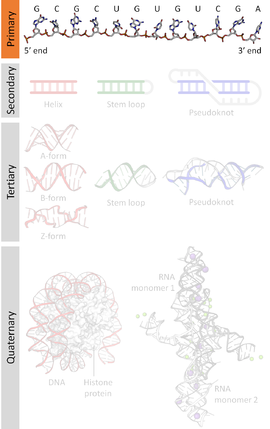

Una secuencia de ácidos nucleicos es una sucesión de bases dentro de los nucleótidos que forman alelos dentro de una molécula de ADN (usando GACT) o ARN (GACU). Esta sucesión se denota por una serie de un conjunto de cinco letras diferentes que indican el orden de los nucleótidos. Por convención, las secuencias generalmente se presentan desde el extremo 5' hasta el extremo 3' . Para el ADN, con su doble hélice, hay dos direcciones posibles para la secuencia anotada; de estas dos, se utiliza la cadena sentido . Debido a que los ácidos nucleicos normalmente son polímeros lineales (no ramificados) , especificar la secuencia es equivalente a definir la estructura covalente de toda la molécula. Por esta razón, la secuencia de ácidos nucleicos también se denomina estructura primaria .
La secuencia representa la información genética . El ácido desoxirribonucleico biológico representa la información que dirige las funciones de un organismo .
Los ácidos nucleicos también tienen una estructura secundaria y una estructura terciaria . A veces se hace referencia erróneamente a la estructura primaria como "secuencia primaria". Sin embargo, no existe un concepto paralelo de secuencia secundaria o terciaria.

Los ácidos nucleicos consisten en una cadena de unidades enlazadas llamadas nucleótidos. Cada nucleótido consta de tres subunidades: un grupo fosfato y un azúcar ( ribosa en el caso del ARN , desoxirribosa en el ADN ) que forman la estructura principal de la cadena de ácido nucleico, y unido al azúcar se encuentra una de un conjunto de nucleobases . Las nucleobases son importantes en el apareamiento de bases de las cadenas para formar estructuras secundarias y terciarias de nivel superior, como la famosa doble hélice .
Las letras posibles son A , C , G y T , que representan las cuatro bases de nucleótidos de una cadena de ADN ( adenina , citosina , guanina y timina ) unidas covalentemente a una cadena principal de fosfodiéster . En el caso típico, las secuencias se imprimen contiguas entre sí sin espacios, como en la secuencia AAAGTCTGAC, que se lee de izquierda a derecha en la dirección 5' a 3' . Con respecto a la transcripción , una secuencia está en la cadena codificante si tiene el mismo orden que el ARN transcrito.
Una secuencia puede ser complementaria a otra secuencia, lo que significa que tienen la base en cada posición en la complementaria (es decir, A a T, C a G) y en el orden inverso. Por ejemplo, la secuencia complementaria a TTAC es GTAA. Si una hebra del ADN bicatenario se considera la hebra sentido, entonces la otra hebra, considerada la hebra antisentido, tendrá la secuencia complementaria a la hebra sentido.
Si bien A, T, C y G representan un nucleótido particular en una posición, también hay letras que representan ambigüedad y que se utilizan cuando más de un tipo de nucleótido podría aparecer en esa posición. Las reglas de la Unión Internacional de Química Pura y Aplicada ( IUPAC ) son las siguientes: [1]
Por ejemplo, W significa que una adenina o una timina podría aparecer en esa posición sin afectar la funcionalidad de la secuencia.
Estos símbolos también son válidos para el ARN, excepto que U (uracilo) reemplaza a T (timina). [1]
Además de la adenina (A), la citosina (C), la guanina (G), la timina (T) y el uracilo (U), el ADN y el ARN también contienen bases que se han modificado después de que se haya formado la cadena de ácido nucleico. En el ADN, la base modificada más común es la 5-metilcitidina (m5C). En el ARN, hay muchas bases modificadas, incluidas la pseudouridina (Ψ), la dihidrouridina (D), la inosina (I), la ribotimidina (rT) y la 7-metilguanosina (m7G). [3] [4] La hipoxantina y la xantina son dos de las muchas bases creadas a través de la presencia de mutágenos , ambas a través de la deaminación (reemplazo del grupo amina con un grupo carbonilo). La hipoxantina se produce a partir de la adenina y la xantina se produce a partir de la guanina . [5] De manera similar, la desaminación de la citosina da como resultado uracilo .
Dadas las dos secuencias de 10 nucleótidos, alinéelas y compare las diferencias entre ellas. Calcule la diferencia porcentual tomando el número de diferencias entre las bases de ADN dividido por el número total de nucleótidos. En este caso hay tres diferencias en la secuencia de 10 nucleótidos. Por lo tanto, hay una diferencia del 30%.
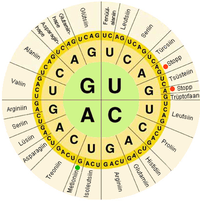
En los sistemas biológicos, los ácidos nucleicos contienen información que una célula viva utiliza para construir proteínas específicas . La secuencia de nucleobases en una cadena de ácido nucleico es traducida por la maquinaria celular en una secuencia de aminoácidos que forman una cadena de proteína. Cada grupo de tres bases, llamado codón , corresponde a un solo aminoácido, y existe un código genético específico por el cual cada posible combinación de tres bases corresponde a un aminoácido específico.
El dogma central de la biología molecular describe el mecanismo por el cual se construyen las proteínas utilizando la información contenida en los ácidos nucleicos. El ADN se transcribe en moléculas de ARNm , que viajan al ribosoma , donde el ARNm se utiliza como plantilla para la construcción de la cadena de proteína. Dado que los ácidos nucleicos pueden unirse a moléculas con secuencias complementarias , existe una distinción entre las secuencias " sentido " que codifican proteínas y la secuencia "antisentido" complementaria, que por sí misma no es funcional, pero puede unirse a la cadena sentido.
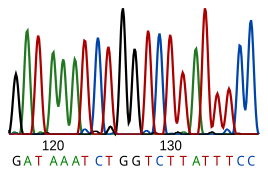
La secuenciación del ADN es el proceso de determinar la secuencia de nucleótidos de un fragmento de ADN determinado . La secuencia del ADN de un ser vivo codifica la información necesaria para que ese ser vivo sobreviva y se reproduzca. Por lo tanto, la determinación de la secuencia es útil en la investigación fundamental sobre por qué y cómo viven los organismos, así como en temas aplicados. Debido a la importancia del ADN para los seres vivos, el conocimiento de una secuencia de ADN puede ser útil en prácticamente cualquier investigación biológica . Por ejemplo, en medicina se puede utilizar para identificar, diagnosticar y potencialmente desarrollar tratamientos para enfermedades genéticas . De manera similar, la investigación sobre patógenos puede conducir a tratamientos para enfermedades contagiosas. La biotecnología es una disciplina floreciente, con potencial para muchos productos y servicios útiles.
El ARN no se secuencia directamente, sino que se copia en ADN mediante la transcriptasa inversa y luego se secuencia este ADN.
Los métodos de secuenciación actuales dependen de la capacidad discriminatoria de las polimerasas de ADN y, por lo tanto, solo pueden distinguir cuatro bases. Una inosina (creada a partir de adenosina durante la edición de ARN ) se lee como una G, y la 5-metilcitosina (creada a partir de citosina por metilación de ADN ) se lee como una C. Con la tecnología actual, es difícil secuenciar pequeñas cantidades de ADN, ya que la señal es demasiado débil para medirla. Esto se soluciona mediante la amplificación por reacción en cadena de la polimerasa (PCR).

Una vez que se obtiene una secuencia de ácido nucleico de un organismo, se almacena in silico en formato digital. Las secuencias genéticas digitales pueden almacenarse en bases de datos de secuencias , analizarse (ver Análisis de secuencias a continuación), modificarse digitalmente y usarse como plantillas para crear nuevo ADN real mediante síntesis genética artificial .
Las secuencias genéticas digitales pueden analizarse utilizando herramientas de la bioinformática para intentar determinar su función.
El ADN del genoma de un organismo puede analizarse para diagnosticar vulnerabilidades a enfermedades hereditarias y también puede utilizarse para determinar la paternidad de un niño (padre genético) o la ascendencia de una persona . Normalmente, cada persona porta dos variaciones de cada gen , una heredada de su madre y la otra heredada de su padre. Se cree que el genoma humano contiene alrededor de 20.000 a 25.000 genes. Además de estudiar los cromosomas a nivel de genes individuales, las pruebas genéticas en un sentido más amplio incluyen pruebas bioquímicas para la posible presencia de enfermedades genéticas o formas mutantes de genes asociados con un mayor riesgo de desarrollar trastornos genéticos.
Las pruebas genéticas identifican cambios en cromosomas, genes o proteínas. [6] Por lo general, las pruebas se utilizan para encontrar cambios asociados con trastornos hereditarios. Los resultados de una prueba genética pueden confirmar o descartar una condición genética sospechada o ayudar a determinar la probabilidad de que una persona desarrolle o transmita un trastorno genético. Actualmente se utilizan cientos de pruebas genéticas y se están desarrollando más. [7] [8]
En bioinformática, un alineamiento de secuencias es una forma de ordenar las secuencias de ADN , ARN o proteínas para identificar regiones de similitud que pueden deberse a relaciones funcionales, estructurales o evolutivas entre las secuencias. [9] Si dos secuencias en un alineamiento comparten un ancestro común, los desajustes se pueden interpretar como mutaciones puntuales y los huecos como mutaciones de inserción o deleción ( indels ) introducidas en uno o ambos linajes en el tiempo desde que divergieron uno del otro. En los alineamientos de secuencias de proteínas, el grado de similitud entre los aminoácidos que ocupan una posición particular en la secuencia se puede interpretar como una medida aproximada de qué tan conservada está una región o motivo de secuencia particular entre linajes. La ausencia de sustituciones, o la presencia de solo sustituciones muy conservadoras (es decir, la sustitución de aminoácidos cuyas cadenas laterales tienen propiedades bioquímicas similares) en una región particular de la secuencia, sugieren [10] que esta región tiene importancia estructural o funcional. Aunque las bases de nucleótidos del ADN y el ARN son más similares entre sí que los aminoácidos, la conservación de pares de bases puede indicar un papel funcional o estructural similar. [11]
La filogenética computacional hace un uso extensivo de los alineamientos de secuencias en la construcción e interpretación de árboles filogenéticos , que se utilizan para clasificar las relaciones evolutivas entre genes homólogos representados en los genomas de especies divergentes. El grado en que las secuencias en un conjunto de consulta difieren está cualitativamente relacionado con la distancia evolutiva de las secuencias entre sí. En términos generales, una alta identidad de secuencia sugiere que las secuencias en cuestión tienen un ancestro común más reciente comparativamente joven , mientras que una baja identidad sugiere que la divergencia es más antigua. Esta aproximación, que refleja la hipótesis del " reloj molecular " de que se puede utilizar una tasa aproximadamente constante de cambio evolutivo para extrapolar el tiempo transcurrido desde que dos genes divergieron por primera vez (es decir, el tiempo de coalescencia ), supone que los efectos de la mutación y la selección son constantes en todos los linajes de secuencias. Por lo tanto, no tiene en cuenta las posibles diferencias entre organismos o especies en las tasas de reparación del ADN o la posible conservación funcional de regiones específicas en una secuencia. (En el caso de las secuencias de nucleótidos, la hipótesis del reloj molecular en su forma más básica también descuenta la diferencia en las tasas de aceptación entre mutaciones silenciosas que no alteran el significado de un codón dado y otras mutaciones que dan como resultado la incorporación de un aminoácido diferente a la proteína). Los métodos estadísticamente más precisos permiten que la tasa evolutiva en cada rama del árbol filogenético varíe, produciendo así mejores estimaciones de los tiempos de coalescencia de los genes.
Con frecuencia, la estructura primaria codifica motivos que son de importancia funcional. Algunos ejemplos de motivos de secuencia son: las cajas C/D [12] y H/ACA [13] de los snoRNA , el sitio de unión Sm encontrado en los ARN espliceosomales como U1 , U2 , U4 , U5 , U6 , U12 y U3 , la secuencia Shine-Dalgarno [14] , la secuencia de consenso Kozak [15] y el terminador de la ARN polimerasa III [16] .
En bioinformática , una entropía de secuencia, también conocida como complejidad de secuencia o perfil de información, [17] es una secuencia numérica que proporciona una medida cuantitativa de la complejidad local de una secuencia de ADN, independientemente de la dirección del procesamiento. Las manipulaciones de los perfiles de información permiten el análisis de las secuencias utilizando técnicas sin alineamiento, como por ejemplo en la detección de motivos y reordenamientos. [17] [18] [19]