La secuenciación unicelular examina la información de la secuencia de ácido nucleico de células individuales con tecnologías de secuenciación optimizadas de próxima generación , proporcionando una mayor resolución de las diferencias celulares y una mejor comprensión de la función de una célula individual en el contexto de su microambiente. [1] Por ejemplo, en el cáncer, la secuenciación del ADN de células individuales puede proporcionar información sobre mutaciones portadas por pequeñas poblaciones de células. En desarrollo, la secuenciación de los ARN expresados por células individuales puede dar información sobre la existencia y el comportamiento de diferentes tipos de células. [2] En los sistemas microbianos, una población de la misma especie puede parecer genéticamente clonal. Aún así, la secuenciación unicelular de ARN o las modificaciones epigenéticas pueden revelar la variabilidad entre células que puede ayudar a las poblaciones a adaptarse rápidamente para sobrevivir en entornos cambiantes. [3]
Una célula humana típica consta de aproximadamente 2 x 3,3 mil millones de pares de bases de ADN y 600 millones de bases de ARNm. Por lo general, se utiliza una mezcla de millones de células para secuenciar el ADN o el ARN utilizando métodos tradicionales como la secuenciación de Sanger o la secuenciación de próxima generación . Mediante la secuenciación profunda del ADN y el ARN de una sola célula, se pueden investigar exhaustivamente las funciones celulares. [1] Al igual que los experimentos típicos de secuenciación de próxima generación, los protocolos de secuenciación unicelular generalmente contienen los siguientes pasos: aislamiento de una sola célula, extracción y amplificación de ácidos nucleicos, preparación de la biblioteca de secuenciación , secuenciación y análisis de datos bioinformáticos . Es más difícil realizar la secuenciación unicelular que la secuenciación de células en masa. La cantidad mínima de materiales de partida de una sola celda hace que la degradación, la pérdida de muestras y la contaminación ejerzan efectos pronunciados en la calidad de los datos de secuenciación. Además, debido al nivel de picogramos de la cantidad de ácidos nucleicos utilizados, [4] a menudo se necesita una amplificación intensa durante la preparación de muestras para la secuenciación unicelular, lo que genera una cobertura desigual, ruido y una cuantificación inexacta de los datos de secuenciación.
Las recientes mejoras técnicas hacen de la secuenciación unicelular una herramienta prometedora para abordar un conjunto de problemas aparentemente inaccesibles. Por ejemplo, mediante la secuenciación unicelular se pueden dilucidar muestras heterogéneas, tipos de células raras, relaciones de linaje celular, mosaicismo de tejidos somáticos, análisis de microbios que no se pueden cultivar y la evolución de enfermedades. [5] La secuenciación unicelular fue seleccionada como el método del año 2013 por Nature Publishing Group. [6]
La secuenciación del genoma del ADN unicelular implica aislar una sola célula, amplificar todo el genoma o la región de interés, construir bibliotecas de secuenciación y luego aplicar la secuenciación del ADN de próxima generación (por ejemplo, Illumina , Ion Torrent , MGI). La secuenciación del ADN unicelular se ha aplicado ampliamente en sistemas de mamíferos para estudiar la fisiología normal y las enfermedades. La resolución unicelular puede descubrir las funciones del mosaicismo genético o la heterogeneidad genética intratumoral en el desarrollo del cáncer o la respuesta al tratamiento. [7] En el contexto de los microbiomas, un genoma de un solo organismo unicelular se conoce como genoma único amplificado (SAG). Los avances en la secuenciación del ADN unicelular han permitido la recopilación de datos genómicos de especies procarióticas no cultivadas presentes en microbiomas complejos. [8] Aunque los SAG se caracterizan por una baja integridad y un sesgo significativo, los avances computacionales recientes han logrado el ensamblaje de genomas casi completos a partir de SAG compuestos. [9] Los datos obtenidos de microorganismos podrían establecer procesos de cultivo en el futuro. [10] Algunas de las herramientas de ensamblaje del genoma utilizadas en la secuenciación unicelular de una sola célula incluyen SPAdes , IDBA-UD, Cortex y HyDA. [11]

Se ha publicado una lista de más de 100 métodos ómicos unicelulares diferentes . [12]
La amplificación por desplazamiento múltiple (MDA) es una técnica ampliamente utilizada que permite amplificar femtogramos de ADN de una bacteria a microgramos para su secuenciación. Los reactivos necesarios para las reacciones de MDA incluyen: cebadores aleatorios y ADN polimerasa del bacteriófago phi29. En una reacción isotérmica de 30 grados, el ADN se amplifica con los reactivos incluidos. A medida que las polimerasas fabrican nuevas cadenas, se produce una reacción de desplazamiento de cadenas, sintetizando múltiples copias de cada ADN molde. Al mismo tiempo, los hilos que se extendieron anteriormente serán desplazados. Los productos MDA tienen una longitud de aproximadamente 12 kb y varían hasta aproximadamente 100 kb, lo que permite su uso en la secuenciación de ADN. [10] En 2017, se introdujo una mejora importante en esta técnica, llamada WGA-X, aprovechando un mutante termoestable de la polimerasa phi29, lo que condujo a una mejor recuperación del genoma de células individuales, en particular aquellas con alto contenido de G+C. . [13] MDA también se ha implementado en un sistema basado en gotas de microfluidos para lograr una amplificación del genoma completo unicelular altamente paralelizada. Al encapsular células individuales en gotitas para la captura y amplificación del ADN, este método ofrece un sesgo reducido y un rendimiento mejorado en comparación con la MDA convencional. [14]
Otro método común es MALBAC . [15] Como se hace en MDA, este método comienza con la amplificación isotérmica, pero los cebadores están flanqueados por una secuencia "común" para la amplificación por PCR posterior. A medida que se generan los amplicones preliminares, la secuencia común promueve la autoligación y la formación de "bucles" para evitar una mayor amplificación. A diferencia de la MDA, la red de ADN altamente ramificada no se forma. En cambio, los bucles se desnaturalizan en otro ciclo de temperatura, lo que permite amplificar los fragmentos con PCR. MALBAC también se implementó en un dispositivo de microfluidos, pero el rendimiento de amplificación no mejoró significativamente mediante la encapsulación en gotas de nanolitros. [dieciséis]
Al comparar MDA y MALBAC, MDA da como resultado una mejor cobertura del genoma, pero MALBAC proporciona una cobertura más uniforme en todo el genoma. MDA podría ser más eficaz para identificar SNP , mientras que se prefiere MALBAC para detectar variantes del número de copias. Si bien realizar MDA con un dispositivo de microfluidos reduce notablemente el sesgo y la contaminación, la química involucrada en MALBAC no demuestra el mismo potencial para mejorar la eficiencia.
Un método particularmente adecuado para el descubrimiento de la variación estructural genómica es la secuenciación de cadenas molde de ADN unicelular (también conocida como Strand-seq). [17] Utilizando el principio de procesamiento tricanal unicelular, que utiliza modelado conjunto de orientación de lectura, profundidad de lectura y fase de haplotipo, Strand-seq permite el descubrimiento del espectro completo de clases de variación estructural somática ≥200 kb en tamaño. Strand-seq supera las limitaciones de los métodos basados en la amplificación del genoma completo para la identificación de clases de variación genética somática en células individuales, [18] porque no es susceptible a las quimeras de lectura que conducen a artefactos de llamada (que se analizan en detalle en la sección siguiente), y es menos afectados por el abandono. La elección del método depende del objetivo de la secuenciación porque cada método presenta diferentes ventajas. [7]
La MDA de genomas de células individuales da como resultado una cobertura del genoma muy desigual, es decir, una representación excesiva e insuficiente relativa de varias regiones de la plantilla, lo que lleva a la pérdida de algunas secuencias. Este proceso tiene dos componentes: a) sobreamplificación y subamplificación estocástica de regiones aleatorias; y b) sesgo sistemático contra regiones con alto porcentaje de GC. El componente estocástico se puede abordar combinando reacciones de MDA unicelulares del mismo tipo de célula, empleando hibridación fluorescente in situ (FISH) y/o confirmación posterior a la secuenciación. [10] El sesgo de MDA contra regiones con alto % de GC se puede abordar mediante el uso de polimerasas termoestables, como en el proceso llamado WGA-X. [13]
Los polimorfismos de un solo nucleótido (SNP), que son una gran parte de la variación genética en el genoma humano , y la variación del número de copias (CNV), plantean problemas en la secuenciación de células individuales, así como la cantidad limitada de ADN extraído de una sola célula. Debido a las escasas cantidades de ADN, el análisis preciso del ADN plantea problemas incluso después de la amplificación, ya que la cobertura es baja y susceptible a errores. Con MDA, la cobertura promedio del genoma es inferior al 80 % y los SNP que no estén cubiertos por las lecturas de secuenciación quedarán excluidos. Además, MDA muestra una alta proporción de abandono de alelos , al no detectar alelos en muestras heterocigotas. Actualmente se utilizan varios algoritmos SNP, pero ninguno es específico de la secuenciación unicelular. MDA con CNV también plantea el problema de identificar CNV falsas que ocultan las CNV reales. Para resolver esto, cuando se pueden generar patrones a partir de CNV falsas, los algoritmos pueden detectar y erradicar este ruido para producir variantes verdaderas. [19]
Strand-seq supera las limitaciones de los métodos basados en la amplificación del genoma completo para la llamada de variantes genéticas: dado que Strand-seq no requiere lecturas (o pares de lecturas) que atraviesen los límites (o puntos de interrupción) de las CNV o clases de variantes estructurales con copia equilibrada, es menos susceptible a artefactos comunes de métodos unicelulares basados en la amplificación del genoma completo, que incluyen abandonos de llamadas de variantes debido a lecturas faltantes en el punto de interrupción de la variante y quimera de lectura. [7] [18] Strand-seq descubre el espectro completo de clases de variación estructural de al menos 200 kb de tamaño, incluidos ciclos de rotura-fusión-puente y eventos de cromotripsis , así como inversiones equilibradas y translocaciones equilibradas o desequilibradas con número de copias. [18] "Las llamadas de variantes estructurales realizadas por Strand-seq se resuelven mediante un haplotipo de longitud de cromosoma , lo que proporciona especificidad adicional de llamada de variantes. [18] Como limitación actual, Strand-seq requiere la división de células para el etiquetado específico de la cadena utilizando bromodesoxiuridina (BrdU ), y el método no detecta variantes de tamaño inferior a 200 kb, como inserciones de elementos móviles .
Los microbiomas se encuentran entre los principales objetivos de la genómica unicelular debido a la dificultad de cultivar la mayoría de los microorganismos en la mayoría de los entornos. La genómica unicelular es una forma poderosa de obtener secuencias de genoma microbiano sin cultivo. Este enfoque se ha aplicado ampliamente en microbiomas marinos, del suelo, del subsuelo, de organismos y de otros tipos para abordar una amplia gama de preguntas relacionadas con la ecología microbiana, la evolución, la salud pública y el potencial de la biotecnología. [20] [21] [22] [23] [24] [25] [26] [27] [28]
La secuenciación del cáncer también es una aplicación emergente de scDNAseq. Los tumores frescos o congelados se pueden analizar y clasificar bastante bien con respecto a SCNA, SNV y reordenamientos utilizando enfoques de ADN de genoma completo. [29] El scDNAseq del cáncer es particularmente útil para examinar la profundidad de la complejidad y las mutaciones compuestas presentes en objetivos terapéuticos amplificados, como los genes del receptor de tirosina quinasa (EGFR, PDGFRA, etc.), donde los enfoques convencionales a nivel de población del tumor en masa no pueden resolver. los patrones de coexistencia de estas mutaciones dentro de células individuales del tumor. Tal superposición puede proporcionar redundancia en la activación de la vía y la resistencia de las células tumorales.

"La secuenciación del metiloma del ADN unicelular cuantifica la metilación del ADN ". Hay varios tipos conocidos de metilación que ocurren en la naturaleza, incluida la 5-metilcitosina (5 mC), la 5-hidroximetilcitosina (5 hmC), la 6-metiladenina (6 mA) y la 4-metilcitosina 4 mC (4 mC). En los eucariotas, especialmente en los animales, el 5mC está muy extendido a lo largo del genoma y desempeña un papel importante en la regulación de la expresión génica mediante la represión de elementos transponibles . [31] La secuenciación de 5 mC en células individuales puede revelar cómo los cambios epigenéticos en células genéticamente idénticas de un solo tejido o población dan lugar a células con diferentes fenotipos.
La secuenciación con bisulfito se ha convertido en el estándar de oro para detectar y secuenciar 5 mC en células individuales. [32] El tratamiento del ADN con bisulfito convierte los residuos de citosina en uracilo, pero no afecta los residuos de 5-metilcitosina. Por tanto, el ADN que ha sido tratado con bisulfito conserva sólo citosinas metiladas. Para obtener la lectura del metiloma, la secuencia tratada con bisulfito se alinea con un genoma no modificado. La secuenciación del genoma completo con bisulfito se logró en células individuales en 2014. [33] El método supera la pérdida de ADN asociada con el procedimiento típico, donde se agregan adaptadores de secuenciación antes de la fragmentación con bisulfito. En cambio, los adaptadores se añaden después de que el ADN se haya tratado y fragmentado con bisulfito, lo que permite amplificar todos los fragmentos mediante PCR. [34] Utilizando secuenciación profunda, este método captura ~40% del total de CpG en cada celda. Con la tecnología existente, el ADN no se puede amplificar antes del tratamiento con bisulfito, ya que la polimerasa no copiará las marcas de 5 mC.
La secuenciación con bisulfito de representación reducida unicelular (scRRBS) es otro método. [35] Este método aprovecha la tendencia de las citosinas metiladas a agruparse en islas CpG (CGI) para enriquecer áreas del genoma con un alto contenido de CpG. Esto reduce el costo de la secuenciación en comparación con la secuenciación con bisulfito del genoma completo, pero limita la cobertura de este método. Cuando se aplica RRBS a muestras masivas, se detecta la mayoría de los sitios CpG en los promotores de genes, pero los sitios en los promotores de genes solo representan el 10% de los sitios CpG en todo el genoma. [36] En células individuales, se detecta el 40% de los sitios CpG de la muestra global. Para aumentar la cobertura, este método también se puede aplicar a un pequeño grupo de células individuales. En una muestra de 20 células individuales agrupadas, se detectó el 63 % de los sitios CpG de la muestra global. La combinación de células individuales es una estrategia para aumentar la cobertura de metiloma, pero a costa de oscurecer la heterogeneidad en la población de células.
Si bien la secuenciación con bisulfito sigue siendo el método más utilizado para la detección de 5 mC, el tratamiento químico es duro y fragmenta y degrada el ADN. Este efecto se exacerba cuando se pasa de muestras masivas a células individuales. Otros métodos para detectar la metilación del ADN incluyen enzimas de restricción sensibles a la metilación. Las enzimas de restricción también permiten la detección de otros tipos de metilación, como 6mA con DpnI. [37] La secuenciación basada en nanoporos también ofrece una ruta para la secuenciación por metilación directa sin fragmentación ni modificación del ADN original. La secuenciación de nanoporos se ha utilizado para secuenciar los metilomas de las bacterias, en los que predominan los 6 mA y 4 mC (a diferencia de los 5 mC en los eucariotas), pero esta técnica aún no se ha reducido a células individuales. [38]
La secuenciación de la metilación del ADN unicelular se ha utilizado ampliamente para explorar diferencias epigenéticas en células genéticamente similares. Para validar estos métodos durante su desarrollo, los datos de metiloma unicelular de una población mixta se clasificaron con éxito mediante agrupación jerárquica para identificar distintos tipos de células. [35] Otra aplicación es el estudio de células individuales durante las primeras divisiones celulares en el desarrollo temprano para comprender cómo surgen diferentes tipos de células de un solo embrión. [39] La secuenciación con bisulfito del genoma completo unicelular también se ha utilizado para estudiar tipos de células raras pero muy activas en el cáncer, como las células tumorales circulantes (CTC). [40]

La secuenciación de cromatina unicelular accesible a transposasa mapea la accesibilidad de la cromatina en todo el genoma. Una transposasa inserta adaptadores de secuenciación directamente en regiones abiertas de cromatina, lo que permite amplificar y secuenciar esas regiones. [41]
Los métodos estándar, como los microarrays y la secuenciación de ARN en masa , analizan la expresión del ARN de grandes poblaciones de células. Estas mediciones pueden ocultar diferencias críticas entre células individuales en poblaciones de células mixtas. [42] [43]
La secuenciación de ARN unicelular (scRNA-seq) proporciona los perfiles de expresión de células individuales y se considera el estándar de oro para definir estados y fenotipos celulares a partir de 2020. [44] Aunque es imposible obtener información completa sobre cada ARN expresado por cada En una célula, debido a la pequeña cantidad de material disponible, los patrones de expresión genética se pueden identificar mediante análisis de agrupación de genes . [45] Esto puede descubrir tipos de células raras dentro de una población de células que quizás nunca antes se hayan visto. Por ejemplo, un grupo de científicos que realizó scRNA-seq en tejido tumoral de neuroblastoma identificó una célula cancerosa de panneuroblastoma rara, que puede resultar atractiva para nuevos enfoques terapéuticos. [46]
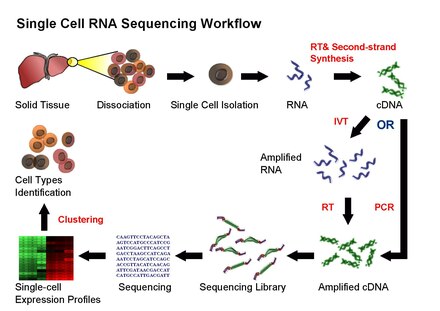
Los protocolos actuales de scRNA-seq implican aislar células individuales y su ARN, y luego seguir los mismos pasos que el RNA-seq en masa: transcripción inversa (RT), amplificación, generación de biblioteca y secuenciación. Los primeros métodos separaban las células individuales en pocillos separados; Los métodos más recientes encapsulan células individuales en gotitas en un dispositivo de microfluidos, donde tiene lugar la reacción de transcripción inversa, convirtiendo los ARN en ADNc. Cada gota lleva un "código de barras" de ADN que etiqueta de forma única los ADNc derivados de una sola célula. Una vez que se completa la transcripción inversa, los ADNc de muchas células se pueden mezclar para la secuenciación, porque las transcripciones de una célula en particular se identifican mediante un código de barras único. [47] [48]
Los desafíos para scRNA-Seq incluyen preservar la abundancia relativa inicial de ARNm en una célula e identificar transcripciones raras. [49] El paso de transcripción inversa es fundamental ya que la eficiencia de la reacción de RT determina qué parte de la población de ARN de la célula será finalmente analizada por el secuenciador. La procesividad de las transcriptasas inversas y las estrategias de cebado utilizadas pueden afectar la producción de ADNc de longitud completa y la generación de bibliotecas sesgadas hacia los extremos 3' o 5' de los genes.
En el paso de amplificación, actualmente se utiliza PCR o transcripción in vitro (IVT) para amplificar el ADNc. Una de las ventajas de los métodos basados en PCR es la capacidad de generar ADNc de longitud completa. Sin embargo, la diferente eficiencia de la PCR en secuencias particulares (por ejemplo, contenido de GC y estructura snapback) también puede amplificarse exponencialmente, produciendo bibliotecas con cobertura desigual. Por otro lado, si bien las bibliotecas generadas por IVT pueden evitar el sesgo de secuencia inducido por la PCR, secuencias específicas pueden transcribirse de manera ineficiente, lo que provoca la pérdida de secuencias o la generación de secuencias incompletas. [1] [42] Se han publicado varios protocolos de scRNA-seq: Tang et al., [50] STRT, [51] SMART-seq, [52] SORT-seq, [53] CEL-seq, [54] RAGE -seq, [55] Cuarzo-seq. [56] y C1-JAULA. [57] Estos protocolos difieren en términos de estrategias para la transcripción inversa, síntesis y amplificación de ADNc, y la posibilidad de acomodar códigos de barras de secuencia específica (es decir, UMI ) o la capacidad de procesar muestras agrupadas. [58]
En 2017, se introdujeron dos enfoques para medir simultáneamente la expresión de proteínas y ARNm de una sola célula a través de anticuerpos marcados con oligonucleótidos conocidos como REAP-seq, [59] y CITE-seq. [60] La recopilación de contenidos celulares después del registro electrofisiológico mediante patch-clamp también ha permitido el desarrollo del método Patch-Seq , que está ganando terreno constantemente en la neurociencia. [61]
Esta plataforma de secuenciación de ARN unicelular permite analizar transcriptomas célula por célula mediante el uso de partición de microfluidos para capturar células individuales y preparar bibliotecas de ADNc de secuenciación de próxima generación (NGS) . [62] La plataforma basada en gotas permite la secuenciación masiva paralela de ARNm en una gran cantidad de células individuales mediante la captura de una sola célula en una gota de aceite. [63]
En general, en una primera etapa las células individuales se capturan por separado y se lisan, luego se realiza la transcripción inversa (RT) del ARNm y se obtiene la biblioteca de ADNc . Para seleccionar el ARNm, la RT se realiza con una secuencia monocatenaria de cebador de desoxitimina (oligo dT) que se une específicamente a la cola poli(A) de las moléculas de ARNm. Posteriormente, la biblioteca de ADNc amplificada se utiliza para la secuenciación. [64]
Entonces, el primer paso del método es la encapsulación de una sola célula y la preparación de la biblioteca. Las células se encapsulan en perlas de gel en emulsión (GEM) gracias a una máquina automática. Para formar estas vesículas, la máquina utiliza un chip de microfluidos y combina todos los componentes con aceite. Cada GEM funcional contiene una sola celda, una sola perla de gel y reactivos RT. En el Gel Bead se unen oligonucleótidos compuestos por 4 partes distintas: cebador de PCR (esencial para la secuenciación); oligonucleótidos con código de barras 10X; Secuencia del Identificador Molecular Único (UMI); Secuencia PolydT (que permite la captura de moléculas de ARNm poliadenilado ). [65] Dentro de cada vesícula de reacción GEM, una sola célula se lisa y se somete a transcripción inversa. El ADNc de una misma célula se identifica gracias a un código de barras común 10X. Además, el número de UMI expresa el nivel de expresión génica y su análisis permite detectar genes muy variables. Esos datos se utilizan a menudo para la clasificación del fenotipo celular o para la identificación de nuevas subpoblaciones. [66]
El último paso de la plataforma es la secuenciación. Las bibliotecas generadas se pueden utilizar directamente para la secuenciación del transcriptoma completo de una sola célula o para flujos de trabajo de secuenciación de objetivos. La secuenciación se realiza utilizando el método de secuenciación de tintes Illumina . Este método de secuenciación se basa en el principio de secuenciación por síntesis (SBS) y el uso de un terminador de colorante reversible que permite la identificación de cada nucleótido individual. Para leer las secuencias de transcripción en un extremo y el código de barras y el UMI en el otro extremo, se requieren lectores de secuenciación de extremos emparejados. [67]
La plataforma basada en gotas permite la detección de tipos de células raras gracias a su alto rendimiento. De hecho, se capturan de 500 a 10.000 células por muestra de una única suspensión celular. El protocolo se realiza fácilmente y permite una alta tasa de recuperación celular de hasta el 65%. El flujo de trabajo global de la plataforma basada en gotas tarda 8 horas y, por tanto, es más rápido que el método basado en Microwell (BD Rhapsody), que tarda 10 horas. Sin embargo, presenta algunas limitaciones como la necesidad de muestras frescas y la detección final de sólo un 10% de ARNm.
La principal diferencia entre el método basado en gotas y el método basado en micropocillos es la técnica utilizada para dividir las células. [64]
La mayoría de los métodos de secuenciación de ARN dependen de la captura de la cola poli (A) para enriquecer el ARNm y agotar el ARNr abundante y poco informativo. Por lo tanto, a menudo se limitan a secuenciar moléculas de ARNm poliadenilado. Sin embargo, estudios recientes están empezando a apreciar la importancia del ARN no poli(A), como el ARN no codificante largo y los microARN, en la regulación de la expresión génica. Small-seq es un método unicelular que captura ARN pequeños (<300 nucleótidos) como microARN, fragmentos de ARNt y ARN nucleolares pequeños en células de mamíferos. [68] Este método utiliza una combinación de “máscaras de oligonucleótidos” (que inhiben la captura de moléculas de ARNr 5.8S muy abundantes) y selección de tamaño para excluir especies de ARN grandes, como otras moléculas de ARNr muy abundantes. Para apuntar a ARN no poli(A) más grandes, como ARNm largo no codificante, ARNm de histonas, ARN circular y ARN potenciador, la selección de tamaño no es aplicable para agotar las moléculas de ARN ribosomal altamente abundantes (ARNr 18S y 28s). [69] RamDA-Seq unicelular es un método que logra esto mediante la realización de transcripción inversa con cebado aleatorio (amplificación por desplazamiento aleatorio) en presencia de cebadores "no tan aleatorios" (NSR) diseñados específicamente para evitar el cebado en la molécula de ARNr. [70] Si bien este método captura con éxito transcripciones de ARN total de longitud completa para la secuenciación y detecta una variedad de ARN no poli(A) con alta sensibilidad, tiene algunas limitaciones. Los cebadores NSR se diseñaron cuidadosamente de acuerdo con las secuencias de ARNr del organismo específico (ratón), y diseñar nuevos conjuntos de cebadores para otras especies requeriría un esfuerzo considerable. Recientemente, un método basado en CRISPR llamado scDASH (agotamiento unicelular de secuencias abundantes por hibridación) demostró otro enfoque para agotar secuencias de ARNr de bibliotecas de secuencias de ARN totales unicelulares. [71]
Actualmente, las bacterias y otros procariotas no son susceptibles a la secuenciación de ARN unicelular debido a la falta de ARNm poliadenilado. Por lo tanto, el desarrollo de métodos de secuenciación de ARN unicelular que no dependan de la captura de la cola poli (A) también será fundamental para permitir estudios de microbioma de resolución unicelular. Los estudios bacterianos en masa suelen aplicar el agotamiento general del ARNr para superar la falta de ARNm poliadenilado en las bacterias, pero a nivel unicelular, el ARN total encontrado en una célula es demasiado pequeño. [69] La falta de ARNm poliadenilado y la escasez de ARN total que se encuentra en células bacterianas individuales son dos barreras importantes que limitan el despliegue de scRNA-seq en bacterias.
scRNA-Seq se está utilizando ampliamente en disciplinas biológicas, incluida la biología del desarrollo , [72] neurología , [73] oncología , [74] [75] [76] inmunología , [77] [78] investigación cardiovascular [79] [80] y Enfermedad infecciosa . [81] [82]
Utilizando métodos de aprendizaje automático , se han utilizado datos de RNA-Seq en masa para aumentar la relación señal/ruido en scRNA-Seq. Específicamente, los científicos han utilizado perfiles de expresión genética de conjuntos de datos pan-cáncer para construir redes de coexpresión y luego los han aplicado en perfiles de expresión genética de células individuales, obteniendo un método más sólido para detectar la presencia de mutaciones en células individuales utilizando niveles de transcripción. [83]
Algunos métodos scRNA-seq también se han aplicado a microorganismos unicelulares. SMART-seq2 se ha utilizado para analizar microbios eucariotas unicelulares, pero como se basa en la captura de la cola poli(A), no se ha aplicado en células procarióticas. [84] Se han utilizado enfoques de microfluidos como Drop-seq y los dispositivos Fluidigm IFC-C1 para secuenciar parásitos individuales de la malaria o células individuales de levadura. [85] [86] El estudio de levadura unicelular buscó caracterizar la tolerancia al estrés heterogéneo en células de levadura isogénicas antes y después de que la levadura se exponga al estrés salino. El análisis unicelular de los diversos factores de transcripción mediante scRNA-seq reveló heterogeneidad en toda la población. Estos resultados sugieren que la regulación varía entre los miembros de una población para aumentar las posibilidades de supervivencia de una fracción de la población.
El primer análisis del transcriptoma unicelular en una especie procariótica se realizó utilizando la enzima exonucleasa terminadora para degradar selectivamente el ARNr y la amplificación por círculo rodante (RCA) del ARNm. [87] En este método, los extremos del ADN monocatenario se ligaron entre sí para formar un círculo y el bucle resultante se utilizó luego como plantilla para la amplificación lineal del ARN. Luego se analizó la biblioteca del producto final mediante microarrays, con bajo sesgo y buena cobertura. Sin embargo, RCA no se ha probado con RNA-seq, que normalmente emplea secuenciación de próxima generación. La secuenciación de ARN unicelular para bacterias sería muy útil para estudiar microbiomas. Abordaría los problemas encontrados en los enfoques convencionales de metatranscriptómica masiva, como no capturar especies presentes en baja abundancia y no resolver la heterogeneidad entre las poblaciones celulares.
scRNA-Seq ha proporcionado información considerable sobre el desarrollo de embriones y organismos, incluido el gusano Caenorhabditis elegans , [88] y la planaria regenerativa Schmidtea mediterranea [89] [90] y el ajolote Ambystoma mexicanum . [91] [92] Los primeros animales vertebrados en ser mapeados de esta manera fueron el pez cebra [93] [94] [95] y Xenopus laevis . [96] En cada caso se estudiaron múltiples etapas del embrión, lo que permitió mapear todo el proceso de desarrollo célula por célula. La ciencia reconoció estos avances como el Avance del Año 2018 . [97]
Se estableció un atlas de células moleculares de testículos de ratones para definir la toxicidad testicular prepuberal inducida por BDE47 utilizando el enfoque ScRNA-seq, lo que proporciona información novedosa sobre nuestra comprensión de los mecanismos y vías subyacentes implicados en la lesión testicular asociada a BDE47 en una resolución unicelular. [98]
Hay varias formas de aislar células individuales antes de la amplificación y secuenciación del genoma completo. La clasificación de células activadas por fluorescencia (FACS) es un enfoque ampliamente utilizado. Las células individuales también se pueden recolectar mediante micromanipulación, por ejemplo mediante dilución en serie o usando una pipeta de parche o nanotubo para recolectar una sola célula. [15] [99] Las ventajas de la micromanipulación son la facilidad y el bajo costo, pero son laboriosas y susceptibles a una identificación errónea de los tipos de células bajo el microscopio. La microdisección por captura láser (LCM) también se puede utilizar para recolectar células individuales. Aunque LCM preserva el conocimiento de la ubicación espacial de una célula muestreada dentro de un tejido, es difícil capturar una sola célula completa sin recolectar también los materiales de las células vecinas. [42] [100] [101] Los métodos de alto rendimiento para el aislamiento de células individuales también incluyen microfluidos . Tanto FACS como los microfluidos son precisos, automáticos y capaces de aislar muestras imparciales. Sin embargo, ambos métodos requieren separar primero las células de sus microambientes, lo que provoca una perturbación en los perfiles transcripcionales en el análisis de expresión de ARN. [102] [103]
En términos generales, para un experimento típico de secuenciación de ARN de células masivas (RNA-seq), se generan diez millones de lecturas y se considera expresado un gen con un umbral superior a 50 lecturas por kb por millón de lecturas (RPKM). Para un gen de 1 kb de largo, esto corresponde a 500 lecturas y un coeficiente de variación (CV) mínimo del 4% bajo el supuesto de la distribución de Poisson . Para una célula de mamífero típica que contiene 200.000 ARNm, es necesario agrupar los datos de secuenciación de al menos 50 células individuales para lograr este valor CV mínimo. Sin embargo, debido a la eficiencia de la transcripción inversa y otros ruidos introducidos en los experimentos, se necesitan más células para realizar análisis de expresión precisos e identificar el tipo de célula. [42]