Mycoplasma laboratorium o Synthia [b 1] se refiere a una cepa sintética de bacteria . El proyecto para construir la nueva bacteria ha evolucionado desde sus inicios. Inicialmente, el objetivo era identificar un conjunto mínimo de genes necesarios para sustentar la vida a partir del genoma de Mycoplasma genitalium y reconstruir estos genes sintéticamente para crear un organismo "nuevo". Inicialmente se eligió como base para este proyecto Mycoplasma genitalium porque en aquel momento tenía el menor número de genes de todos los organismos analizados. Más tarde, la atención se centró en Mycoplasma mycoides y adoptó un enfoque más de prueba y error. [b2]
Para identificar los genes mínimos necesarios para la vida, se eliminó individualmente cada uno de los 482 genes de M. genitalium y se probó la viabilidad de los mutantes resultantes. Esto resultó en la identificación de un conjunto mínimo de 382 genes que teóricamente deberían representar un genoma mínimo. [a 3] En 2008, el conjunto completo de genes de M. genitalium se construyó en el laboratorio con marcas de agua agregadas para identificar los genes como sintéticos. [b 3] [a 4] Sin embargo, M. genitalium crece extremadamente lentamente y se eligió M. mycoides como nuevo foco para acelerar los experimentos destinados a determinar el conjunto de genes realmente necesarios para el crecimiento. [b 4]
En 2010, se sintetizó con éxito el genoma completo de M. mycoides a partir de un registro informático y se trasplantó a una célula existente de Mycoplasma capricolum a la que se le había extraído el ADN. [b 5] Se estima que producir el genoma sintético utilizado para este proyecto costó 40 millones de dólares y 200 años-hombre . [b 4] La nueva bacteria pudo crecer y se llamó JCVI-syn1.0, o Synthia. Después de experimentación adicional para identificar un conjunto más pequeño de genes que podrían producir un organismo funcional, se produjo JCVI-syn3.0, que contiene 473 genes. [b 2] 149 de estos genes tienen una función desconocida. [b 2] Dado que el genoma de JCVI-syn3.0 es nuevo, se considera el primer organismo verdaderamente sintético.
La producción de Synthia es un esfuerzo en biología sintética en el Instituto J. Craig Venter por parte de un equipo de aproximadamente 20 científicos encabezado por el premio Nobel Hamilton Smith y que incluye al investigador de ADN Craig Venter y al microbiólogo Clyde A. Hutchison III . El objetivo general es reducir un organismo vivo a lo esencial y así comprender lo que se requiere para construir un nuevo organismo desde cero. [a 3] El foco inicial fue la bacteria M. genitalium , un parásito intracelular obligado cuyo genoma consta de 482 genes que comprenden 582.970 pares de bases , dispuestos en un cromosoma circular (en el momento en que comenzó el proyecto, este era el genoma más pequeño de todos los conocidos). organismo natural que puede cultivarse en cultivo libre). Utilizaron mutagénesis de transposones para identificar genes que no eran esenciales para el crecimiento del organismo, lo que dio como resultado un conjunto mínimo de 382 genes. [a 3] Este esfuerzo se conoció como Proyecto Genoma Mínimo . [un 5]
Mycoplasma es un género de bacterias de la clase Mollicutes en la división Mycoplasmatota (antes Tenericutes), caracterizada por la falta de pared celular (por lo que es Gram negativa ) debido a su estilo de vida parasitario o comensal . En biología molecular , el género ha recibido mucha atención, tanto por ser un contaminante notoriamente difícil de erradicar en cultivos de células de mamíferos (es inmune a los betalactámicos y otros antibióticos ), [a 6] como por sus usos potenciales como Organismo modelo debido al pequeño tamaño de su genoma. [a 7] La elección del género para el proyecto Synthia se remonta al año 2000, cuando Karl Reich acuñó la frase Mycoplasma laboratorium . [un 2]
En 2005, Pelagibacter ubique (una α-proteobacteria del orden Rickettsiales ) tiene el genoma más pequeño conocido (1.308.759 pares de bases) de cualquier organismo vivo libre y es una de las células autorreplicantes más pequeñas conocidas. Es posiblemente la bacteria más numerosa del mundo (quizás 10 28 células individuales) y, junto con otros miembros del clado SAR11 , se estima que constituye entre un cuarto y la mitad de todas las células bacterianas o arqueas del océano. [a 8] Se identificó en 2002 mediante secuencias de ARNr y se secuenció completamente en 2005. [a 9] Es extremadamente difícil cultivar una especie que no alcanza una alta densidad de crecimiento en cultivo de laboratorio. [a 10] [a 11] Varias especies recientemente descubiertas tienen menos genes que M. genitalium , pero no son de vida libre: muchos genes esenciales faltan en Hodgkinia cicadicola , Sulcia muelleri , Baumannia cicadellinicola (simbiontes de cigarras ) y Carsonella ruddi (simbionte del psílido de la agalla del pecíolo del almez, Pachypsylla venusta [a 12] ) puede estar codificado en el núcleo del huésped. [a 13] El organismo con el conjunto de genes más pequeño conocido en 2013 es Nasuia deltocephalinicola , un simbionte obligado . Tiene sólo 137 genes y un tamaño de genoma de 112 kb. [a 14] [b 6]
Para el proyecto fue necesario desarrollar o adaptar varias técnicas de laboratorio, ya que requería la síntesis y manipulación de fragmentos de ADN de gran tamaño.
En 2007, el equipo de Venter informó que habían logrado transferir el cromosoma de la especie Mycoplasma mycoides a Mycoplasma capricolum mediante:
El término transformación se utiliza para referirse a la inserción de un vector en una célula bacteriana (mediante electroporación o choque térmico). Aquí, el trasplante se utiliza de forma similar al trasplante nuclear .
En 2008, el grupo de Venter describió la producción de un genoma sintético, una copia de la secuencia L43967 de M. genitalium G37, mediante una estrategia jerárquica: [a 16]
El genoma de este resultado de 2008, M. genitalium JCVI-1.0, está publicado en GenBank como CP001621.1. No debe confundirse con los organismos sintéticos posteriores, denominados JCVI-syn, basados en M. mycoides . [un 16]
En 2010, Venter y sus colegas crearon la cepa JCVI-syn1.0 de Mycoplasma mycoides con un genoma sintético. [a 1] Inicialmente la construcción sintética no funcionó, por lo que para identificar el error, que provocó un retraso de 3 meses en todo el proyecto [b 4], se crearon una serie de construcciones semisintéticas. La causa del fracaso fue una única mutación en el marco de lectura en DnaA , un factor de iniciación de la replicación . [un 1]
El propósito de construir una célula con un genoma sintético era probar la metodología, como paso para crear genomas modificados en el futuro. El uso de un genoma natural como plantilla minimizó las posibles fuentes de fracaso. Hay varias diferencias presentes en Mycoplasma mycoides JCVI-syn1.0 en relación con el genoma de referencia, en particular un transposón IS1 de E. coli (una infección de la etapa de 10 kb) y una duplicación de 85 pb, así como elementos necesarios para la propagación en levaduras y residuos de sitios de restricción. [un 1]
Ha habido controversia sobre si JCVI-syn1.0 es un verdadero organismo sintético. Si bien el genoma se sintetizó químicamente en muchas partes, se construyó para que coincidiera estrechamente con el genoma original y se trasplantó al citoplasma de una célula natural. El ADN por sí solo no puede crear una célula viable: se necesitan proteínas y ARN para leer el ADN, y se necesitan membranas lipídicas para compartimentar el ADN y el citoplasma . En JCVI-syn1.0 las dos especies utilizadas como donante y receptor son del mismo género, lo que reduce los posibles problemas de desajustes entre las proteínas del citoplasma del huésped y el nuevo genoma. [a 17] Paul Keim (genetista molecular de la Universidad del Norte de Arizona en Flagstaff ) señaló que "hay grandes desafíos por delante antes de que los ingenieros genéticos puedan mezclar, combinar y diseñar completamente el genoma de un organismo desde cero". [b 4]

Una característica muy publicitada de JCVI-syn1.0 es la presencia de secuencias de marcas de agua. Las 4 marcas de agua (que se muestran en la Figura S1 en el material complementario del artículo [a 1] ) son mensajes codificados escritos en el ADN, de longitud 1246, 1081, 1109 y 1222 pares de bases respectivamente. Estos mensajes no utilizaban el código genético estándar , en el que secuencias de 3 bases de ADN codifican aminoácidos, sino un nuevo código inventado para este fin, que los lectores tenían el desafío de resolver. [b 7] El contenido de las marcas de agua es el siguiente:
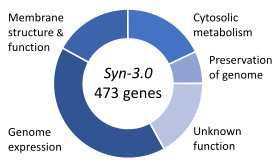
En 2016, el Instituto Venter utilizó genes de JCVI-syn1.0 para sintetizar un genoma más pequeño al que llaman JCVI-syn3.0, que contiene 531.560 pares de bases y 473 genes. [b 8] En 1996, después de comparar M. genitalium con otra pequeña bacteria Haemophilus influenzae , Arcady Mushegian y Eugene Koonin habían propuesto que podría haber un conjunto común de 256 genes que podría ser un conjunto mínimo de genes necesarios para la viabilidad. [b 9] [a 19] En este nuevo organismo, el número de genes sólo puede reducirse a 473, de los cuales 149 tienen funciones completamente desconocidas. [b 9] A partir de 2022, el conjunto desconocido se ha reducido a aproximadamente 100. [b 10] En 2019, se publicó un modelo computacional completo de todas las vías en la célula Syn3.0, que representa el primer modelo in silico completo para un organismo mínimo vivo. . [un 20]
El 6 de octubre de 2007, Craig Venter anunció en una entrevista con el periódico The Guardian del Reino Unido que el mismo equipo había sintetizado químicamente una versión modificada del cromosoma único de Mycoplasma genitalium . El genoma sintetizado aún no había sido trasplantado a una célula funcional. Al día siguiente, el grupo canadiense de bioética ETC Group emitió un comunicado a través de su representante, Pat Mooney , diciendo que la "creación" de Venter era "un chasis sobre el cual se podía construir casi cualquier cosa. Podría ser una contribución a la humanidad, como nuevos medicamentos o una enorme amenaza para la humanidad, como las armas biológicas". Venter comentó: "Estamos tratando con grandes ideas. Estamos tratando de crear un nuevo sistema de valores para la vida. Cuando se trata a esta escala, no se puede esperar que todos sean felices". [b 11]
El 21 de mayo de 2010, Science informó que el grupo Venter había sintetizado con éxito el genoma de la bacteria Mycoplasma mycoides a partir de un registro informático y trasplantado el genoma sintetizado a la célula existente de una bacteria Mycoplasma capricolum a la que se le había eliminado el ADN. La bacteria "sintética" era viable, es decir, capaz de replicarse. [b 1] Venter la describió como "la primera especie... que tiene como padres una computadora". [b 12]
La creación de una nueva bacteria sintética, JCVI-3.0, se anunció en Science el 25 de marzo de 2016. Tiene solo 473 genes. Venter lo llamó "el primer organismo diseñado en la historia" y argumentó que el hecho de que 149 de los genes necesarios tengan funciones desconocidas significa que "a todo el campo de la biología le ha faltado un tercio de lo que es esencial para la vida". [un 21]
El proyecto recibió una gran cobertura por parte de la prensa debido al talento teatral de Venter, hasta el punto de que Jay Keasling , biólogo sintético pionero y fundador de Amyris, comentó que "La única regulación que necesitamos es la de la boca de mi colega". [b 13]
Venter ha argumentado que las bacterias sintéticas son un paso hacia la creación de organismos para fabricar hidrógeno y biocombustibles , y también para absorber dióxido de carbono y otros gases de efecto invernadero . George M. Church , otro pionero en biología sintética , ha expresado la opinión contrastante de que no es necesario crear un genoma completamente sintético ya que E. coli crece más eficientemente que M. genitalium incluso con todo su ADN adicional; Comentó que se han incorporado genes sintéticos a E. coli para realizar algunas de las tareas anteriores. [b 14]
El Instituto J. Craig Venter presentó patentes para el genoma de Mycoplasma laboratorium (el "genoma bacteriano mínimo") en los EE. UU. e internacionalmente en 2006. [b 15] [b 16] [a 22] El grupo ETC, un grupo de bioética canadiense, protestó alegando que el alcance de la patente era demasiado amplio. [b 17]
De 2002 a 2010, un equipo de la Academia de Ciencias de Hungría creó una cepa de Escherichia coli llamada MDS42, que ahora vende Scarab Genomics de Madison, WI con el nombre de "Clean Genome. E.coli", [b 18] donde se eliminó el 15% del genoma de la cepa parental (E. coli K-12 MG1655) para ayudar en la eficiencia de la biología molecular, eliminando elementos IS , pseudogenes y fagos, lo que resultó en un mejor mantenimiento de los genes tóxicos codificados por plásmidos, que a menudo son inactivado por transposones. [a 23] [a 24] [a 25] La bioquímica y la maquinaria de replicación no fueron alteradas.
Además, la genética difícil de estos organismos hace que la verificación posterior de la esencialidad mediante eliminaciones dirigidas sea problemática y prácticamente excluye la posibilidad de realizar una síntesis de novo de 'M. laboratorium', el origen de la atención en la prensa popular.
{{cite journal}}: CS1 maint: multiple names: authors list (link) CS1 maint: numeric names: authors list (link){{cite journal}}: CS1 maint: multiple names: authors list (link) CS1 maint: numeric names: authors list (link){{cite journal}}: CS1 maint: multiple names: authors list (link) CS1 maint: numeric names: authors list (link)