
Una familia de proteínas es un grupo de proteínas relacionadas evolutivamente . En muchos casos, una familia de proteínas tiene una familia de genes correspondiente , en la que cada gen codifica una proteína correspondiente con una relación 1:1. El término "familia de proteínas" no debe confundirse con familia tal como se utiliza en taxonomía.
Las proteínas de una familia descienden de un ancestro común y normalmente tienen estructuras tridimensionales similares , funciones y similitud de secuencia significativa . [1] [2] La similitud de secuencia (generalmente secuencia de aminoácidos) es uno de los indicadores más comunes de homología o ascendencia evolutiva común. [3] [4] Existe un marco bastante bien desarrollado para evaluar la importancia de la similitud entre un grupo de secuencias utilizando métodos de alineación de secuencias . Es poco probable que las proteínas que no comparten un ancestro común muestren una similitud de secuencia estadísticamente significativa, lo que hace que la alineación de secuencias sea una herramienta poderosa para identificar a los miembros de familias de proteínas. [3] [4] Las familias a veces se agrupan en clados más grandes llamados superfamilias basándose en la similitud estructural, incluso si no existe una homología de secuencia identificable.
Actualmente, se han definido más de 60.000 familias de proteínas, [5] aunque la ambigüedad en la definición de "familia de proteínas" lleva a diferentes investigadores a cifras muy variables.
Como ocurre con muchos términos biológicos, el uso de la familia de proteínas depende en cierta medida del contexto; puede indicar grandes grupos de proteínas con el nivel más bajo posible de similitud de secuencia detectable, o grupos muy estrechos de proteínas con secuencia, función y estructura tridimensional casi idénticas, o cualquier tipo de grupo intermedio. Para distinguir entre estas situaciones, el término superfamilia de proteínas se utiliza a menudo para proteínas lejanamente relacionadas cuya relación no es detectable por similitud de secuencia, sino solo por características estructurales compartidas. [6] [7] [8] Estos términos se utilizan jerárquicamente. En el nivel más alto de clasificación se encuentran las superfamilias, que agrupan proteínas lejanamente relacionadas, a menudo basándose en su similitud estructural. [9] Las siguientes superfamilias son familias que agrupan proteínas con un origen evolutivo compartido y tienen una similitud de secuencia significativa . [2] [10] En algunos casos, las proteínas se agrupan además en subfamilias , que son pequeños grupos de proteínas estrechamente relacionadas que a menudo tienen funciones similares o idénticas. [11] Por lo tanto, una superfamilia, como el clan de proteasas PA , tiene una conservación de secuencia mucho menor que una de las familias que contiene, la familia C04.
El concepto de familia de proteínas se concibió cuando se conocían muy pocas estructuras o secuencias de proteínas. En ese momento, la mayoría de las proteínas que se conocían estructuralmente eran proteínas pequeñas de dominio único, como la mioglobina , la hemoglobina y el citocromo c . Desde entonces, se han encontrado muchas proteínas con múltiples unidades o dominios estructurales y funcionales independientes . Debido a la mezcla evolutiva, diferentes dominios de una proteína han evolucionado de forma independiente. Esto ha llevado a centrarse en familias de dominios de proteínas. Varios recursos en línea están dedicados a identificar y catalogar dichos dominios. [12] [13]
Las diferentes regiones de una proteína tienen diferentes limitaciones funcionales (características críticas para la estructura y función de la proteína). Por ejemplo, el sitio activo de una enzima requiere que ciertos residuos de aminoácidos estén orientados con precisión en tres dimensiones. Sin embargo, una interfaz de unión proteína-proteína puede consistir en una gran superficie con limitaciones en la hidrofobicidad o polaridad de los residuos de aminoácidos. Las regiones de proteínas funcionalmente restringidas evolucionan más lentamente que las regiones no restringidas, como los bucles de superficie, dando lugar a bloques discernibles de secuencia conservada cuando se comparan las secuencias de una familia de proteínas (ver alineación de secuencias múltiples ). Estos bloques se denominan más comúnmente motivos, aunque se utilizan muchos otros términos (bloques, firmas, huellas dactilares, etc.). Nuevamente, muchos recursos en línea están dedicados a identificar y catalogar motivos proteicos. [14]
Según el consenso actual, las familias de proteínas surgen de dos maneras. En primer lugar, la separación de una especie parental en dos especies descendientes genéticamente aisladas permite que un gen/proteína acumule de forma independiente variaciones ( mutaciones ) en estos dos linajes. Esto da como resultado una familia de proteínas ortólogas , generalmente con motivos de secuencia conservados. En segundo lugar, la duplicación de un gen puede crear una segunda copia de un gen (denominada parálogo ). Debido a que el gen original aún puede realizar su función, el gen duplicado puede divergir y adquirir nuevas funciones (mediante mutación aleatoria).
Ciertas familias de genes/proteínas, especialmente en eucariotas , sufren expansiones y contracciones extremas en el curso de la evolución, a veces junto con duplicaciones del genoma completo . Las expansiones son menos probables, y las pérdidas más probables, para proteínas intrínsecamente desordenadas y para dominios proteicos cuyos aminoácidos hidrófobos están más alejados del grado óptimo de dispersión a lo largo de la secuencia primaria. [15] Esta expansión y contracción de familias de proteínas es una de las características más destacadas de la evolución del genoma , pero su importancia y ramificaciones no están claras actualmente.
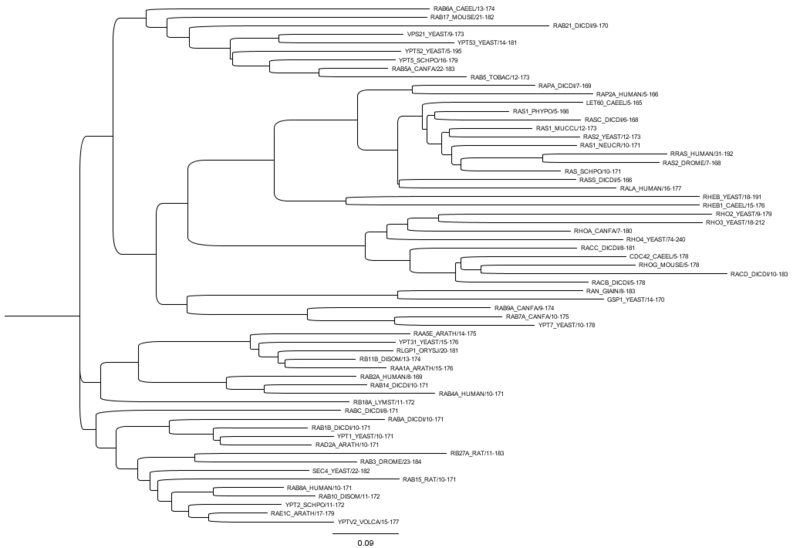
A medida que aumenta el número total de proteínas secuenciadas y se expande el interés en el análisis de proteomas , se está realizando un esfuerzo para organizar las proteínas en familias y describir los dominios y motivos que las componen. La identificación confiable de familias de proteínas es fundamental para el análisis filogenético , la anotación funcional y la exploración de la diversidad de la función de las proteínas en una rama filogenética determinada. La Iniciativa de Función Enzimática utiliza familias y superfamilias de proteínas como base para el desarrollo de una estrategia basada en secuencia/estructura para la asignación funcional a gran escala de enzimas de función desconocida. [16] Los medios algorítmicos para establecer familias de proteínas a gran escala se basan en una noción de similitud.
Muchas bases de datos biológicas registran ejemplos de familias de proteínas y permiten a los usuarios identificar si las proteínas recién identificadas pertenecen a una familia conocida. Aquí están algunos ejemplos:
De manera similar, existen muchos algoritmos de búsqueda en bases de datos, por ejemplo: