La expresión genética espaciotemporal es la activación de genes dentro de tejidos específicos de un organismo en momentos específicos durante el desarrollo . Los patrones de activación genética varían ampliamente en complejidad. Algunos son sencillos y estáticos, como el patrón de tubulina, que se expresa en todas las células en todo momento de la vida. Algunas, por otra parte, son extraordinariamente intrincadas y difíciles de predecir y modelar, y su expresión fluctúa enormemente de un minuto a otro o de una célula a otra. La variación espaciotemporal juega un papel clave en la generación de la diversidad de tipos de células que se encuentran en los organismos desarrollados; Dado que la identidad de una célula está especificada por el conjunto de genes expresados activamente dentro de esa célula, si la expresión génica fuera uniforme espacial y temporalmente, podría haber como máximo un tipo de célula.
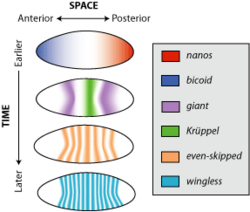
Consideremos el gen sin alas, un miembro de la familia de genes wnt . En el desarrollo embrionario temprano del organismo modelo Drosophila melanogaster , o mosca de la fruta, sin alas se expresa en casi todo el embrión en franjas alternas de tres células separadas. Este patrón se pierde cuando el organismo se convierte en larva, pero la ausencia de alas todavía se expresa en una variedad de tejidos, como los discos imaginales del ala , parches de tejido que se desarrollarán hasta convertirse en las alas adultas. El patrón espaciotemporal de la expresión del gen sin alas está determinado por una red de interacciones reguladoras que consisten en los efectos de muchos genes diferentes, como Even-skipped y Krüppel.
¿Qué causa las diferencias espaciales y temporales en la expresión de un solo gen? Debido a que los patrones de expresión actuales dependen estrictamente de patrones de expresión anteriores, existe un problema regresivo a la hora de explicar qué causó las primeras diferencias en la expresión genética. El proceso por el cual la expresión genética uniforme se vuelve espacial y temporalmente diferencial se conoce como ruptura de simetría . Por ejemplo, en el caso del desarrollo embrionario de Drosophila , los genes nanos y bicoides se expresan asimétricamente en el ovocito porque las células maternas depositan ARN mensajero (ARNm) para estos genes en los polos del óvulo antes de su puesta .

Una forma de identificar el patrón de expresión de un gen particular es colocar un gen indicador aguas abajo de su promotor. En esta configuración, el gen promotor hará que el gen indicador se exprese sólo donde y cuando se exprese el gen de interés. La distribución de la expresión del gen indicador se puede determinar visualizándolo. Por ejemplo, la proteína fluorescente verde del gen informador se puede visualizar estimulándola con luz azul y luego usando una cámara digital para registrar la emisión fluorescente verde .
Si se desconoce el promotor del gen de interés, existen varias formas de identificar su distribución espaciotemporal. La inmunohistoquímica implica preparar un anticuerpo con afinidad específica por la proteína asociada con el gen de interés. Esta distribución de este anticuerpo se puede visualizar mediante una técnica como el marcaje fluorescente. La inmunohistoquímica tiene las ventajas de ser metodológicamente factible y relativamente económica. Sus desventajas incluyen la falta de especificidad del anticuerpo, lo que conduce a una identificación de expresión falsamente positiva . Una mala penetrancia del anticuerpo en el tejido diana puede provocar resultados falsos negativos . Además, dado que la inmunohistoquímica visualiza la proteína generada por el gen, si el producto proteico se difunde entre las células o tiene una vida media particularmente corta o larga en relación con el ARNm que se utiliza para traducir la proteína, esto puede llevar a una interpretación distorsionada de la cual Las células expresan el ARNm .

La hibridación in situ es un método alternativo en el que se añade al tejido una "sonda", un ácido nucleico sintético con una secuencia complementaria al ARNm del gen. Luego, esta sonda se etiqueta químicamente para poder visualizarla más tarde. Esta técnica permite la visualización específicamente de células productoras de ARNm sin ninguno de los artefactos asociados con la inmunohistoquímica. Sin embargo, es muy difícil y requiere conocimiento de la secuencia de ADN correspondiente al gen de interés.
Un método llamado cribado de trampa potenciadora revela la diversidad de patrones de expresión génica espaciotemporal posibles en un organismo. En esta técnica, el ADN que codifica un gen informador se inserta aleatoriamente en el genoma. Dependiendo de los promotores del gen próximos al punto de inserción, el gen indicador se expresará en tejidos particulares en puntos particulares del desarrollo. Si bien los patrones de expresión derivados de la trampa potenciadora no reflejan necesariamente los patrones reales de expresión de genes específicos, revelan la variedad de patrones espaciotemporales que son accesibles a la evolución.
Los genes indicadores se pueden visualizar en organismos vivos, pero tanto la inmunohistoquímica como la hibridación in situ deben realizarse en tejidos fijos . Las técnicas que requieren la fijación de tejido solo pueden generar un único punto temporal por organismo individual. Sin embargo, utilizar animales vivos en lugar de tejido fijo puede ser crucial para comprender dinámicamente los patrones de expresión a lo largo de la vida de un individuo. De cualquier manera, la variación entre individuos puede confundir la interpretación de los patrones de expresión temporal.
Se están buscando varios métodos para controlar la expresión genética espacial, temporal y en diferentes grados. Un método consiste en utilizar un sistema inductor/represor de operones que proporciona control temporal de la expresión génica. Para controlar espacialmente la expresión genética, se están desarrollando impresoras de inyección de tinta para imprimir ligandos en cultivos en gel. [1] Otro método popular implica el uso de luz para controlar la expresión genética en forma espaciotemporal. Dado que la luz también puede controlarse fácilmente en el espacio, el tiempo y el grado, se han desarrollado y están en estudio varios métodos para controlar la expresión genética a nivel de ADN y ARN [2] . Por ejemplo, la interferencia del ARN se puede controlar usando luz [3] [4] y también se ha realizado un patrón de expresión génica en monocapa celular [5] y en embriones de pez cebra usando morfolino enjaulado [6] o ácido nucleico peptídico [7] [8 ] [9] que demuestra el control espaciotemporal de la expresión genética. Recientemente se ha demostrado un control basado en luz a nivel de ADN utilizando un sistema basado en transgenes [10] o oligos formadores de triplex enjaulados [11].