La química combinatoria comprende métodos de síntesis química que permiten preparar una gran cantidad (de decenas a miles o incluso millones) de compuestos en un solo proceso. Estas bibliotecas de compuestos se pueden crear como mezclas, conjuntos de compuestos individuales o estructuras químicas generadas por software de computadora. [1] La química combinatoria se puede utilizar para la síntesis de moléculas pequeñas y de péptidos.
Las estrategias que permiten la identificación de componentes útiles de las bibliotecas también forman parte de la química combinatoria. Los métodos utilizados en la química combinatoria también se aplican fuera de la química.
La química combinatoria fue inventada por Furka Á (de la Universidad Eötvös Loránd en Budapest, Hungría) quien describió el principio de la misma, la síntesis combinatoria y un procedimiento de deconvolución en un documento certificado ante notario en 1982. [2] El principio del método combinatorio es : sintetizar una mezcla de compuestos de múltiples componentes (biblioteca combinatoria) en un solo procedimiento paso a paso y seleccionarla para encontrar candidatos a fármacos u otros tipos de compuestos útiles también en un solo proceso. La innovación más importante del método combinatorio es el uso de mezclas en la síntesis y cribado que aseguran la alta productividad del proceso. Las motivaciones que llevaron a la invención se publicaron en 2002. [3]
La síntesis de moléculas de forma combinatoria puede conducir rápidamente a un gran número de moléculas. Por ejemplo, una molécula con tres puntos de diversidad ( R 1 , R 2 y R 3 ) puede generar posibles estructuras, donde , y son los números de diferentes sustituyentes utilizados. [2]
El principio básico de la química combinatoria es preparar bibliotecas de una gran cantidad de compuestos y luego identificar los componentes útiles de las bibliotecas.
Aunque la química combinatoria sólo ha sido adoptada por la industria desde la década de 1990, [4] sus raíces se remontan a la década de 1960, cuando un investigador de la Universidad Rockefeller , Bruce Merrifield , comenzó a investigar la síntesis de péptidos en fase sólida .
En su forma moderna, la química combinatoria probablemente haya tenido su mayor impacto en la industria farmacéutica . [5] Los investigadores que intentan optimizar el perfil de actividad de un compuesto crean una " biblioteca " de muchos compuestos diferentes pero relacionados. [6] [7] Los avances en robótica han llevado a un enfoque industrial para la síntesis combinatoria, lo que permite a las empresas producir de forma rutinaria más de 100.000 compuestos nuevos y únicos por año. [8]
Para manejar la gran cantidad de posibilidades estructurales, los investigadores suelen crear una "biblioteca virtual", una enumeración computacional de todas las estructuras posibles de un farmacóforo determinado con todos los reactivos disponibles . [9] Una biblioteca de este tipo puede constar de miles a millones de compuestos "virtuales". El investigador seleccionará un subconjunto de la 'biblioteca virtual' para la síntesis real, basándose en varios cálculos y criterios (ver ADME , química computacional y QSAR ).

La síntesis combinatoria de mezcla dividida (dividida y combinada) [10] [11] se basa en la síntesis en fase sólida desarrollada por Merrifield . [12] Si se sintetiza una biblioteca combinatoria de péptidos utilizando 20 aminoácidos (u otros tipos de componentes básicos), el soporte sólido en forma de perla se divide en 20 porciones iguales. A esto le sigue el acoplamiento de un aminoácido diferente a cada porción. El tercer paso es mezclar todas las porciones. Estos tres pasos componen un ciclo. El alargamiento de las cadenas peptídicas se puede realizar simplemente repitiendo los pasos del ciclo.

El procedimiento se ilustra mediante la síntesis de una biblioteca de dipéptidos utilizando los mismos tres aminoácidos como componentes básicos en ambos ciclos. Cada componente de esta biblioteca contiene dos aminoácidos dispuestos en diferentes órdenes. Los aminoácidos utilizados en los acoplamientos están representados por círculos amarillos, azules y rojos en la figura. Las flechas divergentes muestran la división de la resina de soporte sólido (círculos verdes) en porciones iguales, las flechas verticales significan acoplamiento y las flechas convergentes representan mezclar y homogeneizar las porciones del soporte.
La figura muestra que en los dos ciclos sintéticos se forman 9 dipéptidos. En el tercer y cuarto ciclo se formarían 27 tripéptidos y 81 tetrapéptidos, respectivamente.
La "síntesis split-mix" tiene varias características destacadas:
En 1990 tres grupos describieron métodos para preparar bibliotecas de péptidos mediante métodos biológicos [13] [14] [15] y un año después Fodor et al. publicó un método notable para la síntesis de matrices de péptidos en pequeños portaobjetos de vidrio. [dieciséis]
Mario Geysen y sus colegas desarrollaron un método de "síntesis paralela" para la preparación de matrices de péptidos. [17] Sintetizaron 96 péptidos en varillas de plástico (alfileres) recubiertas en sus extremos con un soporte sólido. Las clavijas se sumergieron en la solución de reactivos colocada en los pocillos de una placa de microtitulación . El método se aplica ampliamente, particularmente mediante el uso de sintetizadores paralelos automáticos. Aunque el método paralelo es mucho más lento que el combinatorio real, su ventaja es que se sabe exactamente qué péptido u otro compuesto se forma en cada pin.
Se desarrollaron procedimientos adicionales para combinar las ventajas de la mezcla dividida y la síntesis paralela. En el método descrito por dos grupos [18] [19] el soporte sólido se encerró en cápsulas de plástico permeables junto con una etiqueta de radiofrecuencia que llevaba el código del compuesto que se formaría en la cápsula. El procedimiento se llevó a cabo de manera similar al método de mezcla dividida. Sin embargo, en la etapa dividida, las cápsulas se distribuyeron entre los recipientes de reacción según los códigos leídos en las etiquetas de radiofrecuencia de las cápsulas.
Furka et al. desarrollaron un método diferente para el mismo propósito. [20] se denomina "síntesis de cadenas". En este método, las cápsulas no llevaban ningún código. Se ensartan como las perlas de un collar y se colocan en forma de hilo en los recipientes de reacción. La identidad de las cápsulas, así como su contenido, se almacenan por la posición que ocupan en las cuerdas. Después de cada paso de acoplamiento, las cápsulas se redistribuyen entre nuevas cadenas según reglas definidas.
En el proceso de descubrimiento de fármacos, la síntesis y evaluación biológica de pequeñas moléculas de interés suele ser un proceso largo y laborioso. La química combinatoria ha surgido en las últimas décadas como un enfoque para sintetizar rápida y eficientemente un gran número de posibles fármacos de molécula pequeña. En una síntesis típica, solo se produce una molécula objetivo al final de un esquema sintético, y cada paso de una síntesis produce un solo producto. En una síntesis combinatoria , cuando se utiliza un solo material de partida, es posible sintetizar una gran biblioteca de moléculas utilizando condiciones de reacción idénticas que luego pueden analizarse para determinar su actividad biológica . Luego, este conjunto de productos se divide en tres porciones iguales que contienen cada uno de los tres productos, y luego cada uno de los tres conjuntos individuales reacciona con otra unidad de reactivo B, C o D, produciendo 9 compuestos únicos a partir de los 3 anteriores. Luego, este proceso se repite hasta que se agrega la cantidad deseada de bloques de construcción, generando muchos compuestos. Al sintetizar una biblioteca de compuestos mediante una síntesis de varios pasos, se deben emplear métodos de reacción eficientes y, si se utilizan métodos de purificación tradicionales después de cada paso de reacción, los rendimientos y la eficiencia se verán afectados.
La síntesis en fase sólida ofrece soluciones potenciales para obviar la necesidad de los pasos típicos de enfriamiento y purificación que se utilizan a menudo en la química sintética. En general, una molécula de partida se adhiere a un soporte sólido (típicamente un polímero insoluble), luego se realizan reacciones adicionales y el producto final se purifica y luego se escinde del soporte sólido. Dado que las moléculas de interés están unidas a un soporte sólido, es posible reducir la purificación después de cada reacción a un solo paso de filtración/lavado, eliminando la necesidad de tediosos pasos de extracción líquido-líquido y evaporación de solvente que implica la mayoría de la química sintética. Además, al utilizar reactivos heterogéneos, se puede utilizar un exceso de reactivos para llevar a cabo reacciones lentas hasta su finalización, lo que puede mejorar aún más los rendimientos. El exceso de reactivos puede eliminarse simplemente por lavado sin necesidad de pasos de purificación adicionales como la cromatografía .

A lo largo de los años, se han desarrollado una variedad de métodos para perfeccionar el uso de la síntesis orgánica en fase sólida en la química combinatoria, incluidos esfuerzos para aumentar la facilidad de síntesis y purificación, así como métodos no tradicionales para caracterizar productos intermedios. Aunque la mayoría de los ejemplos descritos aquí emplearán medios de reacción heterogéneos en cada paso de reacción, Booth y Hodges proporcionan un ejemplo temprano del uso de reactivos soportados en sólidos solo durante el paso de purificación de las síntesis tradicionales en fase de solución. [21] En su opinión, la química en fase de solución ofrece las ventajas de evitar las reacciones de unión y escisión necesarias para anclar y eliminar moléculas de las resinas, así como de eliminar la necesidad de recrear análogos en fase sólida de reacciones en fase de solución establecidas.
El único paso de purificación al final de una síntesis permite eliminar una o más impurezas, suponiendo que se conozca la estructura química de la impureza ofensiva. Si bien el uso de reactivos soportados en sólidos simplifica enormemente la síntesis de compuestos, muchas síntesis combinatorias requieren múltiples pasos, cada uno de los cuales aún requiere alguna forma de purificación. Armstrong, et al. describen un método de un solo recipiente para generar bibliotecas combinatorias, llamadas condensaciones de múltiples componentes (MCC). [22] En este esquema, tres o más reactivos reaccionan de manera que cada reactivo se incorpora al producto final en un solo paso, eliminando la necesidad de una síntesis de múltiples pasos que involucra muchos pasos de purificación. En los MCC, no se requiere deconvolución para determinar qué compuestos son biológicamente activos porque cada síntesis en una matriz tiene un solo producto, por lo que la identidad del compuesto debe conocerse de manera inequívoca.
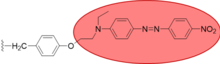
En otra síntesis de matriz, Still generó una gran biblioteca de oligopéptidos mediante síntesis dividida. [23] El inconveniente de fabricar miles de compuestos es que es difícil determinar la estructura de los compuestos formados. Su solución es utilizar etiquetas moleculares, en las que se adhiere una pequeña cantidad (1 pmol/cuenta) de un tinte a las cuentas, y la identidad de una determinada cuenta se puede determinar analizando qué etiquetas están presentes en la cuenta. A pesar de lo fácil que resulta la identificación de los receptores mediante la fijación de etiquetas, sería bastante imposible analizar individualmente cada compuesto para determinar su capacidad de unión al receptor, por lo que se adjuntó un tinte a cada receptor, de modo que sólo aquellos receptores que se unen a su sustrato produzcan un cambio de color.
Cuando es necesario ejecutar muchas reacciones en una matriz (como las 96 reacciones descritas en una de las matrices MCC de Armstrong), algunos de los aspectos más tediosos de la síntesis se pueden automatizar para mejorar la eficiencia. Este trabajo, el "método DIVERSOMER", fue iniciado en Parke-Davis a principios de la década de 1990 para ejecutar hasta 40 reacciones químicas en paralelo. Estos esfuerzos llevaron al primer equipo disponible comercialmente para química combinatoria (el sintetizador Diversómero vendido por Chemglass) y al primer uso de robótica de manipulación de líquidos dentro de un laboratorio de química. [24] [25] Este método utiliza un dispositivo que automatiza los ciclos de carga y lavado de resina, así como el monitoreo y la purificación del ciclo de reacción, y demuestra la viabilidad de su método y aparato usándolo para sintetizar una variedad de clases de moléculas. como hidantoínas y benzodiazepinas , ejecutando 8 o 40 reacciones individuales en paralelo. Este y varios otros esfuerzos pioneros en química combinatoria se presentaron como artículos "clásicos" en este campo en 1999. [26]
A menudo, no es posible utilizar equipos costosos y Schwabacher, et al. Describe un método simple para combinar la síntesis paralela de miembros de la biblioteca y la evaluación de bibliotecas completas de compuestos. [27] En su método, un hilo que se divide en diferentes regiones se enrolla alrededor de un cilindro, donde luego se acopla un reactivo diferente a cada región que contiene una sola especie. Luego, el hilo se vuelve a dividir y se enrolla alrededor de un cilindro de diferente tamaño, y luego se repite este proceso. La belleza de este método es que la identidad de cada producto puede conocerse simplemente por su ubicación a lo largo del hilo, y la actividad biológica correspondiente se identifica mediante la transformación de Fourier de señales de fluorescencia.
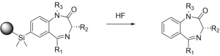
En la mayoría de las síntesis descritas aquí, es necesario unir y retirar el reactivo de partida a/de un soporte sólido. Esto puede conducir a la generación de un grupo hidroxilo, que potencialmente puede afectar la actividad biológica de un compuesto objetivo. Ellman utiliza soportes de fase sólida en un esquema de síntesis de múltiples pasos para obtener 192 derivados individuales de 1,4-benzodiazepina, que son agentes terapéuticos bien conocidos. [28] Para eliminar la posibilidad de una posible interferencia del grupo hidroxilo, se utiliza un método novedoso que utiliza la química de silil-arilo para unir las moléculas al soporte sólido que se escinde del soporte y no deja rastro del conector.

Al anclar una molécula a un soporte sólido, los intermediarios no se pueden aislar entre sí sin escindir la molécula de la resina. Dado que muchas de las técnicas de caracterización tradicionales utilizadas para rastrear el progreso de la reacción y confirmar la estructura del producto se basan en soluciones, se deben utilizar diferentes técnicas. Se han utilizado espectroscopia de RMN 13 C en fase de gel, espectrometría de masas MALDI y espectroscopia IR para confirmar la estructura y monitorear el progreso de las reacciones en fase sólida. [29] Gordon et al., describen varios estudios de casos que utilizan iminas y peptidilfosfonatos para generar bibliotecas combinatorias de moléculas pequeñas. [29] Para generar la biblioteca de iminas, se hace reaccionar un aminoácido unido a una resina en presencia de un aldehído. Los autores demuestran el uso de espectroscopia de RMN en fase de gel de 13 C rápida y espectroscopia de RMN de 1 H con giro de ángulo mágico para monitorear el progreso de las reacciones y demostraron que la mayoría de las iminas se podían formar en tan solo 10 minutos a temperatura ambiente cuando se usaba ortoformiato de trimetilo como el disolvente. Luego, las iminas formadas se derivatizaron para generar 4-tiazolidinonas, B-lactámicos y pirrolidinas.
El uso de soportes en fase sólida simplifica enormemente la síntesis de grandes bibliotecas combinatorias de compuestos. Esto se hace anclando un material de partida a un soporte sólido y luego ejecutando reacciones posteriores hasta que se construye una biblioteca suficientemente grande, después de lo cual los productos se escinden del soporte. También se ha demostrado el uso de la purificación en fase sólida para su uso en esquemas de síntesis en fase de solución junto con técnicas de purificación por extracción líquido-líquido estándar.
Las bibliotecas combinatorias son mezclas especiales de múltiples componentes de compuestos químicos de moléculas pequeñas que se sintetizan en un solo proceso paso a paso. Se diferencian de una colección de compuestos individuales, así como de una serie de compuestos preparados mediante síntesis paralela. Es una característica importante que se utilicen mezclas en su síntesis. El uso de mezclas garantiza una altísima eficiencia del proceso. Ambos reactivos pueden ser mezclas y en este caso el procedimiento sería aún más eficiente. Sin embargo, por razones prácticas, es aconsejable utilizar el método de mezcla dividida, en el que una de dos mezclas se reemplaza por bloques de construcción individuales (BB). Las mezclas son tan importantes que no existen bibliotecas combinatorias sin utilizar mezcla en la síntesis, y si se utiliza una mezcla en un proceso inevitablemente se forman bibliotecas combinatorias. La síntesis de mezcla dividida generalmente se realiza utilizando un soporte sólido, pero también es posible aplicarla en solución. Dado que él estructura los componentes son desconocidos, es necesario utilizar métodos de deconvolución en la detección. Una de las características más importantes de las bibliotecas combinatorias es que toda la mezcla se puede cribar en un solo proceso. Esto hace que estas bibliotecas sean muy útiles en la investigación farmacéutica. También se pueden sintetizar bibliotecas parciales de bibliotecas combinatorias completas. Algunos de ellos se pueden utilizar en deconvolución [30]
Si las moléculas sintetizadas de una biblioteca combinatoria se escinden del soporte sólido, se forma una mezcla soluble. En dicha solución se pueden encontrar millones de compuestos diferentes. Cuando se desarrolló este método sintético, al principio parecía imposible identificar las moléculas y encontrar moléculas con propiedades útiles. Sin embargo, se han desarrollado estrategias para identificar los componentes útiles para resolver el problema. Todas estas estrategias se basan en la síntesis y prueba de bibliotecas parciales. La estrategia iterativa más antigua se describe en el documento de Furka antes mencionado, certificado ante notario en 1982 y. [2] El método fue publicado posteriormente de forma independiente por Erb et al. bajo el nombre "Deconvolución recursiva" [31]
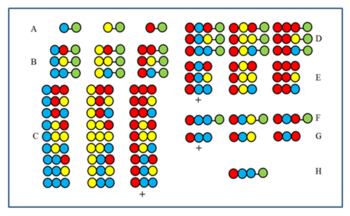
El método se explica mediante la figura. Se sintetiza una biblioteca de péptidos de 27 miembros a partir de tres aminoácidos. Después del primer (A) y segundo (B) ciclos, las muestras se reservaron antes de mezclarlas. Los productos del tercer ciclo (C) se escinden antes de mezclarlos y luego se prueba su actividad. Supongamos que el grupo etiquetado con el signo + está activo. Todos los miembros tienen el aminoácido rojo en la última posición de acoplamiento (CP). En consecuencia, el miembro activo también tiene el aminoácido rojo en el último CP. Luego, el aminoácido rojo se acopla a las tres muestras reservadas después del segundo ciclo (B) para obtener las muestras D. Después de la escisión, se forman las tres muestras E. Si después de realizar la prueba la muestra marcada con + es la activa muestra que el aminoácido azul ocupa el segundo CP en el componente activo. Luego, a las tres muestras A, primero se acopla el aminoácido azul y luego el rojo (F) y luego se analiza nuevamente después de la escisión (G). Si el componente + resulta estar activo, la secuencia del componente activo se determina y se muestra en H.
El escaneo posicional fue introducido de forma independiente por Furka et al. [32] y Pinilla et al. [33] El método se basa en la síntesis y prueba de series de subbibliotecas. en el que una determinada posición de la secuencia está ocupada por el mismo aminoácido. La figura muestra las nueve subbibliotecas (B1-D3) de una biblioteca de trímeros peptídicos completos (A) elaborada a partir de tres aminoácidos. En las subbibliotecas hay una posición que ocupa el mismo aminoácido en todos los componentes. En la síntesis de una subbiblioteca el soporte no se divide y sólo se acopla un aminoácido a toda la muestra. Como resultado, una posición está realmente ocupada por el mismo aminoácido en todos los componentes. Por ejemplo, en la subbiblioteca B2, la posición 2 está ocupada por el aminoácido "amarillo" en los nueve componentes. Si en una prueba de detección esta subbiblioteca da una respuesta positiva, significa que la posición 2 en el péptido activo también está ocupada por el aminoácido "amarillo". La secuencia de aminoácidos se puede determinar analizando las nueve (o en ocasiones menos) subbibliotecas.

Por omisión de bibliotecas [34] [35] , falta un determinado aminoácido en todos los péptidos de la mezcla. La figura muestra la biblioteca completa y las tres bibliotecas omitidas. En la parte superior se muestran los aminoácidos omitidos. Si la biblioteca de omisiones da un resultado negativo, el aminoácido omitido está presente en el componente activo.
Si los péptidos no se escinden del soporte sólido, se trata de una mezcla de perlas, conteniendo cada perla un único péptido. Smith y sus colegas [36] demostraron anteriormente que los péptidos también podían probarse en forma ligada. Este enfoque también se utilizó en la selección de bibliotecas de péptidos. La biblioteca de péptidos unidos se probó con una proteína diana disuelta. Se seleccionaron las perlas a las que estaba unida la proteína, se eliminó la proteína de la perla y luego se identificó el péptido unido mediante secuenciación. Taylor y Morken siguieron un enfoque algo diferente. [37] Utilizaron termografía infrarroja para identificar catalizadores en bibliotecas no unidas a péptidos. El método se basa en el calor que se desprende de las perlas que contienen un catalizador cuando la biblioteca atada se sumerge en una solución de un sustrato. Cuando las perlas se examinan a través de un microscopio de infrarrojos, las perlas que contienen el catalizador aparecen como puntos brillantes y se pueden distinguir.
Si se trata de una biblioteca orgánica no peptídica, no es tan sencillo determinar la identidad del contenido de una perla como en el caso de una de péptidos. Para superar esta dificultad, se han desarrollado métodos para unir a las perlas, en paralelo con la síntesis de la biblioteca, moléculas que codifican la estructura del compuesto formado en la perla. Ohlmeyer y sus colegas publicaron un método de codificación binaria [38] . Utilizaron mezclas de 18 moléculas marcadas que, después de separarlas de las cuentas, podían identificarse mediante cromatografía de gases por captura de electrones. Sarkar et al. describieron oligómeros quirales de amidas pentenoicas (COPA) que pueden usarse para construir bibliotecas OBOC codificadas en masa. [39] Kerr y cols. introdujo un método de codificación innovador [40] Se adjuntó a las cuentas un conector bifuncional removible protegido ortogonalmente. Se utilizó un extremo del conector para unir los bloques de construcción no naturales de la biblioteca, mientras que al otro extremo se unieron los tripletes de aminoácidos codificantes. Los componentes básicos eran aminoácidos no naturales y la serie de sus tripletes de aminoácidos codificantes podía determinarse mediante la degradación de Edman. El aspecto importante de este tipo de codificación era la posibilidad de escindir de las cuentas los miembros de la biblioteca junto con sus etiquetas de codificación adjuntas, formando una biblioteca soluble. El mismo enfoque fue utilizado por Nikolajev et al. para codificar con péptidos. [41] En 1992, Brenner y Lerner introdujeron secuencias de ADN para codificar las perlas del soporte sólido que resultó ser el método de codificación más exitoso. [42] Nielsen, Brenner y Janda también utilizaron el enfoque de Kerr para implementar la codificación del ADN. [43] En el último período hubo importantes avances en la secuenciación del ADN. Las técnicas de próxima generación permiten secuenciar una gran cantidad de muestras en paralelo, lo que es muy importante en la detección de bibliotecas codificadas por ADN. Hubo otra innovación que contribuyó al éxito de la codificación del ADN. En 2000, Halpin y Harbury omitieron el soporte sólido en la síntesis de mezcla dividida de las bibliotecas combinatorias codificadas por ADN y lo reemplazaron por los oligómeros de ADN codificantes. En la síntesis agrupada y dividida en fase sólida, el número de componentes de las bibliotecas no puede exceder el número de perlas del soporte. Gracias al novedoso enfoque de los autores, se eliminó esta restricción y se hizo posible preparar nuevos compuestos en un número prácticamente ilimitado. [44] ¡ La empresa danesa Nuevolution, por ejemplo, sintetizó una biblioteca codificada de ADN que contiene 40 billones! componentes [45]Las bibliotecas codificadas por ADN son solubles, lo que hace posible aplicar la unión por afinidad eficaz en el cribado. Algunos autores aplican DEL para acromim de bibliotecas combinatorias codificadas por ADN, otros utilizan DECL. Esto último parece mejor ya que en este nombre se expresa claramente la naturaleza combinatoria de estas bibliotecas. En la primera década del presente milenio se habían introducido y descrito varios tipos de bibliotecas combinatorias codificadas por ADN. Estas bibliotecas se aplican con mucho éxito en la investigación de fármacos.
Los detalles sobre su síntesis y aplicación se encuentran en la página Biblioteca química codificada por ADN . Las bibliotecas combinatorias solubles codificadas por ADN también tienen desventajas. En primer lugar, se pierde por completo la ventaja derivada del uso de un soporte sólido. Además, el carácter poliiónico de las cadenas que codifican el ADN limita la utilidad de disolventes no acuosos en la síntesis. Por este motivo, muchos laboratorios optan por desarrollar reacciones compatibles con el ADN para su uso en la síntesis de DECL. Ya se han descrito bastantes de los disponibles [51] [52] [53]
La ciencia de los materiales ha aplicado las técnicas de la química combinatoria al descubrimiento de nuevos materiales. Este trabajo fue iniciado por PG Schultz et al. a mediados de los años noventa [54] en el contexto de materiales luminiscentes obtenidos por codeposición de elementos sobre un sustrato de silicio. Su trabajo fue precedido por JJ Hanak en 1970 [55] pero las herramientas informáticas y robóticas no estaban disponibles para que el método se extendiera en ese momento. Varios grupos académicos [56] [57] [58] [59] , así como empresas con grandes programas de investigación y desarrollo ( Symyx Technologies , GE , Dow Chemical , etc.), han continuado el trabajo . La técnica se ha utilizado ampliamente para catálisis, [60] recubrimientos, [61] electrónica, [62] y muchos otros campos. [63] La aplicación de herramientas informáticas apropiadas es fundamental para manejar, administrar y almacenar los grandes volúmenes de datos producidos. [64] También se han desarrollado nuevos tipos de métodos de diseño de experimentos para abordar de manera eficiente los grandes espacios experimentales que pueden abordarse utilizando métodos combinatorios. [sesenta y cinco]
Aunque la química combinatoria ha sido una parte esencial del descubrimiento temprano de fármacos durante más de dos décadas, hasta ahora la FDA solo ha aprobado para uso clínico una sustancia química sintetizada mediante química combinatoria de novo ( sorafenib , un inhibidor multiquinasa indicado para el cáncer renal avanzado). . [66] Se ha sugerido que el análisis de la escasa tasa de éxito del enfoque se conecta con el espacio químico bastante limitado cubierto por los productos de la química combinatoria. [67] Al comparar las propiedades de los compuestos en bibliotecas de química combinatoria con las de medicamentos y productos naturales aprobados, Feher y Schmidt [67] observaron que las bibliotecas de química combinatoria sufren particularmente de falta de quiralidad , así como de rigidez estructural, las cuales son ampliamente considerados como propiedades similares a las de las drogas. Aunque el descubrimiento de fármacos de productos naturales probablemente no haya sido la tendencia más de moda en la industria farmacéutica en los últimos tiempos, [ cita necesaria ] una gran proporción de nuevas entidades químicas todavía son compuestos derivados de la naturaleza, [68] [69] [70] [ 71] [72] [73] y, por lo tanto, se ha sugerido que la eficacia de la química combinatoria podría mejorarse mejorando la diversidad química de las bibliotecas de detección. [74] Como la quiralidad y la rigidez son las dos características más importantes que distinguen los medicamentos aprobados y los productos naturales de los compuestos en las bibliotecas de química combinatoria, estas son las dos cuestiones que se enfatizan en las llamadas bibliotecas orientadas a la diversidad, es decir, colecciones de compuestos que tienen como objetivo cubrir la sustancia química. espacio, en lugar de simplemente grandes cantidades de compuestos. [75] [76] [77] [78] [79] [80]
En la octava edición de la Clasificación Internacional de Patentes (IPC), que entró en vigor el 1 de enero de 2006, se creó una subclase especial para solicitudes de patentes y patentes relacionadas con invenciones en el ámbito de la química combinatoria: "C40B".