
Una mutación de cambio de marco (también llamada error de encuadre o cambio de marco de lectura ) es una mutación genética causada por indeles ( inserciones o eliminaciones ) de una cantidad de nucleótidos en una secuencia de ADN que no es divisible por tres. Debido a la naturaleza triplete de la expresión génica mediante codones , la inserción o eliminación puede cambiar el marco de lectura (la agrupación de los codones), dando como resultado una traducción completamente diferente a la original. Cuanto antes se produzca la eliminación o inserción en la secuencia, más alterada estará la proteína. [1] Una mutación por desplazamiento del marco de lectura no es lo mismo que un polimorfismo de un solo nucleótido en el que se reemplaza un nucleótido, en lugar de insertarlo o eliminarlo. En general, una mutación por cambio de marco provocará que la lectura de los codones después de la mutación codifique diferentes aminoácidos. La mutación de cambio de marco también alterará el primer codón de parada ("UAA", "UGA" o "UAG") encontrado en la secuencia. El polipéptido que se está creando podría ser anormalmente corto o anormalmente largo y lo más probable es que no sea funcional. [2]
Las mutaciones de cambio de marco son evidentes en enfermedades genéticas graves como la enfermedad de Tay-Sachs ; aumentan la susceptibilidad a ciertos cánceres y clases de hipercolesterolemia familiar ; en 1997, [3] una mutación en el marco de lectura se relacionó con la resistencia a la infección por el retrovirus VIH. Las mutaciones por desplazamiento de marco se han propuesto como fuente de novedad biológica, al igual que con la supuesta creación de la nailonasa , sin embargo, esta interpretación es controvertida. Un estudio realizado por Negoro et al (2006) [4] encontró que era poco probable que la causa fuera una mutación por desplazamiento del marco de lectura y que más bien una sustitución de dos aminoácidos en el sitio activo de una esterasa ancestral dio como resultado la nailonasa.
La información contenida en el ADN determina la función de las proteínas en las células de todos los organismos. La transcripción y la traducción permiten que esta información se comunique para producir proteínas. Sin embargo, un error al leer esta comunicación puede causar que la función de las proteínas sea incorrecta y eventualmente causar enfermedades incluso cuando la célula incorpora una variedad de medidas correctivas. La información genética es transmitida por el ADN para la síntesis de proteínas dentro de las células. Una mala interpretación puede provocar una función defectuosa y enfermedades, a pesar de los mecanismos de corrección celular.
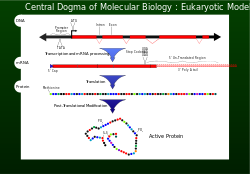
En 1956 , Francis Crick describió como dogma central el flujo de información genética desde el ADN hasta una disposición de aminoácidos específica para producir una proteína . [1] Para que una célula funcione correctamente, es necesario que las proteínas se produzcan con precisión para sus actividades estructurales y catalíticas . Una proteína producida incorrectamente puede tener efectos perjudiciales sobre la viabilidad celular y, en la mayoría de los casos, provocar que el organismo superior se vuelva insalubre debido a funciones celulares anormales. Para garantizar que el genoma transmita la información con éxito, en la replicación del ADN se incorporan mecanismos de corrección , como exonucleasas y sistemas de reparación de errores de coincidencia . [1]

Después de la replicación del ADN, la lectura de una sección seleccionada de información genética se logra mediante transcripción . [1] Los nucleótidos que contienen la información genética ahora se encuentran en una plantilla mensajera de una sola hebra llamada ARNm . El ARNm se incorpora a una subunidad del ribosoma e interactúa con un ARNr . La información genética contenida en los codones del ARNm ahora es leída (decodificada) por los anticodones del ARNt. A medida que se lee cada codón (triplete), los aminoácidos se van uniendo hasta alcanzar un codón de parada (UAG, UGA o UAA). En este punto el polipéptido (proteína) ha sido sintetizado y liberado. [1] Por cada 1000 aminoácidos incorporados a la proteína, no más de uno es incorrecto. Esta fidelidad del reconocimiento de codones, manteniendo la importancia del marco de lectura adecuado, se logra mediante el emparejamiento de bases adecuado en el sitio A del ribosoma, la actividad de hidrólisis de GTP de EF-Tu , una forma de estabilidad cinética, y un mecanismo de corrección a medida que se libera EF-Tu. . [1]
El cambio de marco también puede ocurrir durante la traducción en profase , produciendo diferentes proteínas a partir de marcos de lectura abiertos superpuestos, como las proteínas retrovirales gag-pol-env . Esto es bastante común en virus y también ocurre en bacterias y levaduras (Farabaugh, 1996). Se cree que la transcriptasa inversa , a diferencia de la ARN polimerasa II , es una causa más importante de la aparición de mutaciones de cambio de marco. En los experimentos, sólo entre el 3% y el 13% de todas las mutaciones de cambio de marco se produjeron debido a la ARN polimerasa II. En los procariotas, la tasa de error que induce mutaciones por cambio de marco está sólo en el rango de 0,0001 y 0,00001. [5]
Existen varios procesos biológicos que ayudan a prevenir las mutaciones por cambio de marco. Se producen mutaciones inversas que cambian la secuencia mutada a la secuencia original de tipo salvaje . Otra posibilidad para la corrección de mutaciones es el uso de una mutación supresora . Esto compensa el efecto de la mutación original al crear una mutación secundaria, cambiando la secuencia para permitir que se lean los aminoácidos correctos. El ARN guía también se puede utilizar para insertar o eliminar uridina en el ARNm después de la transcripción, lo que permite el marco de lectura correcto. [1]

Un codón es un conjunto de tres nucleótidos , un triplete que codifica un determinado aminoácido . El primer codón establece el marco de lectura, mediante el cual comienza un nuevo codón. La secuencia principal de aminoácidos de una proteína está definida por tripletes contiguos. [6] Los codones son clave para la traducción de información genética para la síntesis de proteínas. El marco de lectura se establece cuando comienza la traducción del ARNm y se mantiene mientras lee un triplete al siguiente. La lectura del código genético está sujeta a tres reglas que controlan los codones en el ARNm. Primero, los codones se leen en dirección 5' a 3'. En segundo lugar, los codones no se superponen y el mensaje no tiene espacios. La última regla, como se indicó anteriormente, es que el mensaje se traduzca en un marco de lectura fijo. [1]

Las mutaciones por desplazamiento de marco pueden ocurrir al azar o ser causadas por un estímulo externo. La detección de mutaciones de cambio de marco puede ocurrir mediante varios métodos diferentes. Los cambios de marco son solo un tipo de mutación que puede conducir a proteínas incompletas o incorrectas, pero representan un porcentaje significativo de errores en el ADN. En un gen inalterado, los codones (tripletes de nucleótidos) se interpretan secuencialmente, y cada codón codifica un amino específico. ácido. Esto se conoce como marco de lectura estándar. Sin embargo, en los casos de mutaciones por cambio de marco, se inserta un nucleótido adicional (o más) en la secuencia de ADN, lo que altera el marco de lectura típico y provoca un cambio en la secuencia.
Esta inserción provoca un cambio en el marco de lectura debido a la naturaleza triple del código genético. Por ejemplo, la adición de una "A" adicional conduce a un cambio de secuencia, lo que desencadena la lectura de un conjunto de codones completamente diferente. Esta desviación en la información genética hace que el ribosoma, que lee el ARNm para la síntesis de proteínas, malinterprete los datos genéticos. En consecuencia, se genera una serie de aminoácidos completamente diferente, lo que da como resultado la generación de una secuencia de proteínas alterada. En la mayoría de los casos, el nuevo marco de lectura da como resultado un encuentro temprano con un codón de parada, lo que lleva a la formación de una proteína acortada y generalmente inactiva. Esta forma de mutación se denomina codón de parada temprana o mutación sin sentido.
Se trata de una mutación genética a nivel de bases de nucleótidos. Continuamente se investiga por qué y cómo se producen las mutaciones por desplazamiento del marco de lectura. Se realizó un estudio ambiental, específicamente la producción de mutaciones de cambio de marco inducidas por UV por ADN polimerasas deficientes en la actividad exonucleasa 3′ → 5′. La secuencia normal 5 'GTC GTT TTA CAA 3' se cambió a GTC GTT T TTA CAA (MIDT) de GTC GTT C TTA CAA (MIDC) para estudiar los cambios de marco. Las enzimas mutantes E. coli pol I Kf y T7 ADN polimerasa desprovistas de actividad exonucleasa 3' → 5' producen reversiones inducidas por UV con mayor frecuencia que sus contrapartes competentes en exonucleasa . Los datos indican que la pérdida de actividad de corrección aumenta la frecuencia de los cambios de fotograma inducidos por los rayos UV. [7]
Los efectos de las bases vecinas y la estructura secundaria para detectar la frecuencia de mutaciones por desplazamiento de marco se han investigado en profundidad mediante fluorescencia . El ADN marcado con fluorescencia, mediante análogos de bases, permite estudiar los cambios locales de una secuencia de ADN. [8] Los estudios sobre los efectos de la longitud de la cadena del cebador revelan que se observó una mezcla en equilibrio de cuatro conformaciones de hibridación cuando las bases plantilla formaban un bulto, es decir, una estructura flanqueada en ambos lados por ADN dúplex. Por el contrario, se observó una estructura de doble bucle con una conformación inusual de ADN no apilado en su borde aguas abajo cuando las bases extruidas se colocaron en la unión cebador-plantilla, lo que demuestra que las desalineaciones pueden ser modificadas por la estructura secundaria de ADN vecina. [9]
.jpg/440px-Frameshift_deletion_(13062713935).jpg)
La secuenciación de Sanger y la pirosecuenciación son dos métodos que se han utilizado para detectar mutaciones de cambio de marco; sin embargo, es probable que los datos generados no sean de la más alta calidad. Aún así, se han identificado 1,96 millones de indeles mediante la secuenciación de Sanger que no se superponen con otras bases de datos. Cuando se observa una mutación de cambio de marco, se compara con la Base de datos de mutaciones del genoma humano (HGMD) para determinar si la mutación tiene un efecto dañino. Esto se hace observando cuatro características. Primero, la proporción entre el ADN afectado y conservado, segundo la ubicación de la mutación en relación con la transcripción, tercero la proporción de aminoácidos conservados y afectados y finalmente la distancia del indel al final del exón . [10]
La secuenciación masiva paralela es un método más nuevo que se puede utilizar para detectar mutaciones. Con este método, se pueden secuenciar hasta 17 gigabases a la vez, a diferencia de los rangos limitados para la secuenciación de Sanger de solo aproximadamente 1 kilobase. Hay varias tecnologías disponibles para realizar esta prueba y se está estudiando su uso en aplicaciones clínicas. [11] Cuando se realizan pruebas para detectar diferentes carcinomas, los métodos actuales solo permiten observar un gen a la vez. La secuenciación masiva paralela puede detectar una variedad de mutaciones que causan cáncer a la vez, en lugar de varias pruebas específicas. [12] Un experimento para determinar la precisión de este método de secuenciación más nuevo probó 21 genes y no tuvo llamadas falsas positivas para mutaciones de cambio de marco. [13]
Una patente estadounidense (5.958.684) de 1999 de Leeuwen detalla los métodos y reactivos para el diagnóstico de enfermedades causadas por o asociadas con un gen que tiene una mutación somática que da lugar a una mutación por cambio de marco. Los métodos incluyen proporcionar una muestra de tejido o líquido y realizar un análisis genético para detectar una mutación por cambio de marco o una proteína de este tipo de mutación. La secuencia de nucleótidos del gen sospechoso se obtiene a partir de secuencias genéticas publicadas o de la clonación y secuenciación del gen sospechoso. Luego se predice la secuencia de aminoácidos codificada por el gen. [14] Secuenciación NA: Se puede utilizar la secuenciación de Sanger o la secuenciación de próxima generación (NGS) para secuenciar directamente el ADN e identificar inserciones o eliminaciones. Reacción en cadena de la polimerasa (PCR): se puede utilizar la PCR para amplificar la región específica que contiene la mutación para análisis posterior.Amplificación de sonda dependiente de ligadura múltiple (MLPA): MLPA es una técnica utilizada para detectar variaciones en el número de copias y pequeñas inserciones o eliminaciones.Hibridación genómica comparativa (CGH): la CGH se utiliza para detectar desequilibrios cromosómicos, que pueden incluir grandes inserciones o eliminaciones.
A pesar de las reglas que rigen el código genético y los diversos mecanismos presentes en una célula para asegurar la correcta transferencia de información genética tanto durante el proceso de replicación del ADN como durante la traducción, las mutaciones ocurren; La mutación por cambio de marco no es el único tipo. Existen al menos otros dos tipos de mutaciones puntuales reconocidas, específicamente la mutación sin sentido y la mutación sin sentido . [1] Una mutación por desplazamiento de marco puede cambiar drásticamente la capacidad de codificación (información genética) del mensaje. [1] Las pequeñas inserciones o eliminaciones (aquellas de menos de 20 pares de bases) constituyen el 24% de las mutaciones que se manifiestan en enfermedades genéticas actualmente reconocidas. [10]
Se ha descubierto que las mutaciones por desplazamiento de marco son más comunes en regiones repetidas del ADN. Una razón de esto es el deslizamiento de la enzima polimerasa en regiones repetidas, lo que permite que las mutaciones entren en la secuencia . [15] Se pueden realizar experimentos para determinar la frecuencia de la mutación de cambio de marco agregando o eliminando un número preestablecido de nucleótidos. Se realizaron experimentos agregando cuatro pares de bases, llamados experimentos +4, pero un equipo de la Universidad de Emory observó la diferencia en la frecuencia de la mutación agregando y eliminando un par de bases. Se demostró que no había diferencia en la frecuencia entre la adición y eliminación de un par de bases. Sin embargo, existe una diferencia en el resultado final de la proteína. [15]
La enfermedad de Huntington es uno de los nueve trastornos de reiteración de codones causados por mutaciones de expansión de poliglutamina que incluyen ataxia espinocerebelosa (SCA) 1, 2, 6, 7 y 3, atrofia muscular espinobulbar y palidoluysianatrofia dentatorubal. Puede haber un vínculo entre las enfermedades causadas por mutaciones de expansión de poliglutamina y polialanina, como el cambio de marco del producto del gen SCA3 original que codifica CAG/poliglutaminas a GCA/polialaninas. Se ha propuesto el deslizamiento ribosomal durante la traducción de la proteína SCA3 como el mecanismo que resulta en el cambio del marco codificante de poliglutamina al marco codificante de polialanina. Una eliminación de dinucleótido o inserción de un solo nucleótido dentro del tracto de poliglutamina del exón 1 de la Huntingtina cambiaría el marco codificante de poliglutamina, CAG, en +1 (desplazamiento de marco +1) al marco codificante de polialanina, GCA, e introduciría un nuevo epítopo en el extremo C terminal de Htt exón 1 (APAAAPAATRPGCG). [dieciséis]
Varias enfermedades tienen mutaciones de cambio de marco como al menos parte de la causa. Conocer las mutaciones prevalentes también puede ayudar en el diagnóstico de la enfermedad. Actualmente hay intentos de utilizar mutaciones de cambio de marco de forma beneficiosa en el tratamiento de enfermedades, cambiando el marco de lectura de los aminoácidos.


Se sabe que las mutaciones de cambio de marco son un factor en el cáncer colorrectal , así como en otros cánceres con inestabilidad de microsatélites . Como se indicó anteriormente, es más probable que se produzcan mutaciones por cambio de marco en una región de secuencia repetida. Cuando la reparación de errores de coincidencia del ADN no soluciona la adición o eliminación de bases, es más probable que estas mutaciones sean patógenas. Esto puede deberse en parte a que no se le dice al tumor que deje de crecer. Los experimentos con levaduras y bacterias ayudan a mostrar características de los microsatélites que pueden contribuir a la reparación defectuosa de las discordancias del ADN. Estos incluyen la longitud del microsatélite , la composición del material genético y la pureza de las repeticiones. Según resultados experimentales, los microsatélites más largos tienen una mayor tasa de mutaciones por desplazamiento de marco. El ADN flanqueante también puede contribuir a mutaciones de cambio de marco. [17] En el cáncer de próstata, una mutación en el marco de lectura cambia el marco de lectura abierto (ORF) y evita que se produzca la apoptosis . Esto conduce a un crecimiento desregulado del tumor . Si bien existen factores ambientales que contribuyen a la progresión del cáncer de próstata , también existe un componente genético. Durante las pruebas de regiones codificantes para identificar mutaciones, se descubrieron 116 variantes genéticas, incluidas 61 mutaciones de cambio de marco. [18] Hay más de 500 mutaciones en el cromosoma 17 que parecen desempeñar un papel en el desarrollo del cáncer de mama y de ovario en el gen BRCA1, muchas de las cuales cambian el marco de lectura. [19]
La enfermedad de Crohn tiene una asociación con el gen NOD2. La mutación es una inserción de una citosina en la posición 3020. Esto conduce a un codón de parada prematuro, acortando la proteína que se supone debe transcribirse. Cuando la proteína puede formarse normalmente, responde a los liposacáridos bacterianos, donde la mutación 3020insC impide que la proteína responda. [20]
La fibrosis quística (FQ) es una enfermedad basada en mutaciones en el gen regulador de la conductancia transmembrana de la FQ (CFTR). Se han identificado más de 1.500 mutaciones, pero no todas causan la enfermedad. [21] La mayoría de los casos de fibrosis quística son el resultado de la mutación ∆F508, que elimina todo el aminoácido. Dos mutaciones de cambio de marco son de interés en el diagnóstico de la FQ, CF1213delT y CF1154-insTC. Ambas mutaciones suelen ocurrir junto con al menos otra mutación. Ambos conducen a una pequeña disminución en la función de los pulmones y ocurren en aproximadamente el 1% de los pacientes evaluados. Estas mutaciones se identificaron mediante secuenciación de Sanger. [22]
CCR5 es uno de los cofactores de entrada celular asociados con el VIH, más frecuentemente involucrado con cepas no inductoras de sincitio, y es más evidente en pacientes con VIH que en pacientes con SIDA. Se ha identificado una deleción de 32 pares de bases en CCR5 como una mutación que niega la probabilidad de una infección por VIH. Esta región en el ORF del marco de lectura abierto contiene una mutación de desplazamiento del marco de lectura que conduce a un codón de parada prematuro. Esto conduce a la pérdida de la función del correceptor del VIH in vitro. CCR5-1 se considera el tipo salvaje y CCR5-2 se considera el alelo mutante. Aquellos con una mutación heterocigota para el CCR5 eran menos susceptibles al desarrollo del VIH. En un estudio, a pesar de la alta exposición al virus del VIH, no hubo ningún homocigoto para la mutación CCR5 que dio positivo al VIH. [3]
La enfermedad de Tay-Sachs es una enfermedad mortal que afecta al sistema nervioso central. Se encuentra con mayor frecuencia en bebés y niños pequeños. La progresión de la enfermedad comienza en el útero , pero los síntomas no aparecen hasta aproximadamente los 6 meses de edad. No existe cura para la enfermedad. [23] Se sabe que las mutaciones en el gen de la β-hexosaminidasa A (Hex A) afectan la aparición de Tay-Sachs; se describen 78 mutaciones de diferentes tipos, 67 de las cuales se sabe que causan la enfermedad. La mayoría de las mutaciones observadas (65/78) son sustituciones de una sola base o SNP, 11 deleciones, 1 grande y 10 pequeñas, y 2 inserciones. 8 de las mutaciones observadas son cambios de marco, 6 deleciones y 2 inserciones. Se observa una inserción de 4 pares de bases en el exón 11 en el 80% de los casos de enfermedad de Tay-Sachs en la población judía asquenazí . Las mutaciones de cambio de marco conducen a un codón de parada temprano que se sabe que desempeña un papel en la enfermedad en los bebés. La enfermedad de aparición tardía parece ser causada por 4 mutaciones diferentes, una de las cuales es una deleción de 3 pares de bases. [24]
El síndrome de Smith-Magenis (SMS) es un síndrome complejo que involucra discapacidad intelectual, alteraciones del sueño, problemas de conducta y una variedad de anomalías craneofaciales, esqueléticas y viscerales. La mayoría de los casos de SMS albergan una deleción común de ~3,5 Mb que abarca el gen 1 inducido por ácido retinoico ( RAI1 ). Otros casos ilustran una variabilidad en el fenotipo SMS que no se había mostrado previamente para la mutación RAI1, incluida la pérdida de audición, la ausencia de conductas autoabusivas y retrasos globales leves. La secuenciación de RAI1 reveló una mutación de un tracto C heptamérico (CCCCCCC) en el exón 3, lo que resulta en mutaciones de cambio de marco. De las siete mutaciones de cambio de marco reportadas que ocurren en los tractos poli C en RAI1, cuatro casos (~57%) ocurren en este tracto C heptamérico. Los resultados indican que este tracto C heptamérico es un punto de acceso preferencial de inserción/eliminación de recombinación (SNindels) y, por lo tanto, un objetivo principal para el análisis en pacientes con sospecha de mutaciones en RAI1. [25]
La miocardiopatía hipertrófica es la causa más común de muerte súbita en los jóvenes, incluidos los atletas entrenados, y está causada por mutaciones en genes que codifican proteínas del sarcómero cardíaco. Las mutaciones en el gen de la troponina C ( TNNC1 ) son una causa genética poco común de miocardiopatía hipertrófica. Un estudio reciente ha indicado que una mutación del marco de lectura (c.363dupG o p.Gln122AlafsX30) en la troponina C fue la causa de la miocardiopatía hipertrófica (y muerte súbita cardíaca) en un hombre de 19 años. [26]
Es raro encontrar una cura para las enfermedades causadas por mutaciones de cambio de marco. La investigación sobre esto está en curso. Un ejemplo es la inmunodeficiencia primaria (PID), una condición hereditaria que puede provocar un aumento de las infecciones. Hay 120 genes y 150 mutaciones que desempeñan un papel en las inmunodeficiencias primarias. El tratamiento estándar actualmente es la terapia génica , pero se trata de un tratamiento de gran riesgo y que muchas veces puede derivar en otras enfermedades, como la leucemia. Los procedimientos de terapia génica incluyen la modificación de la proteína de fusión de la nucleasa con franja de zinc, escindiendo ambos extremos de la mutación, lo que a su vez la elimina de la secuencia. La omisión de exones mediada por oligonucleótidos antisentido es otra posibilidad de distrofia muscular de Duchenne . Este proceso permite pasar por alto la mutación para que el resto de la secuencia permanezca en marco y la función de la proteína permanezca intacta. Sin embargo, esto no cura la enfermedad, sólo trata los síntomas y sólo es práctico en proteínas estructurales u otros genes repetitivos. Una tercera forma de reparación es el mosaicismo revertido , que ocurre naturalmente al crear una mutación inversa o una mutación en un segundo sitio que corrige el marco de lectura. Esta reversión puede ocurrir por recombinación intragénica , conversión de genes mitóticos , deslizamiento del ADN en el segundo sitio o reversión específica del sitio. Esto es posible en varias enfermedades, como la inmunodeficiencia combinada grave ligada al cromosoma X (SCID), el síndrome de Wiskott-Aldrich y el síndrome de Bloom . No existen medicamentos ni otros métodos farmacogenómicos que ayuden con las EIP. [27]
Una patente europea (EP1369126A1) de 2003 de Bork registra un método utilizado para la prevención de cánceres y para el tratamiento curativo de cánceres y precánceres, como los tumores esporádicos con deficiencia en la reparación de desajustes del ADN (MMR) y los tumores asociados con HNPCC. La idea es utilizar inmunoterapia con mezclas combinatorias de péptidos derivados de mutaciones de cambio de marco específicos de tumores para provocar una respuesta de células T citotóxicas dirigida específicamente contra las células tumorales. [28]