Un par de cromosomas homólogos , u homólogos , es un conjunto de un cromosoma materno y uno paterno que se aparean entre sí dentro de una célula durante la fertilización . Los homólogos tienen los mismos genes en los mismos loci , donde proporcionan puntos a lo largo de cada cromosoma que permiten que un par de cromosomas se alineen correctamente entre sí antes de separarse durante la meiosis . [1] Esta es la base de la herencia mendeliana , que caracteriza los patrones de herencia del material genético de un organismo a su célula de desarrollo parental descendiente en el momento y área determinados. [2]
Los cromosomas son disposiciones lineales de ácido desoxirribonucleico (ADN) condensado y proteínas histonas , que forman un complejo llamado cromatina . [2] Los cromosomas homólogos están formados por pares de cromosomas de aproximadamente la misma longitud, posición del centrómero y patrón de tinción, para genes con los mismos loci correspondientes . Un cromosoma homólogo se hereda de la madre del organismo; el otro se hereda del padre del organismo. Después de que ocurre la mitosis dentro de las células hijas, tienen la cantidad correcta de genes que son una mezcla de los genes de los dos padres. En los organismos diploides (2n), el genoma está compuesto por un conjunto de cada par de cromosomas homólogos, en comparación con los organismos tetraploides que pueden tener dos conjuntos de cada par de cromosomas homólogos. Los alelos en los cromosomas homólogos pueden ser diferentes, lo que resulta en diferentes fenotipos de los mismos genes. Esta mezcla de rasgos maternos y paternos se ve potenciada por el entrecruzamiento durante la meiosis , en la que las longitudes de los brazos cromosómicos y el ADN que contienen dentro de un par de cromosomas homólogos se intercambian entre sí. [3]
A principios de la década de 1900, William Bateson y Reginald Punnett estudiaban la herencia genética y observaron que algunas combinaciones de alelos aparecían con mayor frecuencia que otras. Thomas Morgan exploró más a fondo esos datos e información . Mediante experimentos de cruce de prueba , reveló que, en el caso de un solo progenitor, los alelos de los genes cercanos entre sí a lo largo del cromosoma se mueven juntos. Utilizando esta lógica, concluyó que los dos genes que estaba estudiando se encontraban en cromosomas homólogos. Más tarde, durante la década de 1930, Harriet Creighton y Barbara McClintock estudiaban la meiosis en células de maíz y examinaban los loci genéticos en los cromosomas del maíz. [2] Creighton y McClintock descubrieron que las nuevas combinaciones de alelos presentes en la descendencia y el evento de cruzamiento estaban directamente relacionados. [2] Esto demostró la recombinación genética intercromosómica. [2]
Los cromosomas homólogos son pares de cromosomas en un organismo diploide que tienen genes similares, aunque no necesariamente idénticos. [4] Hay dos propiedades principales de los cromosomas homólogos: 1) la longitud de los brazos cromosómicos y 2) la ubicación del centrómero. [5]
La longitud real del brazo, de acuerdo con las ubicaciones de los genes, es de importancia crítica para una alineación adecuada. La ubicación del centrómero en el cromosoma se puede caracterizar por cuatro disposiciones principales, ya sea metacéntrica , submetacéntrica , acrocéntrica o telocéntrica . Ambas propiedades (es decir, la longitud de los brazos cromosómicos y la ubicación del centrómero cromosómico) son los factores principales para crear homología estructural entre cromosomas. Por lo tanto, cuando existen dos cromosomas que contienen la misma estructura relativamente (por ejemplo, el cromosoma 15 materno y el cromosoma 15 paterno), pueden aparearse entre sí a través del proceso de sinapsis para formar cromosomas homólogos. [6]
Dado que los cromosomas homólogos no son idénticos ni se originan en el mismo organismo, son diferentes de las cromátidas hermanas . Las cromátidas hermanas surgen después de que se ha producido la replicación del ADN y, por lo tanto, son idénticas, duplicadas una al lado de la otra. [7]
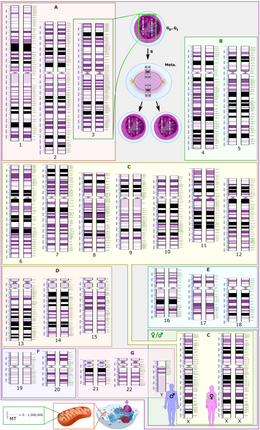
Los seres humanos tienen un total de 46 cromosomas, pero sólo hay 22 pares de cromosomas autosómicos homólogos . El par 23 adicional son los cromosomas sexuales, X e Y.
Tenga en cuenta que el par de cromosomas sexuales puede ser homólogo o no, dependiendo del sexo del individuo. Por ejemplo, las mujeres contienen XX, por lo que tienen un par homólogo de cromosomas sexuales. Esto significa que las mujeres tienen 23 pares de cromosomas homólogos en total (es decir, 22 pares de cromosomas no sexuales (autosomas), 1 par de cromosomas sexuales). Por el contrario, los hombres contienen XY, lo que significa que tienen un par no homólogo de cromosomas sexuales como su par de cromosomas número 23.
En los seres humanos, los 22 pares de cromosomas autosómicos homólogos contienen los mismos genes pero codifican diferentes rasgos en sus formas alélicas, ya que uno se hereda de la madre y el otro del padre. [8]
Por lo tanto, los humanos tienen dos conjuntos de 23 cromosomas en cada célula que contiene un núcleo. Un conjunto de 23 cromosomas (n) proviene de la madre (22 autosomas, 1 cromosoma sexual (X solamente)) y un conjunto de 23 cromosomas (n) proviene del padre (22 autosomas, 1 cromosoma sexual (X o Y)). En definitiva, esto significa que los humanos son organismos diploides (2n). [2]
Los cromosomas homólogos son importantes en los procesos de meiosis y mitosis, ya que permiten la recombinación y la segregación aleatoria de material genético de la madre y el padre en nuevas células. [9]


La meiosis es una ronda de dos divisiones celulares que dan como resultado cuatro células hijas haploides que contienen cada una la mitad del número de cromosomas que la célula madre. [10] Reduce el número de cromosomas en una célula germinal a la mitad separando primero los cromosomas homólogos en la meiosis I y luego las cromátidas hermanas en la meiosis II . El proceso de la meiosis I es generalmente más largo que el de la meiosis II porque lleva más tiempo que la cromatina se replique y que los cromosomas homólogos se orienten y segreguen adecuadamente mediante los procesos de apareamiento y sinapsis en la meiosis I. [7] Durante la meiosis, la recombinación genética (por segregación aleatoria) y el entrecruzamiento producen células hijas que contienen cada una diferentes combinaciones de genes codificados materna y paternamente. [10] Esta recombinación de genes permite la introducción de nuevos emparejamientos de alelos y variación genética. [2] La variación genética entre organismos ayuda a que una población sea más estable al proporcionar una gama más amplia de rasgos genéticos sobre los que puede actuar la selección natural . [2]
En la profase I de la meiosis I, cada cromosoma está alineado con su pareja homóloga y se aparea completamente. En la profase I, el ADN ya ha experimentado la replicación, por lo que cada cromosoma consta de dos cromátidas idénticas conectadas por un centrómero común. [10] Durante la etapa de cigoteno de la profase I, los cromosomas homólogos se aparean entre sí. [10] Este apareamiento ocurre mediante un proceso de sinapsis donde el complejo sinaptonémico , un andamiaje de proteínas, se ensambla y une los cromosomas homólogos a lo largo de sus longitudes. [7] La reticulación de cohesina ocurre entre los cromosomas homólogos y los ayuda a resistir la separación hasta la anafase . [8] El entrecruzamiento genético , un tipo de recombinación, ocurre durante la etapa de paquiteno de la profase I. [10] Además, con frecuencia ocurre otro tipo de recombinación denominada recocido de cadena dependiente de síntesis (SDSA). La recombinación SDSA implica el intercambio de información entre cromátidas homólogas emparejadas , pero no el intercambio físico. La recombinación SDSA no causa entrecruzamiento.
En el proceso de entrecruzamiento, los genes se intercambian mediante la ruptura y unión de porciones homólogas de las longitudes de los cromosomas. [7] Las estructuras llamadas quiasmas son el sitio del intercambio. Los quiasmas unen físicamente los cromosomas homólogos una vez que se produce el entrecruzamiento y durante todo el proceso de segregación cromosómica durante la meiosis. [7] Tanto los tipos de recombinación sin entrecruzamiento como los de entrecruzamiento funcionan como procesos para reparar el daño del ADN , en particular las roturas de doble cadena. En la etapa de diploteno de la profase I, el complejo sinaptonémico se desmonta antes de que los cromosomas homólogos se separen, mientras que las cromátidas hermanas permanecen asociadas por sus centrómeros. [7]
En la metafase I de la meiosis I, los pares de cromosomas homólogos, también conocidos como bivalentes o tétradas , se alinean en un orden aleatorio a lo largo de la placa de metafase . [10] La orientación aleatoria es otra forma en que las células introducen variación genética. Los husos meióticos que emanan de polos opuestos del huso se unen a cada uno de los homólogos (cada par de cromátidas hermanas) en el cinetocoro . [8]
En la anafase I de la meiosis I, los cromosomas homólogos se separan entre sí. Los homólogos son escindidos por la enzima separasa para liberar la cohesina que mantenía unidos los brazos de los cromosomas homólogos. [8] Esto permite que los quiasmas se liberen y los homólogos se muevan a polos opuestos de la célula. [8] Los cromosomas homólogos ahora se segregan aleatoriamente en dos células hijas que experimentarán la meiosis II para producir cuatro células germinales hijas haploides . [2]
Después de que las tétradas de cromosomas homólogos se separan en la meiosis I, las cromátidas hermanas de cada par se separan. Las dos células hijas haploides (el número de cromosomas se ha reducido a la mitad: antes había dos juegos de cromosomas, pero ahora cada juego existe en dos células hijas diferentes que han surgido de la única célula madre diploide por meiosis I) resultantes de la meiosis I experimentan otra división celular en la meiosis II, pero sin otra ronda de replicación cromosómica. Las cromátidas hermanas en las dos células hijas se separan durante la anafase II por las fibras del huso nuclear, lo que da como resultado cuatro células hijas haploides. [2]
Los cromosomas homólogos no funcionan de la misma manera en la mitosis que en la meiosis. Antes de cada división mitótica que experimenta una célula, los cromosomas de la célula madre se replican a sí mismos. Los cromosomas homólogos dentro de la célula normalmente no se emparejan ni experimentan una recombinación genética entre sí. [10] En cambio, los replicantes, o cromátidas hermanas, se alinean a lo largo de la placa metafásica y luego se separan de la misma manera que en la meiosis II: separándose en sus centrómeros por los husos mitóticos nucleares. [11] Si se produce algún entrecruzamiento entre las cromátidas hermanas durante la mitosis, no produce ningún genotipo recombinante nuevo. [2]
En la mayoría de los contextos, el emparejamiento homólogo se refiere a las células de la línea germinal, pero también ocurre en las células somáticas. Por ejemplo, en los humanos, las células somáticas tienen un emparejamiento homólogo regulado de manera muy estricta (separadas en territorios cromosómicos y emparejamiento en loci específicos bajo el control de la señalización del desarrollo). Sin embargo, otras especies (en particular, Drosophila ) muestran un emparejamiento homólogo con mucha más frecuencia. En Drosophila, el emparejamiento homólogo respalda un fenómeno de regulación genética llamado transvección en el que un alelo en un cromosoma afecta la expresión del alelo homólogo en el cromosoma homólogo. [12] Una función notable de esto es la regulación sexualmente dimórfica de los genes ligados al cromosoma X. [13]

La segregación incorrecta de los cromosomas puede tener graves consecuencias, como problemas de fertilidad , muerte de embriones , defectos congénitos y cáncer . [14] Aunque los mecanismos de apareamiento y adhesión de cromosomas homólogos varían entre organismos, el funcionamiento adecuado de dichos mecanismos es fundamental para que el material genético final se clasifique correctamente. [14]
La separación adecuada de los cromosomas homólogos en la meiosis I es crucial para la separación de las cromátidas hermanas en la meiosis II. [14] La falta de separación adecuada se conoce como no disyunción. Existen dos tipos principales de no disyunción que ocurren: trisomía y monosomía . La trisomía es causada por la presencia de un cromosoma adicional en el cigoto en comparación con el número normal, y la monosomía se caracteriza por la presencia de un cromosoma menos en el cigoto en comparación con el número normal. Si esta división desigual ocurre en la meiosis I, entonces ninguna de las células hijas tendrá una distribución cromosómica adecuada y pueden producirse efectos atípicos, incluido el síndrome de Down. [15] La división desigual también puede ocurrir durante la segunda división meiótica. La no disyunción que ocurre en esta etapa puede dar como resultado células hijas normales y células deformadas. [5]
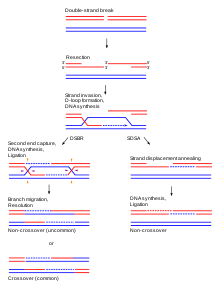
Si bien la función principal de los cromosomas homólogos es su uso en la división nuclear, también se utilizan para reparar roturas de doble cadena del ADN . [16] Estas roturas de doble cadena pueden ocurrir en el ADN replicante y, con mayor frecuencia, son el resultado de la interacción del ADN con moléculas dañinas naturales, como las especies reactivas de oxígeno . Los cromosomas homólogos pueden reparar este daño alineándose con cromosomas de la misma secuencia genética. [16] Una vez que los pares de bases se han emparejado y orientado correctamente entre las dos cadenas, los cromosomas homólogos realizan un proceso que es muy similar a la recombinación o entrecruzamiento como se ve en la meiosis. Parte de la secuencia de ADN intacta se superpone con la secuencia del cromosoma dañado . Luego, las proteínas y los complejos de replicación se reclutan en el sitio del daño, lo que permite que se produzca la reparación y la replicación adecuada. A través de este funcionamiento, las roturas de doble cadena se pueden reparar y el ADN puede funcionar normalmente. [16]
La investigación actual y futura sobre el tema de los cromosomas homólogos se centra en gran medida en las funciones de varias proteínas durante la recombinación o durante la reparación del ADN. En un artículo publicado recientemente por Pezza et al. [ which? ] la proteína conocida como HOP2 es responsable tanto de la sinapsis de los cromosomas homólogos como de la reparación de la rotura de doble cadena mediante la recombinación homóloga. La eliminación de HOP2 en ratones tiene grandes repercusiones en la meiosis. [17] Otros estudios actuales también se centran en proteínas específicas implicadas en la recombinación homóloga.
Actualmente se están realizando investigaciones sobre la capacidad de los cromosomas homólogos para reparar las roturas de la doble cadena de ADN. Los investigadores están estudiando la posibilidad de explotar esta capacidad para la medicina regenerativa. [18] Esta medicina podría ser muy común en relación con el cáncer, ya que se cree que el daño del ADN contribuye a la carcinogénesis. La manipulación de la función de reparación de los cromosomas homólogos podría permitir mejorar el sistema de respuesta al daño de una célula. Si bien las investigaciones aún no han confirmado la eficacia de este tratamiento, puede convertirse en una terapia útil para el cáncer. [19]