Los biomarcadores del envejecimiento son biomarcadores que podrían predecir la capacidad funcional a una edad posterior mejor que la edad cronológica. [1] Dicho de otra manera, los biomarcadores del envejecimiento darían la verdadera "edad biológica", que puede ser diferente de la edad cronológica.
Los biomarcadores de envejecimiento validados permitirían probar intervenciones para extender la esperanza de vida , porque los cambios en los biomarcadores serían observables a lo largo de la vida del organismo. [1] Aunque la esperanza de vida máxima sería un medio para validar los biomarcadores de envejecimiento, no sería un medio práctico para especies de larga vida como los humanos porque los estudios longitudinales tomarían demasiado tiempo. [2] Idealmente, los biomarcadores de envejecimiento deberían analizar el proceso biológico del envejecimiento y no una predisposición a la enfermedad, deberían causar una cantidad mínima de trauma para analizar en el organismo y deberían ser reproduciblemente medibles durante un intervalo corto en comparación con la esperanza de vida del organismo. [1] Un conjunto de datos de biomarcadores para un organismo podría denominarse su "ageotipo". [3]
Aunque el encanecimiento del cabello aumenta con la edad, [4] no se puede decir que sea un biomarcador del envejecimiento. De manera similar, las arrugas de la piel y otros cambios comunes que se observan con el envejecimiento no son mejores indicadores de la funcionalidad futura que la edad cronológica. Los biogerontólogos han continuado sus esfuerzos para encontrar y validar biomarcadores del envejecimiento, pero hasta ahora el éxito ha sido limitado. Los niveles de células T de memoria CD4 y CD8 y de células T ingenuas se han utilizado para proporcionar buenas predicciones de la esperanza de vida esperada de ratones de mediana edad. [5]
Los avances en el análisis de macrodatos permitieron el desarrollo de nuevos tipos de "relojes de envejecimiento". El reloj epigenético es un biomarcador prometedor del envejecimiento y puede predecir con precisión la edad cronológica humana. [6] La bioquímica sanguínea básica y los recuentos celulares también se pueden utilizar para predecir con precisión la edad cronológica. [7] Estudios posteriores del reloj hematológico en los grandes conjuntos de datos de poblaciones de Corea del Sur, Canadá y Europa del Este demostraron que los biomarcadores del envejecimiento pueden ser específicos de la población y predecir la mortalidad. [8] También es posible predecir la edad cronológica humana utilizando el reloj transcriptómico. [9]
Una nueva marca epigenética encontrada en estudios de células envejecidas es la pérdida de histonas . La mayoría de la evidencia muestra que la pérdida de histonas está relacionada con la división celular. En levaduras envejecidas y en división , la MNasa-seq (secuenciación de nucleasa microcócica) mostró una pérdida de nucleosomas de ~50%. La dosis adecuada de histonas es importante en la levadura, como lo demuestran las vidas prolongadas observadas en cepas que sobreexpresan histonas. [10] Una consecuencia de la pérdida de histonas en la levadura es la amplificación de la transcripción . En células más jóvenes, los genes que se inducen más con la edad tienen estructuras de cromatina específicas, como posicionamiento nuclear difuso, falta de una región agotada de nucleosomas (NDR) en el promotor , fase débil de la cromatina, una mayor frecuencia de elementos TATA y mayor ocupación de factores represivos de la cromatina. Sin embargo, en células más viejas, la pérdida de nucleosomas de los mismos genes en el promotor es más frecuente, lo que conduce a una mayor transcripción de estos genes. [10]
Este fenómeno no sólo se observa en levaduras, sino que también se ha observado en gusanos en proceso de envejecimiento, durante el envejecimiento de fibroblastos primarios diploides humanos y en células humanas senescentes . En fibroblastos primarios humanos, se observó que la síntesis reducida de nuevas histonas era una consecuencia de telómeros acortados que activan la respuesta al daño del ADN. La pérdida de histonas centrales puede ser una marca epigenética general del envejecimiento en muchos organismos. [11]
Además de las histonas centrales, H2A, H2B, H3 y H4, existen otras versiones de las proteínas histonas que pueden ser significativamente diferentes en su secuencia y son importantes para regular la dinámica de la cromatina. La histona H3.3 es una variante de la histona H3 que se incorpora al genoma independientemente de la replicación. Es la forma principal de la histona H3 que se observa en la cromatina de las células humanas senescentes, y parece que el exceso de H3.3 puede impulsar la senescencia . [11]
Existen múltiples variantes de la histona 2, la que está más implicada en el envejecimiento es la macroH2A. Se ha asumido generalmente que la función de la macroH2A es el silenciamiento transcripcional; más recientemente, se ha sugerido que la macroH2A es importante en la represión de la transcripción en los focos de heterocromatina asociados a la senescencia (SAHF). [11] La cromatina que contiene macroH2A es inmune a las proteínas de remodelación dependientes de ATP y a la unión de factores de transcripción . [12]
El aumento de la acetilación de las histonas contribuye a que la cromatina adopte un estado más eucromático a medida que el organismo envejece, similar al aumento de la transcripción observado debido a la pérdida de histonas. [13] También hay una reducción en los niveles de H3K56ac durante el envejecimiento y un aumento en los niveles de H4K16ac . [10] El aumento de H4K16ac en células de levadura viejas se asocia con la disminución de los niveles de HDAC Sir2, que puede aumentar la esperanza de vida cuando se sobreexpresa. [10]
La metilación de histonas se ha relacionado con la regulación de la esperanza de vida en muchos organismos, específicamente H3K4me3, una marca activadora, y H4K27me3, una marca represora. En C. elegans , la pérdida de cualquiera de las tres proteínas Trithorax que catalizan la trimetilación de H3K4, como WDR-5 y las metiltransferasas SET-2 y ASH-2, reduce los niveles de H3K4me3 y aumenta la esperanza de vida. La pérdida de la enzima que desmetila H3K4me3, RB-2, aumenta los niveles de H3K4me3 en C. elegans y disminuye su esperanza de vida. [13] En la corteza prefrontal del cerebro del macaco rhesus , H3K4me2 aumenta en promotores y potenciadores durante el desarrollo posnatal y el envejecimiento . [14] Estos aumentos reflejan estructuras de cromatina progresivamente más activas y transcripcionalmente accesibles (o abiertas) , que a menudo se asocian con respuestas al estrés, como la respuesta al daño del ADN . Estos cambios pueden formar una memoria epigenética de las tensiones y los daños que experimenta el organismo a medida que se desarrolla y envejece. [14]
UTX-1, una desmetilasa de H3K27me3 , desempeña un papel fundamental en el envejecimiento de C.elegans : el aumento de la expresión de utx-1 se correlaciona con una disminución de H3K27me3 y una disminución de la esperanza de vida. La eliminación de UTX-1 mostró un aumento de la esperanza de vida [13] . Los cambios en los niveles de H3K27me3 también tienen efectos sobre el envejecimiento de las células en Drosophila y humanos.
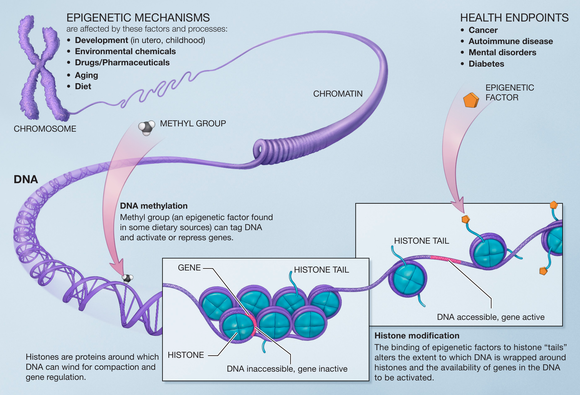
La metilación del ADN es una modificación común en las células de los mamíferos . La base de citosina se metila y se convierte en 5-metilcitosina , con mayor frecuencia cuando se encuentra en el contexto de CpG . La hipermetilación de las islas CpG se asocia con la represión transcripcional y la hipometilación de estos sitios se asocia con la activación transcripcional. Muchos estudios han demostrado que hay una pérdida de metilación del ADN durante el envejecimiento en muchas especies, como ratas, ratones, vacas, hámsteres y humanos. También se ha demostrado que DNMT1 y DNMT3a disminuyen con el envejecimiento y DNMT3b aumenta. [15]
La hipometilación del ADN puede reducir la estabilidad genómica, inducir la reactivación de elementos transponibles y causar la pérdida de la impronta , todo lo cual puede contribuir a la progresión y patogénesis del cáncer . [15]
Datos recientes sugieren que una mayor frecuencia de células T CD8+ senescentes en la sangre periférica está asociada con el desarrollo de hiperglucemia a partir de un estado prediabético, lo que sugiere que la senescencia desempeña un papel en el envejecimiento metabólico. Las células T CD8+ senescentes podrían utilizarse como un biomarcador para señalar la transición de la prediabetes a la hiperglucemia manifiesta. [16]
Recientemente, Hashimoto y colaboradores analizaron miles de células inmunes circulantes de supercentenarios con una resolución de célula única. Identificaron un aumento único de células T CD4 citotóxicas en estos supercentenarios. Generalmente, las células T CD4 tienen funciones auxiliares, pero no citotóxicas, en condiciones fisiológicas; sin embargo, estos supercentenarios, sometidos a un análisis de células individuales de sus receptores de células T, revelaron acumulaciones de células T CD4 citotóxicas a través de la expansión clonal. La conversión de células T CD4 auxiliares a una variedad citotóxica podría ser una adaptación a la etapa tardía del envejecimiento que ayuda a combatir las infecciones y potencialmente mejora la vigilancia tumoral. [17]
Los principales mecanismos identificados como posibles biomarcadores del envejecimiento son la metilación del ADN, la pérdida de histonas y la modificación de las histonas. Los usos de los biomarcadores del envejecimiento son ubicuos e identificar un parámetro físico del envejecimiento biológico permitiría a los humanos determinar nuestra edad real, mortalidad y morbilidad. [10] El cambio en el biomarcador físico debería ser proporcional al cambio en la edad de la especie. Por lo tanto, después de establecer un biomarcador del envejecimiento, los humanos podrían sumergirse en la investigación sobre la prolongación de la esperanza de vida y la búsqueda de cronogramas para la aparición de posibles enfermedades genéticas.
Una de las aplicaciones de este hallazgo permitiría identificar la edad biológica de una persona. La metilación del ADN utiliza la estructura del ADN en diferentes etapas de la vida para determinar una edad. La metilación del ADN es la metilación de la cisteína en la región CG o Cpg. La hipermetilación de esta región se asocia con una menor actividad transcripcional y lo contrario para la hipometilación. En otras palabras, cuanto más "firme" esté la región del ADN, más estable y "joven" será la especie. Al observar las propiedades de la metilación del ADN en los tejidos, se encontró que era casi cero para los tejidos embrionarios, se puede utilizar para determinar la aceleración de la edad y los resultados se pueden reproducir en el tejido del chimpancé. [18]
Más recientemente, los biomarcadores del envejecimiento se han utilizado en múltiples ensayos clínicos para medir la desaceleración o la reversión del deterioro relacionado con la edad o el envejecimiento biológico. [19] El Consorcio de Biomarcadores del Envejecimiento (https://www.agingconsortium.org) está examinando actualmente la aplicación de estos biomarcadores para identificar intervenciones de longevidad y formas de validarlas. [20] Además, los recursos de código abierto, como el paquete R methylCIPHER [21] y el paquete Python pyaging [22] están disponibles para el público como centros para varios biomarcadores del envejecimiento.
{{cite journal}}: Requiere citar revista |journal=( ayuda )