El algoritmo Smith-Waterman realiza un alineamiento de secuencias local , es decir, determina regiones similares entre dos cadenas de secuencias de ácidos nucleicos o secuencias de proteínas . En lugar de observar la secuencia completa , el algoritmo Smith-Waterman compara segmentos de todas las longitudes posibles y optimiza la medida de similitud .
El algoritmo fue propuesto por primera vez por Temple F. Smith y Michael S. Waterman en 1981. [1] Al igual que el algoritmo Needleman-Wunsch , del cual es una variación, Smith-Waterman es un algoritmo de programación dinámica . Como tal, tiene la propiedad deseable de que está garantizado que encontrará la alineación local óptima con respecto al sistema de puntuación que se esté utilizando (que incluye la matriz de sustitución y el esquema de puntuación de brecha ). La principal diferencia con el algoritmo Needleman-Wunsch es que las celdas de la matriz de puntuación negativa se establecen en cero. El procedimiento de rastreo comienza en la celda de la matriz de puntuación más alta y continúa hasta que se encuentra una celda con puntuación cero, lo que produce la alineación local de puntuación más alta. Debido a su complejidad de tiempo cúbico, a menudo no se puede aplicar prácticamente a problemas de gran escala y se reemplaza a favor de alternativas computacionalmente más eficientes como (Gotoh, 1982), [2] ( Altschul y Erickson, 1986), [3] y ( Myers y Miller, 1988). [4]
En 1970, Saul B. Needleman y Christian D. Wunsch propusieron un algoritmo de homología heurística para la alineación de secuencias, también conocido como algoritmo Needleman-Wunsch. [5] Es un algoritmo de alineación global que requiere pasos de cálculo ( y son las longitudes de las dos secuencias que se alinean). Utiliza el cálculo iterativo de una matriz con el fin de mostrar la alineación global. En la década siguiente, Sankoff, [6] Reichert, [7] Beyer [8] y otros formularon algoritmos heurísticos alternativos para analizar secuencias de genes. Sellers introdujo un sistema para medir distancias de secuencias. [9] En 1976, Waterman et al. añadieron el concepto de huecos al sistema de medición original. [10] En 1981, Smith y Waterman publicaron su algoritmo Smith-Waterman para calcular la alineación local.
El algoritmo Smith-Waterman es bastante exigente en cuanto al tiempo: para alinear dos secuencias de longitudes y , se requiere tiempo. Gotoh [2] y Altschul [3] optimizaron el algoritmo a pasos. Myers y Miller [4] optimizaron la complejidad espacial de a (lineal), donde es la longitud de la secuencia más corta, para el caso en el que solo se desea una de las muchas alineaciones óptimas posibles. Chowdhury, Le y Ramachandran [11] luego optimizaron el rendimiento de la caché del algoritmo mientras mantenían el uso del espacio lineal en la longitud total de las secuencias de entrada.
En los últimos años, los proyectos genómicos realizados en una variedad de organismos generaron cantidades masivas de datos de secuencias de genes y proteínas, lo que requiere un análisis computacional. La alineación de secuencias muestra las relaciones entre genes o entre proteínas, lo que conduce a una mejor comprensión de su homología y funcionalidad. La alineación de secuencias también puede revelar dominios y motivos conservados .
Una de las motivaciones para la alineación local es la dificultad de obtener alineaciones correctas en regiones de baja similitud entre secuencias biológicas distantemente relacionadas, porque las mutaciones han añadido demasiado "ruido" a lo largo del tiempo evolutivo para permitir una comparación significativa de esas regiones. La alineación local evita por completo dichas regiones y se centra en aquellas con una puntuación positiva, es decir, aquellas con una señal de similitud conservada evolutivamente. Un prerrequisito para la alineación local es una puntuación de expectativa negativa. La puntuación de expectativa se define como la puntuación promedio que el sistema de puntuación ( matriz de sustitución y penalizaciones por brecha ) produciría para una secuencia aleatoria.
Otra motivación para utilizar alineaciones locales es que existe un modelo estadístico confiable (desarrollado por Karlin y Altschul) para alineaciones locales óptimas. La alineación de secuencias no relacionadas tiende a producir puntuaciones de alineación local óptimas que siguen una distribución de valores extremos. Esta propiedad permite que los programas produzcan un valor esperado para la alineación local óptima de dos secuencias, que es una medida de la frecuencia con la que dos secuencias no relacionadas producirían una alineación local óptima cuya puntuación es mayor o igual que la puntuación observada. Valores de expectativa muy bajos indican que las dos secuencias en cuestión podrían ser homólogas , lo que significa que podrían compartir un ancestro común.
Sean y las secuencias a alinear, donde y son las longitudes de y respectivamente.
El algoritmo Smith-Waterman alinea dos secuencias mediante coincidencias o desajustes (también conocidos como sustituciones), inserciones y eliminaciones. Tanto las inserciones como las eliminaciones son operaciones que introducen espacios vacíos, que se representan mediante guiones. El algoritmo Smith-Waterman consta de varios pasos:
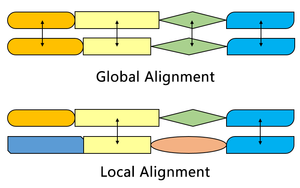
El algoritmo Smith-Waterman encuentra los segmentos en dos secuencias que tienen similitudes, mientras que el algoritmo Needleman-Wunsch alinea dos secuencias completas. Por lo tanto, tienen propósitos diferentes. Ambos algoritmos utilizan los conceptos de una matriz de sustitución, una función de penalización por espacios, una matriz de puntuación y un proceso de rastreo. Las tres diferencias principales son:
Una de las distinciones más importantes es que no se asigna ninguna puntuación negativa en el sistema de puntuación del algoritmo Smith-Waterman, lo que permite la alineación local. Cuando cualquier elemento tiene una puntuación inferior a cero, significa que las secuencias hasta esa posición no tienen similitudes; ese elemento se pondrá entonces a cero para eliminar la influencia de la alineación anterior. De esta manera, el cálculo puede continuar para encontrar la alineación en cualquier posición posteriormente.
La matriz de puntuación inicial del algoritmo Smith-Waterman permite la alineación de cualquier segmento de una secuencia con una posición arbitraria en la otra secuencia. Sin embargo, en el algoritmo Needleman-Wunsch, también se debe considerar la penalización por espacio en los extremos para alinear las secuencias completas.
A cada sustitución de base o de aminoácido se le asigna una puntuación. En general, a las coincidencias se les asigna una puntuación positiva y a las discordancias se les asigna una puntuación relativamente más baja. Tomemos como ejemplo la secuencia de ADN. Si las coincidencias obtienen +1, las discordancias obtienen -1, entonces la matriz de sustitución es:
Esta matriz de sustitución se puede describir como:
Diferentes sustituciones de bases o de aminoácidos pueden tener diferentes puntuaciones. La matriz de sustitución de aminoácidos suele ser más complicada que la de las bases. Véase PAM , BLOSUM .
La penalización por hueco designa puntuaciones para la inserción o eliminación. Una estrategia sencilla de penalización por hueco es utilizar una puntuación fija para cada hueco. Sin embargo, en biología, la puntuación debe contarse de forma diferente por razones prácticas. Por un lado, la similitud parcial entre dos secuencias es un fenómeno común; por otro lado, un único evento de mutación genética puede dar lugar a la inserción de un único hueco largo. Por lo tanto, los huecos conectados que forman un hueco largo suelen ser más favorecidos que los múltiples huecos cortos y dispersos. Para tener en cuenta esta diferencia, se han añadido los conceptos de apertura de hueco y extensión de hueco al sistema de puntuación. La puntuación de apertura de hueco suele ser superior a la puntuación de extensión de hueco. Por ejemplo, los parámetros predeterminados en EMBOSS Water son: apertura de hueco = 10, extensión de hueco = 0,5.
Aquí analizamos dos estrategias comunes para la penalización por brecha. Consulte Penalización por brecha para obtener más estrategias. Sea la función de penalización por brecha para una brecha de longitud :
Una penalización por brecha lineal tiene las mismas puntuaciones para abrir y extender una brecha:
,
¿Dónde está el costo de un solo espacio?
La penalización por brecha es directamente proporcional a la longitud de la brecha. Cuando se utiliza una penalización por brecha lineal, el algoritmo de Smith-Waterman se puede simplificar de la siguiente manera:
El algoritmo simplificado utiliza pasos. Cuando se puntúa un elemento, solo se deben tener en cuenta las penalizaciones por espacios de los elementos que están directamente adyacentes a este elemento.
Una penalización por brecha afín considera la apertura y la extensión de la brecha por separado:
,
donde es la penalización por apertura de hueco y es la penalización por extensión de hueco. Por ejemplo, la penalización por un hueco de longitud 2 es .
En el artículo original sobre el algoritmo Smith-Waterman se utilizó una penalización por brecha arbitraria. Utiliza pasos, por lo tanto, es bastante exigente en cuanto a tiempo. Gotoh optimizó los pasos para una penalización por brecha afín a , [2] pero el algoritmo optimizado solo intenta encontrar una alineación óptima, y no se garantiza que se encuentre la alineación óptima. [3] Altschul modificó el algoritmo de Gotoh para encontrar todas las alineaciones óptimas manteniendo la complejidad computacional. [3] Más tarde, Myers y Miller señalaron que el algoritmo de Gotoh y Altschul se puede modificar aún más según el método publicado por Hirschberg en 1975, [12] y aplicaron este método. [4] El algoritmo de Myers y Miller puede alinear dos secuencias utilizando el espacio, siendo la longitud de la secuencia más corta. Chowdhury, Le y Ramachandran [11] mostraron más tarde cómo ejecutar el algoritmo de Gotoh de manera eficiente en caché en el espacio lineal utilizando una estrategia recursiva de dividir y vencer diferente a la utilizada por Hirschberg. El algoritmo resultante se ejecuta más rápido que el algoritmo de Myers y Miller en la práctica debido a su rendimiento de caché superior. [11]
Tomemos como ejemplo la alineación de las secuencias TACGGGCCCGCTAC y TAGCCCTATCGGTCA . Cuando se utiliza la función de penalización por brecha lineal, el resultado es (Alineaciones realizadas por EMBOSS Water. La matriz de sustitución es DNAfull (puntuación de similitud: +5 para caracteres coincidentes, de lo contrario -4). La apertura y la extensión de la brecha son 0,0 y 1,0 respectivamente):
TACGGGCCCGCTA-CTA---G-CC-CTATC
Cuando se utiliza la penalización por espacio afín, el resultado es (la apertura y la extensión del espacio son 5,0 y 1,0 respectivamente):
TACGGGCCCGCTATA---CCG--CTA
Este ejemplo muestra que una penalización por espacio afín puede ayudar a evitar pequeños espacios dispersos.
La función de la matriz de puntuación es realizar comparaciones uno a uno entre todos los componentes de dos secuencias y registrar los resultados de alineación óptima. El proceso de puntuación refleja el concepto de programación dinámica. La alineación óptima final se encuentra expandiendo iterativamente la alineación óptima creciente. En otras palabras, la alineación óptima actual se genera al decidir qué camino (coincidencia/discordancia o inserción de espacio) brinda la puntuación más alta de la alineación óptima anterior. El tamaño de la matriz es la longitud de una secuencia más 1 por la longitud de la otra secuencia más 1. La primera fila y la primera columna adicionales sirven para alinear una secuencia con cualquier posición en la otra secuencia. Tanto la primera línea como la primera columna se establecen en 0 para que el espacio final no se penalice. La matriz de puntuación inicial es:
Tomemos como ejemplo la alineación de las secuencias de ADN TGTTACGG y GGTTGACTA . Utilice el siguiente esquema:
Inicialice y complete la matriz de puntuación, como se muestra a continuación. Esta figura muestra el proceso de puntuación de los primeros tres elementos. El color amarillo indica las bases que se están considerando. El color rojo indica la puntuación más alta posible para la celda que se está evaluando.
La matriz de puntuación finalizada se muestra a continuación a la izquierda. El color azul muestra la puntuación más alta. Un elemento puede recibir puntuación de más de un elemento, cada uno formará una ruta diferente si se rastrea este elemento. En caso de múltiples puntuaciones más altas, el rastreo debe realizarse comenzando con cada puntuación más alta. El proceso de rastreo se muestra a continuación a la derecha. La mejor alineación local se genera en la dirección inversa.
El resultado de la alineación es:
GTT - AC GTTGAC
Una implementación del algoritmo Smith-Waterman, SSEARCH, está disponible en el paquete de análisis de secuencias FASTA de UVA FASTA Downloads. Esta implementación incluye código acelerado de Altivec para procesadores PowerPC G4 y G5 que acelera las comparaciones entre 10 y 20 veces, utilizando una modificación del enfoque de Wozniak, 1997, [13] y una vectorización SSE2 desarrollada por Farrar [14], lo que hace que las búsquedas óptimas en bases de datos de secuencias de proteínas sean bastante prácticas. Una biblioteca, SSW, extiende la implementación de Farrar para devolver información de alineación además de la puntuación óptima de Smith-Waterman. [15]
Cray demostró la aceleración del algoritmo Smith-Waterman utilizando una plataforma informática reconfigurable basada en chips FPGA , con resultados que muestran una aceleración de hasta 28x sobre las soluciones basadas en microprocesadores estándar. Otra versión basada en FPGA del algoritmo Smith-Waterman muestra aceleraciones de FPGA (Virtex-4) de hasta 100x [16] sobre un procesador Opteron de 2,2 GHz. [17] Los sistemas TimeLogic DeCypher y CodeQuest también aceleran Smith-Waterman y Framesearch utilizando tarjetas FPGA PCIe.
Una tesis de maestría de 2011 [18] incluye un análisis de la aceleración Smith-Waterman basada en FPGA.
En una publicación de 2016, Código OpenCL compilado con Xilinx SDAccel acelera la secuenciación del genoma, supera el rendimiento de CPU/GPU/W en 12-21x, se presentó una implementación muy eficiente. Al utilizar una tarjeta FPGA PCIe equipada con un FPGA Xilinx Virtex-7 2000T, el nivel de rendimiento por vatio fue 12-21x mejor que el de CPU/GPU.
El Laboratorio Nacional Lawrence Livermore y el Instituto Conjunto del Genoma del Departamento de Energía de los Estados Unidos (EE. UU.) implementaron una versión acelerada de las búsquedas de alineamiento de secuencias locales de Smith-Waterman utilizando unidades de procesamiento gráfico (GPU) con resultados preliminares que muestran una aceleración dos veces mayor que las implementaciones de software. [19] Ya se ha implementado un método similar en el software Biofacet desde 1997, con el mismo factor de aceleración. [20]
También están disponibles varias implementaciones de GPU del algoritmo en la plataforma CUDA C de NVIDIA . [21] En comparación con la implementación de CPU más conocida (que utiliza instrucciones SIMD en la arquitectura x86), de Farrar, las pruebas de rendimiento de esta solución utilizando una sola tarjeta NVidia GeForce 8800 GTX muestran un ligero aumento en el rendimiento para secuencias más pequeñas, pero una ligera disminución en el rendimiento para secuencias más grandes. Sin embargo, las mismas pruebas que se ejecutan en tarjetas duales NVidia GeForce 8800 GTX son casi el doble de rápidas que la implementación de Farrar para todos los tamaños de secuencia probados.
Ahora está disponible una implementación más reciente de GPU CUDA de SW que es más rápida que las versiones anteriores y también elimina las limitaciones en las longitudes de consulta. Consulte CUDASW++.
Se han informado once implementaciones de SW diferentes en CUDA, tres de las cuales informan aceleraciones de 30X. [22]
Por último, se pueden encontrar otras implementaciones aceleradas por GPU del Smith-Waterman en NVIDIA Parabricks , la suite de software de NVIDIA para el análisis del genoma. [23]
En 2000, Rognes y Seeberg describieron en una publicación una implementación rápida del algoritmo Smith-Waterman utilizando la tecnología SIMD ( instrucción única, múltiples datos ) disponible en los procesadores Intel Pentium MMX y tecnologías similares. [24] A diferencia del enfoque de Wozniak (1997), la nueva implementación se basaba en vectores paralelos a la secuencia de consulta, no en vectores diagonales. La empresa Sencel Bioinformatics ha solicitado una patente que cubre este enfoque. Sencel está desarrollando aún más el software y proporciona ejecutables para uso académico de forma gratuita.
Ahora está disponible una vectorización SSE2 del algoritmo (Farrar, 2007) que proporciona una aceleración de 8 a 16 veces en procesadores Intel/AMD con extensiones SSE2. [14] Cuando se ejecuta en un procesador Intel que utiliza la microarquitectura Core, la implementación SSE2 logra un aumento de 20 veces. La implementación SSE2 de Farrar está disponible como el programa SSEARCH en el paquete de comparación de secuencias FASTA . SSEARCH está incluido en el conjunto de programas de búsqueda de similitudes del Instituto Europeo de Bioinformática .
La empresa danesa de bioinformática CLC bio ha logrado aceleraciones cercanas a 200 veces con respecto a las implementaciones de software estándar con SSE2 en una CPU Intel Core 2 Duo de 2,17 GHz, según un informe técnico disponible públicamente.
La versión acelerada del algoritmo Smith-Waterman, en servidores Linux basados en Intel y Advanced Micro Devices (AMD) , es compatible con el paquete GenCore 6, ofrecido por Biocceleration. Los puntos de referencia de rendimiento de este paquete de software muestran una aceleración de velocidad hasta diez veces mayor que la implementación de software estándar en el mismo procesador.
Actualmente, la única empresa en bioinformática que ofrece soluciones SSE y FPGA que aceleran Smith–Waterman, CLC bio ha logrado aceleraciones de más de 110 veces con respecto a las implementaciones de software estándar con CLC Bioinformatics Cube. [ cita requerida ]
La implementación más rápida del algoritmo en CPUs con SSSE3 se puede encontrar en el software SWIPE (Rognes, 2011), [25] que está disponible bajo la Licencia Pública General Affero de GNU . En paralelo, este software compara residuos de dieciséis secuencias de bases de datos diferentes con un residuo de consulta. Usando una secuencia de consulta de 375 residuos, se logró una velocidad de 106 mil millones de actualizaciones de celdas por segundo (GCUPS) en un sistema con procesador dual Intel Xeon X5650 de seis núcleos, que es más de seis veces más rápido que el software basado en el enfoque "striped" de Farrar. Es más rápido que BLAST cuando se usa la matriz BLOSUM50.
Una implementación de Smith–Waterman denominada diagonalsw , en C y C++ , utiliza conjuntos de instrucciones SIMD ( SSE4.1 para la plataforma x86 y AltiVec para la plataforma PowerPC). Se publica bajo una licencia MIT de código abierto .
En 2008, Farrar [26] describió un puerto del Striped Smith–Waterman [14] para el Cell Broadband Engine y reportó velocidades de 32 y 12 GCUPS en un blade IBM QS20 y una Sony PlayStation 3 , respectivamente.
La rápida expansión de los datos genéticos pone a prueba la velocidad de los algoritmos actuales de alineamiento de secuencias de ADN. Las necesidades esenciales de un método eficiente y preciso para el descubrimiento de variantes de ADN exigen enfoques innovadores para el procesamiento paralelo en tiempo real.
{{cite journal}}: CS1 maint: multiple names: authors list (link){{cite web}}: CS1 maint: archived copy as title (link), "Copia archivada" (PDF) . Archivado desde el original (PDF) el 5 de julio de 2008. Consultado el 17 de octubre de 2007 .{{cite web}}: CS1 maint: archived copy as title (link), y "Copia archivada" (PDF) . Archivado desde el original (PDF) el 20 de julio de 2011. Consultado el 17 de octubre de 2007 .{{cite web}}: CS1 maint: archived copy as title (link){{cite journal}}: Requiere citar revista |journal=( ayuda )