En el campo del modelado molecular , el acoplamiento es un método que predice la orientación preferida de una molécula con respecto a una segunda cuando un ligando y un objetivo están unidos entre sí para formar un complejo estable . [1] El conocimiento de la orientación preferida a su vez se puede utilizar para predecir la fuerza de asociación o la afinidad de unión entre dos moléculas utilizando, por ejemplo, funciones de puntuación .
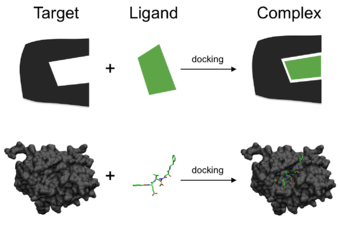
Las asociaciones entre moléculas biológicamente relevantes, como proteínas , péptidos , ácidos nucleicos , carbohidratos y lípidos , desempeñan un papel central en la transducción de señales . Además, la orientación relativa de los dos socios que interactúan puede afectar el tipo de señal producida (por ejemplo, agonismo vs. antagonismo ). Por lo tanto, el acoplamiento es útil para predecir tanto la fuerza como el tipo de señal producida.
El acoplamiento molecular es uno de los métodos más utilizados en el diseño de fármacos basado en la estructura , debido a su capacidad para predecir la conformación de unión de ligandos de moléculas pequeñas al sitio de unión objetivo adecuado . La caracterización del comportamiento de unión desempeña un papel importante en el diseño racional de fármacos , así como para dilucidar procesos bioquímicos fundamentales. [2] [3]
Se puede pensar en el acoplamiento molecular como un problema de “cerradura y llave” , en el que se quiere encontrar la orientación relativa correcta de la “llave” que abrirá la “cerradura” (en qué parte de la superficie de la cerradura está el orificio de la cerradura, en qué dirección girar la llave después de insertarla, etc.). Aquí, la proteína puede considerarse como la “cerradura” y el ligando como una “llave”. El acoplamiento molecular puede definirse como un problema de optimización, que describiría la orientación de “mejor ajuste” de un ligando que se une a una proteína particular de interés. Sin embargo, dado que tanto el ligando como la proteína son flexibles, una analogía de “mano en guante” es más apropiada que “cerradura y llave” . [4] Durante el proceso de acoplamiento, el ligando y la proteína ajustan su conformación para lograr un “mejor ajuste” general y este tipo de ajuste conformacional que resulta en la unión general se conoce como “ajuste inducido” . [5]
La investigación sobre acoplamiento molecular se centra en simular computacionalmente el proceso de reconocimiento molecular . Su objetivo es lograr una conformación optimizada tanto para la proteína como para el ligando y una orientación relativa entre la proteína y el ligando de modo que se minimice la energía libre del sistema general.
Hay dos enfoques particularmente populares dentro de la comunidad de acoplamiento molecular.
Ambos enfoques tienen ventajas significativas, pero también algunas limitaciones, que se describen a continuación.
Los métodos de complementariedad de forma/coincidencia geométrica describen la proteína y el ligando como un conjunto de características que los hacen acoplables. [10] Estas características pueden incluir descriptores de superficie molecular /superficie complementaria. En este caso, la superficie molecular del receptor se describe en términos de su área de superficie accesible al solvente y la superficie molecular del ligando se describe en términos de su descripción de superficie coincidente. La complementariedad entre las dos superficies equivale a la descripción de coincidencia de forma que puede ayudar a encontrar la pose complementaria de acoplamiento de las moléculas objetivo y ligando. Otro enfoque es describir las características hidrofóbicas de la proteína utilizando giros en los átomos de la cadena principal. Otro enfoque más es utilizar una técnica de descriptor de forma de Fourier. [11] [12] [13] Si bien los enfoques basados en la complementariedad de forma son típicamente rápidos y robustos, por lo general no pueden modelar los movimientos o cambios dinámicos en las conformaciones ligando/proteína con precisión, aunque los desarrollos recientes permiten que estos métodos investiguen la flexibilidad del ligando. Los métodos de complementariedad de formas pueden analizar rápidamente varios miles de ligandos en cuestión de segundos y determinar si pueden unirse al sitio activo de la proteína, y suelen ser escalables incluso para interacciones proteína-proteína. También son mucho más adecuados para los enfoques basados en farmacóforos , ya que utilizan descripciones geométricas de los ligandos para encontrar la unión óptima.
Simular el proceso de acoplamiento es mucho más complicado. En este enfoque, la proteína y el ligando están separados por una cierta distancia física, y el ligando encuentra su posición en el sitio activo de la proteína después de una cierta cantidad de "movimientos" en su espacio conformacional. Los movimientos incorporan transformaciones de cuerpo rígido, como traslaciones y rotaciones, así como cambios internos en la estructura del ligando, incluidas rotaciones de ángulos de torsión. Cada uno de estos movimientos en el espacio conformacional del ligando induce un costo energético total del sistema. Por lo tanto, la energía total del sistema se calcula después de cada movimiento.
La ventaja obvia de la simulación de acoplamiento es que la flexibilidad de los ligandos se incorpora fácilmente, mientras que las técnicas de complementariedad de formas deben utilizar métodos ingeniosos para incorporar la flexibilidad en los ligandos. Además, modela la realidad con mayor precisión, mientras que las técnicas de complementariedad de formas son más una abstracción.
Es evidente que la simulación es costosa en términos computacionales, ya que se debe explorar un gran panorama energético. Las técnicas basadas en la red, los métodos de optimización y la mayor velocidad de los ordenadores han hecho que la simulación de acoplamiento sea más realista.
Para realizar un análisis de acoplamiento, el primer requisito es una estructura de la proteína de interés. Por lo general, la estructura se ha determinado mediante una técnica biofísica como
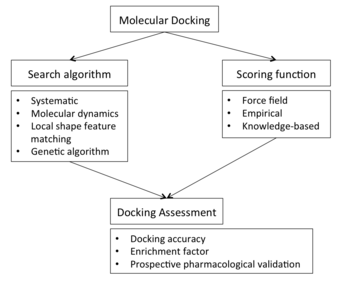
pero también puede derivarse de la construcción de modelos de homología . Esta estructura de proteína y una base de datos de ligandos potenciales sirven como entradas para un programa de acoplamiento. El éxito de un programa de acoplamiento depende de dos componentes: el algoritmo de búsqueda y la función de puntuación .
En teoría, el espacio de búsqueda consta de todas las posibles orientaciones y conformaciones de la proteína emparejada con el ligando. Sin embargo, en la práctica, con los recursos computacionales actuales, es imposible explorar exhaustivamente el espacio de búsqueda; esto implicaría enumerar todas las posibles distorsiones de cada molécula (las moléculas son dinámicas y existen en un conjunto de estados conformacionales) y todas las posibles orientaciones rotacionales y translacionales del ligando en relación con la proteína en un nivel dado de granularidad . La mayoría de los programas de acoplamiento en uso tienen en cuenta todo el espacio conformacional del ligando (ligando flexible) y varios intentan modelar un receptor proteico flexible. Cada "instantánea" del par se denomina pose . [14]
Se han aplicado diversas estrategias de búsqueda conformacional al ligando y al receptor, entre ellas:
Las conformaciones del ligando se pueden generar en ausencia del receptor y posteriormente acoplarse [15] o se pueden generar conformaciones sobre la marcha en presencia de la cavidad de unión del receptor, [16] o con total flexibilidad rotacional de cada ángulo diedro utilizando acoplamiento basado en fragmentos. [17] La evaluación de la energía del campo de fuerza se utiliza con mayor frecuencia para seleccionar conformaciones energéticamente razonables, [18] pero también se han utilizado métodos basados en el conocimiento. [19]
Los péptidos son moléculas muy flexibles y de tamaño relativamente grande, lo que hace que modelar su flexibilidad sea una tarea difícil. Se desarrollaron varios métodos para permitir un modelado eficiente de la flexibilidad de los péptidos durante el acoplamiento proteína-péptido. [20]
La capacidad computacional ha aumentado drásticamente durante la última década, lo que ha hecho posible el uso de métodos más sofisticados y computacionalmente intensivos en el diseño de fármacos asistido por computadora. Sin embargo, abordar la flexibilidad del receptor en las metodologías de acoplamiento sigue siendo un tema espinoso. [21] La principal razón detrás de esta dificultad es el gran número de grados de libertad que deben considerarse en este tipo de cálculos. Sin embargo, descuidarlo, en algunos casos, puede conducir a malos resultados de acoplamiento en términos de predicción de la posición de unión. [22]
A menudo se utilizan múltiples estructuras estáticas determinadas experimentalmente para la misma proteína en diferentes conformaciones para emular la flexibilidad del receptor. [23] Alternativamente, se pueden buscar bibliotecas de rotámeros de cadenas laterales de aminoácidos que rodean la cavidad de unión para generar conformaciones de proteínas alternativas pero energéticamente razonables. [24] [25]
Los programas de acoplamiento generan una gran cantidad de posibles posiciones de ligando, de las cuales algunas pueden rechazarse inmediatamente debido a colisiones con la proteína. El resto se evalúa utilizando una función de puntuación, que toma una posición como entrada y devuelve un número que indica la probabilidad de que la posición represente una interacción de unión favorable y clasifica un ligando en relación con otro.
La mayoría de las funciones de puntuación son campos de fuerza de mecánica molecular basados en la física que estiman la energía de la posición dentro del sitio de unión. Las diversas contribuciones a la unión se pueden escribir como una ecuación aditiva:
Los componentes consisten en efectos del solvente, cambios conformacionales en la proteína y el ligando, energía libre debido a interacciones proteína-ligando, rotaciones internas, energía de asociación del ligando y el receptor para formar un solo complejo y energía libre debido a cambios en los modos vibracionales. [26] Una energía baja (negativa) indica un sistema estable y, por lo tanto, una probable interacción de enlace.
Los enfoques alternativos utilizan funciones de puntuación modificadas para incluir restricciones basadas en interacciones proteína-ligando clave conocidas, [27] o potenciales basados en el conocimiento derivados de interacciones observadas en grandes bases de datos de estructuras proteína-ligando (por ejemplo, el Protein Data Bank ). [28]
Existe una gran cantidad de estructuras obtenidas mediante cristalografía de rayos X para complejos entre proteínas y ligandos de alta afinidad, pero comparativamente menos para ligandos de baja afinidad, ya que estos últimos complejos tienden a ser menos estables y, por lo tanto, más difíciles de cristalizar. Las funciones de puntuación entrenadas con estos datos pueden acoplar correctamente los ligandos de alta afinidad, pero también darán conformaciones acopladas plausibles para ligandos que no se unen. Esto da una gran cantidad de resultados positivos falsos , es decir, ligandos que se predice que se unirán a la proteína pero que en realidad no lo hacen cuando se colocan juntos en un tubo de ensayo.
Una forma de reducir el número de falsos positivos es recalcular la energía de las poses con mayor puntuación utilizando técnicas (potencialmente) más precisas pero computacionalmente más intensivas, como los métodos de Born generalizado o Poisson-Boltzmann . [9]
La interdependencia entre la función de muestreo y la de puntuación afecta la capacidad de acoplamiento para predecir posiciones plausibles o afinidades de unión para compuestos nuevos. Por lo tanto, generalmente se requiere una evaluación de un protocolo de acoplamiento (cuando se dispone de datos experimentales) para determinar su capacidad predictiva. La evaluación del acoplamiento se puede realizar utilizando diferentes estrategias, como:
La precisión de acoplamiento [30] [31] representa una medida para cuantificar la aptitud de un programa de acoplamiento al racionalizar la capacidad de predecir la postura correcta de un ligando con respecto a la observada experimentalmente. [32]
Las pantallas de acoplamiento también pueden evaluarse mediante el enriquecimiento de ligandos anotados de aglutinantes conocidos de entre una gran base de datos de moléculas “ señuelo ” presuntamente no aglutinantes. [29] De esta manera, el éxito de una pantalla de acoplamiento se evalúa por su capacidad para enriquecer el pequeño número de compuestos activos conocidos en los primeros puestos de una pantalla de entre un número mucho mayor de moléculas señuelo en la base de datos. El área bajo la curva de la característica operativa del receptor (ROC) se utiliza ampliamente para evaluar su rendimiento.
Los resultados de los análisis de acoplamiento se someten a una validación farmacológica (por ejemplo, mediciones de IC50 , afinidad o potencia ). Solo los estudios prospectivos constituyen una prueba concluyente de la idoneidad de una técnica para un objetivo en particular. [33] En el caso de los receptores acoplados a proteína G (GPCR), que son objetivos de más del 30% de los fármacos comercializados, el acoplamiento molecular condujo al descubrimiento de más de 500 ligandos de GPCR. [34]
El potencial de los programas de acoplamiento para reproducir modos de enlace determinados por cristalografía de rayos X se puede evaluar mediante una variedad de conjuntos de referencia de acoplamiento.
Para moléculas pequeñas, existen varios conjuntos de datos de referencia para acoplamiento y cribado virtual, por ejemplo, Astex Diverse Set, que consiste en estructuras cristalinas de rayos X de proteína-ligando de alta calidad, [35] el Directory of Useful Decoys (DUD) para la evaluación del rendimiento del cribado virtual, [29] o el conjunto de datos LEADS-FRAG para fragmentos [36].
Se puede realizar una evaluación de los programas de acoplamiento para determinar su potencial para reproducir modos de unión de péptidos mediante Lecciones para la evaluación de la eficiencia del acoplamiento y la puntuación (LEADS-PEP). [37]
Una interacción de unión entre un ligando de molécula pequeña y una proteína enzimática puede dar como resultado la activación o inhibición de la enzima. Si la proteína es un receptor, la unión del ligando puede dar como resultado agonismo o antagonismo . El acoplamiento se utiliza con mayor frecuencia en el campo del diseño de fármacos : la mayoría de los fármacos son moléculas orgánicas pequeñas y el acoplamiento se puede aplicar a:
de Internet que calcula el sitio, la geometría y la energía de las moléculas pequeñas que interactúan con las proteínas.