La recombinación V(D)J (reordenamiento de unión de variable-diversidad) es el mecanismo de recombinación somática que ocurre sólo en los linfocitos en desarrollo durante las primeras etapas de la maduración de las células T y B. Da como resultado un repertorio muy diverso de anticuerpos/inmunoglobulinas y receptores de células T (TCR) que se encuentran en las células B y T , respectivamente. El proceso es una característica definitoria del sistema inmunológico adaptativo .
La recombinación de V(D)J en los mamíferos ocurre en los órganos linfoides primarios ( médula ósea para las células B y timo para las células T) y de manera casi aleatoria reorganiza la variable (V), la unión (J) y, en algunos casos, la diversidad ( D) segmentos de genes. En última instancia, el proceso da como resultado nuevas secuencias de aminoácidos en las regiones de unión a antígenos de las inmunoglobulinas y TCR que permiten el reconocimiento de antígenos de casi todos los patógenos, incluidas bacterias , virus , parásitos y gusanos , así como "células propias alteradas", como se ve en cáncer . El reconocimiento también puede ser de naturaleza alérgica ( por ejemplo , al polen u otros alérgenos ) o puede coincidir con los tejidos del huésped y conducir a autoinmunidad .
En 1987, Susumu Tonegawa recibió el Premio Nobel de Fisiología o Medicina "por su descubrimiento del principio genético para la generación de diversidad de anticuerpos". [1]
Las moléculas de anticuerpos humanos (incluidos los receptores de células B ) están compuestas de cadenas pesadas y ligeras, cada una de las cuales contiene regiones constantes (C) y variables (V), codificadas genéticamente en tres loci :
Cada gen de cadena pesada o cadena ligera contiene múltiples copias de tres tipos diferentes de segmentos de genes para las regiones variables de las proteínas del anticuerpo. Por ejemplo, la región de la cadena pesada de la inmunoglobulina humana contiene 2 segmentos de genes constantes (Cμ y Cδ) y 44 segmentos de genes variables (V), además de 27 segmentos de genes de diversidad (D) y 6 segmentos de genes de unión (J). [2] Los genes de cadena ligera poseen un solo segmento de gen constante (Cκ) o cuatro (Cλ) con numerosos segmentos de genes V y J, pero no tienen segmentos de genes D. [3] El reordenamiento del ADN hace que una copia de cada tipo de segmento genético vaya a cualquier linfocito, generando un enorme repertorio de anticuerpos; Son posibles aproximadamente 3×10 11 combinaciones, aunque algunas se eliminan debido a la autorreactividad.
La mayoría de los receptores de células T están compuestos por una cadena alfa variable y una cadena beta. Los genes del receptor de células T son similares a los genes de inmunoglobulinas en que también contienen múltiples segmentos de genes V, D y J en sus cadenas beta (y segmentos de genes V y J en sus cadenas alfa) que se reordenan durante el desarrollo del linfocito para formar proporcionar a esa célula un receptor de antígeno único. El receptor de células T en este sentido es el equivalente topológico de un fragmento del anticuerpo que se une al antígeno, siendo ambos parte de la superfamilia de las inmunoglobulinas.
Se previene una respuesta autoinmune eliminando células que reaccionan espontáneamente. Esto ocurre en el timo al probar la célula contra una serie de autoantígenos expresados a través de la función del regulador autoinmune (AIRE). El locus de la cadena ligera lambda de inmunoglobulina contiene genes codificadores de proteínas que pueden perderse con su reordenamiento. Esto se basa en un mecanismo fisiológico y no es patogénico en leucemias o linfomas. Una célula persiste si crea un producto exitoso que no reacciona automáticamente; de lo contrario, se poda mediante apoptosis .
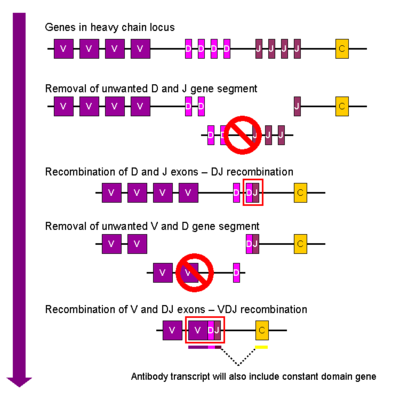
En la célula B en desarrollo , el primer evento de recombinación que ocurre es entre un segmento de gen D y uno J del locus de la cadena pesada. Se elimina cualquier ADN entre estos dos segmentos de genes. A esta recombinación DJ le sigue la unión de un segmento del gen V, de una región aguas arriba del complejo DJ recién formado, formando un segmento del gen VDJ reordenado. Todos los demás segmentos de genes entre los segmentos V y D ahora se eliminan del genoma de la célula. Se genera un transcrito primario (ARN no empalmado) que contiene la región VDJ de la cadena pesada y las cadenas mu y delta constantes (C μ y C δ ). (es decir, la transcripción primaria contiene los segmentos: VDJC μ -C δ ). El ARN primario se procesa para agregar una cola poliadenilada (poli-A) después de la cadena C μ y para eliminar la secuencia entre el segmento VDJ y este segmento genético constante. La traducción de este ARNm conduce a la producción de la proteína de cadena pesada IgM .
Las cadenas kappa (κ) y lambda (λ) de los loci de cadenas ligeras de inmunoglobulinas se reorganizan de forma muy similar, excepto que las cadenas ligeras carecen de un segmento D. En otras palabras, el primer paso de la recombinación de las cadenas ligeras implica la unión de las cadenas V y J para dar un complejo VJ antes de la adición del gen de la cadena constante durante la transcripción primaria. La traducción del ARNm empalmado para las cadenas kappa o lambda da como resultado la formación de la proteína de cadena ligera de Ig κ o Ig λ.
El ensamblaje de la cadena pesada de Ig μ y una de las cadenas ligeras da como resultado la formación de una forma unida a la membrana de la inmunoglobulina IgM que se expresa en la superficie de la célula B inmadura.
Durante el desarrollo de los timocitos , las cadenas del receptor de células T (TCR) experimentan esencialmente la misma secuencia de eventos de recombinación ordenada que la descrita para las inmunoglobulinas. La recombinación D a J ocurre primero en la cadena β del TCR. Este proceso puede implicar la unión del segmento del gen D β 1 a uno de los seis segmentos J β 1 o la unión del segmento del gen D β 2 a uno de los seis segmentos J β 2. [3] La recombinación de DJ se sigue (como anteriormente) con reordenamientos de V β a D β J β . Todos los segmentos de genes entre los segmentos de genes Vβ - Dβ - Jβ en el complejo recién formado se eliminan y se sintetiza el transcrito primario que incorpora el gen de dominio constante (Vβ - Dβ - Jβ - Cβ ) . La transcripción de ARNm corta cualquier secuencia intermedia y permite la traducción de la proteína de longitud completa para la cadena β de TCR.
El reordenamiento de la cadena alfa (α) del TCR sigue al reordenamiento de la cadena β y se asemeja al reordenamiento de V a J descrito para las cadenas ligeras de Ig (ver arriba). El ensamblaje de las cadenas β y α da como resultado la formación del αβ-TCR que se expresa en la mayoría de las células T.
El proceso de recombinación V(D)J está mediado por la recombinasa VDJ, que es un conjunto diverso de enzimas. Las enzimas clave implicadas son los genes activadores de recombinación 1 y 2 (RAG), la desoxinucleotidil transferasa terminal (TdT) y la Artemis nucleasa, un miembro de la ubicua vía de unión de extremos no homólogos (NHEJ) para la reparación del ADN. [4] Se sabe que varias otras enzimas participan en el proceso e incluyen la proteína quinasa dependiente de ADN (DNA-PK), la proteína 4 complementaria de reparación de rayos X (XRCC4), la ADN ligasa IV , la unión de extremos no homólogos factor 1 (NHEJ1; también conocido como Cernunnos o factor similar a XRCC4 [XLF]), el parálogo recientemente descubierto de XRCC4 y XLF (PAXX) y ADN polimerasas λ y μ. [5] Algunas enzimas implicadas son específicas de los linfocitos ( p. ej. , RAG, TdT), mientras que otras se encuentran en otros tipos de células e incluso de forma ubicua ( p . ej ., componentes de NHEJ).
Para mantener la especificidad de la recombinación, la recombinasa V(D)J reconoce y se une a secuencias señal de recombinación (RSS) que flanquean los segmentos de los genes variable (V), de diversidad (D) y de unión (J). Los RSS se componen de tres elementos: un heptámero de siete nucleótidos conservados, una región espaciadora de 12 o 23 pares de bases de longitud y un no nombre de nueve nucleótidos conservados. Si bien la mayoría de los RSS varían en secuencia, las secuencias consenso de heptámero y no nombre son CACAGTG y ACAAAAACC, respectivamente; y aunque la secuencia de la región espaciadora está poco conservada, la longitud está muy conservada. [6] [7] La longitud de la región espaciadora corresponde aproximadamente a una (12 pares de bases) o dos vueltas (23 pares de bases) de la hélice del ADN. Siguiendo lo que se conoce como la regla 12/23, los segmentos de genes que se van a recombinar suelen ser adyacentes a RSS de diferentes longitudes de espaciador ( es decir , uno tiene un "12RSS" y el otro tiene un "23RSS"). [8] Esta es una característica importante en la regulación de la recombinación V(D)J. [9]
La recombinación V(D)J comienza cuando la recombinasa V(D)J (a través de la actividad de RAG1) se une a un RSS que flanquea un segmento de gen codificante (V, D o J) y crea una mella monocatenaria en el ADN entre la primera base del RSS (justo antes del heptámero) y el segmento codificante. Esto es esencialmente neutral desde el punto de vista energético (no necesita hidrólisis de ATP ) y da como resultado la formación de un grupo hidroxilo 3' libre y un grupo fosfato 5' en la misma cadena. La recombinasa posiciona el grupo hidroxilo reactivo para atacar el enlace fosfodiéster de la cadena opuesta, formando dos extremos del ADN: una horquilla (tallo-bucle) en el segmento codificante y un extremo romo en el segmento señal. [10] El modelo actual es que el corte del ADN y la formación de horquillas se producen en ambas hebras simultáneamente (o casi) en un complejo conocido como centro de recombinación . [11] [12] [13] [14]
Los extremos romos de la señal se ligan al ras para formar una pieza circular de ADN que contiene todas las secuencias intermedias entre los segmentos codificantes conocida como unión de señal (aunque de naturaleza circular, no debe confundirse con un plásmido ). Si bien originalmente se pensaba que se perdían durante las sucesivas divisiones celulares, existe evidencia de que las uniones de señales pueden volver a ingresar al genoma y provocar patologías al activar oncogenes o interrumpir las funciones de los genes supresores de tumores [Ref].
Los extremos codificantes se procesan aún más antes de su ligamiento mediante varios eventos que finalmente conducen a la diversidad de unión. [15] El procesamiento comienza cuando DNA-PK se une a cada extremo del ADN roto y recluta varias otras proteínas, incluidas Artemisa, XRCC4, ADN ligasa IV, Cernunnos y varias ADN polimerasas. [16] DNA-PK forma un complejo que conduce a su autofosforilación , lo que resulta en la activación de Artemisa. Las horquillas del extremo codificante se abren gracias a la actividad de Artemisa. [17] Si se abren por el centro, el resultado será un extremo de ADN romo; sin embargo, en muchos casos, la apertura está "descentrada" y da como resultado que queden bases adicionales en una hebra (un saliente). Estos se conocen como nucleótidos palindrómicos (P) debido a la naturaleza palindrómica de la secuencia producida cuando las enzimas reparadoras del ADN resuelven el saliente. [18] El proceso de apertura de la horquilla por parte de Artemisa es un paso crucial de la recombinación de V(D)J y es defectuoso en el modelo de ratón con inmunodeficiencia combinada grave (scid) .
A continuación, XRCC4, Cernunnos y DNA-PK alinean los extremos del ADN y reclutan la desoxinucleotidil transferasa terminal (TdT), una ADN polimerasa independiente de la plantilla que agrega nucleótidos sin plantilla (N) al extremo codificante. La adición es mayoritariamente aleatoria, pero la TdT muestra preferencia por los nucleótidos G/C. [19] Como ocurre con todas las ADN polimerasas conocidas, la TdT agrega nucleótidos a una cadena en una dirección de 5' a 3'. [20]
Por último, las exonucleasas pueden eliminar bases de los extremos codificantes (incluido cualquier nucleótido P o N que pueda haberse formado). Las ADN polimerasas λ y μ luego insertan nucleótidos adicionales según sea necesario para que los dos extremos sean compatibles para la unión. Este es un proceso estocástico, por lo tanto, puede ocurrir cualquier combinación de la adición de nucleótidos P y N y la eliminación exonucleolítica (o ninguna). Finalmente, los extremos codificantes procesados se ligan entre sí mediante la ADN ligasa IV. [21]
Todos estos eventos de procesamiento dan como resultado un paratopo que es altamente variable, incluso cuando se recombinan los mismos segmentos de genes. La recombinación V(D)J permite la generación de inmunoglobulinas y receptores de células T para antígenos que ni el organismo ni sus ancestros necesitan haber encontrado previamente, lo que permite una respuesta inmune adaptativa a nuevos patógenos que se desarrollan o a aquellos que frecuentemente cambio ( p. ej. , gripe estacional ). Sin embargo, una advertencia importante para este proceso es que la secuencia de ADN debe permanecer en el marco para mantener la secuencia de aminoácidos correcta en el producto proteico final. Si la secuencia resultante está fuera de marco, el desarrollo de la célula se detendrá y la célula no sobrevivirá hasta la madurez. Por lo tanto, la recombinación V(D)J es un proceso muy costoso que debe estar (y está) estrictamente regulado y controlado.