Sedoreoviridae (anteriormente Reoviridae ) es una familia de virus de ARN bicatenario . Los virus miembros tienen una amplia gama de huéspedes , incluidos vertebrados , invertebrados , plantas, protistas y hongos. [1] Carecen de envolturas lipídicas y empaquetan su genoma segmentado dentro de cápsides de varias capas . La falta de una envoltura lipídica ha permitido obtener estructuras tridimensionales de estos grandes virus complejos (diámetro ~60–100 nm ), lo que revela una relación estructural y probablemente evolutiva con la familia de bacteriófagos cistovirus . [2] Actualmente hay 97 especies en esta familia, divididas en 15 géneros en dos subfamilias. [3] Los reovirus pueden afectar el sistema gastrointestinal (como los rotavirus ) y el tracto respiratorio . [ 4] El nombre "reo-" es un acrónimo de " virus huérfanos entéricos respiratorios " . [5] El término " virus huérfano " se refiere al hecho de que se ha observado que algunos de estos virus no están asociados con ninguna enfermedad conocida. Aunque más recientemente se han identificado virus de la familia Reoviridae con diversas enfermedades, todavía se utiliza el nombre original.
Las infecciones por reovirus ocurren con frecuencia en humanos, pero la mayoría de los casos son leves o subclínicos. Los rotavirus , sin embargo, pueden causar diarrea grave y malestar intestinal en los niños, y estudios de laboratorio en ratones han implicado a los ortoreovirus en la expresión de la enfermedad celíaca en individuos predispuestos. [6] El virus se puede detectar fácilmente en las heces y también se puede recuperar de las secreciones faríngeas o nasales , la orina, el líquido cefalorraquídeo y la sangre. A pesar de la facilidad para encontrar reovirus en muestras clínicas, su papel en las enfermedades o tratamientos humanos aún es incierto.
Algunos virus de esta familia, como los fitoreovirus y los oryzavirus , infectan a las plantas. La mayoría de los reovirus que infectan plantas se transmiten entre plantas a través de insectos vectores . Los virus se replican tanto en la planta como en el insecto, y generalmente causan enfermedades en la planta, pero poco o ningún daño al insecto infectado. [7] : 148
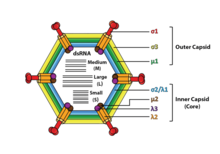
Los reovirus no tienen envoltura y tienen una cápside icosaédrica compuesta por una cubierta proteica externa ( T =13) e interna (T=2). [1] [8] Los estudios de ultraestructura muestran que las cápsides de viriones están compuestas de dos o tres capas separadas, lo que depende del tipo de especie. La capa más interna (núcleo) tiene simetría icosaédrica T=1 y está compuesta por 60 tipos diferentes de proteínas estructurales. El núcleo contiene los segmentos del genoma, cada uno de ellos codifica una variedad de estructura enzimática necesaria para la transcripción. El núcleo está cubierto por una capa de cápside con simetría icosaédrica T = 13. Los reovirus tienen una estructura única que contiene una proteína de pico glicolizada en la superficie. [9]
Los genomas de los virus de la familia Reoviridae contienen de 9 a 12 segmentos que se agrupan en tres categorías correspondientes a su tamaño: L (grande), M (mediano) y S (pequeño). Los segmentos varían de aproximadamente 0,2 a 3 kpb y cada segmento codifica de 1 a 3 proteínas (de 10 a 14 proteínas en total [1] ). Las proteínas de los virus de la familia Reoviridae se indican con el carácter griego correspondiente al segmento del que se tradujeron (el segmento L codifica proteínas λ, el segmento M codifica proteínas μ y el segmento S codifica proteínas σ). [8]

Los virus de la familia Reoviridae tienen genomas que consisten en ARN bicatenario (dsRNA) segmentado. [4] Debido a esto, la replicación ocurre exclusivamente en el citoplasma y el virus codifica varias proteínas que son necesarias para la replicación y conversión del genoma del ARNbc en ARN de sentido positivo. [10]
El virus puede ingresar a la célula huésped a través de un receptor en la superficie celular. Se desconoce el receptor, pero se cree que incluye ácido siálico y moléculas de adhesión de unión (JAM). [10] El virus no está parcialmente recubierto por proteasas en el endolisosoma, donde la cápside se digiere parcialmente para permitir una mayor entrada celular. Luego, la partícula central ingresa al citoplasma mediante un proceso aún desconocido en el que el genoma se transcribe de manera conservadora, lo que provoca un exceso de hebras de sentido positivo, que se utilizan como plantillas de ARN mensajero para sintetizar hebras de sentido negativo. [10]
El genoma del rotavirus se divide en 11 segmentos. Estos segmentos están asociados con la molécula VP1 que es responsable de la síntesis de ARN. En eventos tempranos, el proceso de selección ocurre para que la entrada de los 11 segmentos diferentes de ARN vaya a la célula. Este procedimiento lo realizan los ARN recién sintetizados. Este evento garantiza que se reciba uno de cada uno de los 11 segmentos de ARN diferentes. En eventos tardíos, el proceso de transcripción ocurre nuevamente, pero este tiempo no está limitado a diferencia de los eventos tempranos. Para los virus se requieren diferentes cantidades de ARN, por lo que durante el paso de traducción existe una maquinaria de control. Hay la misma cantidad de segmentos de ARN pero diferente cantidad de proteínas. La razón de esto es que los segmentos de ARN no se traducen al mismo ritmo. [7]
Las partículas virales comienzan a acumularse en el citoplasma entre 6 y 7 horas después de la infección. La traducción se lleva a cabo mediante escaneo con fugas, supresión de la terminación y omisión ribosómica . El virus sale de la célula huésped mediante un movimiento viral monopartito no guiado por túbulos, movimiento de célula a célula y existe en cuerpos de oclusión después de la muerte celular y permanece infeccioso hasta encontrar otro huésped. [1]
La reactivación de multiplicidad (MR) es el proceso mediante el cual dos o más genomas de virus, cada uno de los cuales contiene daño genómico inactivante, pueden interactuar dentro de una célula infectada para formar un genoma de virus viable. McClain y Spendlove [11] demostraron RM para tres tipos de reovirus después de la exposición a irradiación ultravioleta. En sus experimentos, se expusieron partículas de reovirus a dosis de luz ultravioleta que serían letales en infecciones individuales. Sin embargo, cuando se permitió que dos o más virus inactivados infectaran células huésped individuales, se produjo MR y se produjo una progenie viable. Como afirmaron, la reactivación de la multiplicidad por definición implica algún tipo de reparación. Michod et al. [12] revisaron numerosos ejemplos de RM en diferentes virus y sugirieron que la RM es una forma común de interacción sexual en virus que proporciona el beneficio de la reparación recombinacional de los daños del genoma. [ cita necesaria ]
La familia Reoviridae se divide en dos subfamilias [13] según la presencia de una proteína "torreta" en la cápside interna. [14] [15] De comunicaciones de ICTV: "El nombre Spinareovirinae se utilizará para identificar la subfamilia que contiene los virus con puntas o torretas y se deriva de 'reovirus' y la palabra latina 'spina' como prefijo, que significa punta, que denota la presencia de púas o torretas en la superficie de las partículas centrales. El término "púas" es una alternativa a "torretas", que se utilizó en las primeras investigaciones para describir la estructura de la partícula, particularmente con los cypovirus. El nombre Sedoreovirinae usarse para identificar la subfamilia que contiene los géneros de virus sin torretas y se deriva de 'reovirus' y de la palabra latina 'sedo', que significa suave, y denota la ausencia de púas o torretas en las partículas centrales de estos virus, que tienen una Morfología relativamente suave." [dieciséis]

La familia Reoviridae se divide en las siguientes subfamilias y géneros:
Aunque los reovirus en su mayoría no son patógenos en humanos, estos virus han servido como modelos experimentales muy productivos para estudios de patogénesis viral . [17] Los ratones recién nacidos son extremadamente sensibles a las infecciones por reovirus y se han utilizado como sistema experimental preferido para estudios de patogénesis de reovirus. [2]
Se ha demostrado que los reovirus tienen propiedades oncolíticas (que matan el cáncer), lo que fomenta el desarrollo de terapias basadas en reovirus para el tratamiento del cáncer. [18] [19] [20]
Reolysin es una formulación de reovirus ( cepa de serotipo 3 de ortoreovirus de mamíferos [21] ) que se encuentra actualmente en ensayos clínicos para el tratamiento de diversos cánceres, [22] incluidos estudios actualmente desarrollados para investigar el papel de Reolysin combinado con otras inmunoterapias. [21]