En bioinformática , el ensamblaje de secuencias se refiere a la alineación y fusión de fragmentos de una secuencia de ADN más larga para reconstruir la secuencia original. [1] Esto es necesario ya que la tecnología de secuenciación de ADN podría no ser capaz de "leer" genomas completos de una sola vez, sino que lee pequeños fragmentos de entre 20 y 30.000 bases, dependiendo de la tecnología utilizada. [1] Por lo general, los fragmentos cortos (lecturas) resultan de la secuenciación shotgun de ADN genómico o transcripción genética ( EST ). [1]
El problema de ensamblar secuencias puede compararse con tomar muchas copias de un libro, pasar cada una de ellas por una trituradora con un cortador diferente y volver a unir el texto del libro con solo mirar los trozos triturados. Además de la dificultad obvia de esta tarea, existen algunos problemas prácticos adicionales: el original puede tener muchos párrafos repetidos y algunos fragmentos pueden modificarse durante la trituración para que tengan errores tipográficos. También pueden agregarse extractos de otro libro y algunos fragmentos pueden ser completamente irreconocibles.
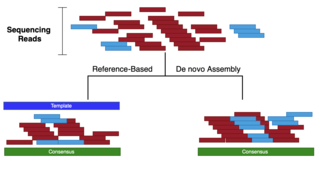
Hay tres enfoques para recopilar datos de secuenciación:
El ensamblaje guiado por referencia es una combinación de los otros tipos. Este tipo se aplica en lecturas largas para imitar las ventajas de las lecturas cortas (es decir, la calidad de la llamada). La lógica detrás de esto es agrupar las lecturas en ventanas más pequeñas dentro de la referencia. Las lecturas en cada grupo se reducirán en tamaño utilizando el enfoque k-mere para seleccionar la de mayor calidad y la más probable contigua (contig). Luego, los contigs se unirán para crear un andamiaje. El sentido final se logra cerrando los espacios en el andamiaje.
Los primeros ensambladores de secuencias comenzaron a aparecer a finales de los años 1980 y principios de los años 1990 como variantes de programas de alineamiento de secuencias más simples para unir grandes cantidades de fragmentos generados por instrumentos de secuenciación automática llamados secuenciadores de ADN . [2] A medida que los organismos secuenciados crecieron en tamaño y complejidad (desde pequeños virus hasta plásmidos, bacterias y finalmente eucariotas ), los programas de ensamblaje utilizados en estos proyectos genómicos necesitaban estrategias cada vez más sofisticadas para manejar:
Ante el desafío de ensamblar los primeros genomas eucariotas más grandes (la mosca de la fruta Drosophila melanogaster en 2000 y el genoma humano solo un año después), los científicos desarrollaron ensambladores como Celera Assembler [4] y Arachne [5] capaces de manejar genomas de 130 millones (por ejemplo, la mosca de la fruta D. melanogaster ) a 3 mil millones (por ejemplo, el genoma humano) de pares de bases. Después de estos esfuerzos, varios otros grupos, principalmente en los principales centros de secuenciación de genomas, construyeron ensambladores a gran escala, y se lanzó un esfuerzo de código abierto conocido como AMOS [6] para reunir todas las innovaciones en tecnología de ensamblaje de genomas bajo el marco de código abierto .

La etiqueta de secuencia expresada o ensamblaje EST fue una estrategia temprana, que data de mediados de la década de 1990 a mediados de la década de 2000, para ensamblar genes individuales en lugar de genomas completos. [7] El problema difiere del ensamblaje del genoma en varias formas. Las secuencias de entrada para el ensamblaje EST son fragmentos del ARNm transcrito de una célula y representan solo un subconjunto de todo el genoma. [7] Una serie de problemas algorítmicos difieren entre el ensamblaje del genoma y EST. Por ejemplo, los genomas a menudo tienen grandes cantidades de secuencias repetitivas, concentradas en las regiones intergénicas. Los genes transcritos contienen muchas menos repeticiones, lo que hace que el ensamblaje sea algo más fácil. Por otro lado, algunos genes se expresan (transcriben) en cantidades muy altas (por ejemplo, genes de mantenimiento ), lo que significa que, a diferencia de la secuenciación shotgun del genoma completo, las lecturas no se muestrean uniformemente en todo el genoma.
El ensamblaje de EST se vuelve mucho más complicado por características como el empalme alternativo (cis) , el empalme trans , el polimorfismo de un solo nucleótido y la modificación postranscripcional . A partir de 2008, cuando se inventó el ARN-Seq , la secuenciación de EST fue reemplazada por esta tecnología mucho más eficiente, descrita en el ensamblaje del transcriptoma de novo .
En términos de complejidad y requisitos de tiempo, los ensamblajes de novo son órdenes de magnitud más lentos y consumen más memoria que los ensamblajes de mapeo. Esto se debe principalmente al hecho de que el algoritmo de ensamblaje necesita comparar cada lectura con cada una de las otras lecturas (una operación que tiene una complejidad temporal ingenua de O( n 2 )). Los ensambladores de genomas de novo actuales pueden utilizar diferentes tipos de algoritmos basados en grafos, como: [8]
En referencia a la comparación que se hace con los libros destrozados en la introducción: mientras que para los ensamblajes de mapas se tendría un libro muy similar como plantilla (quizás con los nombres de los personajes principales y algunas ubicaciones modificadas), los ensamblajes de novo presentan un desafío más abrumador, ya que no se sabría de antemano si se convertiría en un libro de ciencia, una novela, un catálogo o incluso varios libros. Además, cada fragmento se compararía con todos los demás fragmentos.
El manejo de repeticiones en un ensamblaje de novo requiere la construcción de un gráfico que represente las repeticiones vecinas. Esta información se puede obtener leyendo un fragmento largo que cubra las repeticiones en su totalidad o solo sus dos extremos . Por otro lado, en un ensamblaje de mapeo, las partes con múltiples coincidencias o sin coincidencias generalmente se dejan para que las examine otra técnica de ensamblaje. [3]
La complejidad del ensamblaje de secuencias depende de dos factores principales: la cantidad de fragmentos y sus longitudes. Si bien la cantidad de fragmentos y la longitud de los mismos permiten una mejor identificación de las superposiciones de secuencias, también plantean problemas, ya que los algoritmos subyacentes muestran un comportamiento de complejidad cuadrática o incluso exponencial tanto en relación con la cantidad de fragmentos como con su longitud. Y si bien las secuencias más cortas son más rápidas de alinear, también complican la fase de diseño de un ensamblaje, ya que las lecturas más cortas son más difíciles de usar con repeticiones o repeticiones casi idénticas.
En los primeros tiempos de la secuenciación del ADN, los científicos sólo podían obtener unas pocas secuencias de longitud corta (unas docenas de bases) tras semanas de trabajo en laboratorios. Por lo tanto, estas secuencias podían alinearse en unos minutos a mano.
En 1975 se inventó el método de terminación didesoxi (también conocido como secuenciación de Sanger ) y hasta poco después de 2000, la tecnología se mejoró hasta un punto en el que máquinas totalmente automatizadas podían generar secuencias en un modo altamente paralelizado las 24 horas del día. Grandes centros genómicos de todo el mundo albergaban granjas completas de estas máquinas de secuenciación, lo que a su vez llevó a la necesidad de que los ensambladores estuvieran optimizados para secuencias de proyectos de secuenciación shotgun de genoma completo donde las lecturas
Con la tecnología de Sanger, se podían reunir fácilmente en una sola computadora proyectos bacterianos con entre 20.000 y 200.000 lecturas. Los proyectos más grandes, como el genoma humano con aproximadamente 35 millones de lecturas, necesitaban grandes granjas de computación y computación distribuida.
En 2004/2005, la pirosecuenciación había alcanzado viabilidad comercial gracias a 454 Life Sciences . [9] Este nuevo método de secuenciación generó lecturas mucho más cortas que las de la secuenciación Sanger: inicialmente alrededor de 100 bases, ahora 400-500 bases. [9] Su rendimiento mucho mayor y su menor costo (en comparación con la secuenciación Sanger) impulsaron la adopción de esta tecnología por parte de los centros de genoma, lo que a su vez impulsó el desarrollo de ensambladores de secuencias que pudieran manejar eficientemente los conjuntos de lecturas. La gran cantidad de datos, junto con los patrones de error específicos de la tecnología en las lecturas, retrasaron el desarrollo de ensambladores; a principios de 2004, solo estaba disponible el ensamblador Newbler de 454. Lanzada a mediados de 2007, la versión híbrida del ensamblador MIRA de Chevreux et al. [10] fue el primer ensamblador disponible gratuitamente que podía ensamblar lecturas 454, así como mezclas de lecturas 454 y lecturas Sanger. El ensamblaje de secuencias de diferentes tecnologías de secuenciación se denominó posteriormente ensamblaje híbrido . [10]
Desde 2006, la tecnología Illumina (anteriormente Solexa) está disponible y puede generar alrededor de 100 millones de lecturas por ejecución en una sola máquina de secuenciación. Compárese esto con los 35 millones de lecturas del proyecto del genoma humano, que necesitaron varios años para producirse en cientos de máquinas de secuenciación. [11] Illumina inicialmente estaba limitada a una longitud de solo 36 bases, lo que la hacía menos adecuada para el ensamblaje de novo (como el ensamblaje de transcriptoma de novo ), pero las iteraciones más nuevas de la tecnología logran longitudes de lectura superiores a 100 bases desde ambos extremos de un clon de 3-400 pb. [11] Anunciado a fines de 2007, el ensamblador SHARCGS [12] por Dohm et al. fue el primer ensamblador publicado que se utilizó para un ensamblaje con lecturas Solexa. Fue seguido rápidamente por varios otros.
Más tarde, se lanzaron nuevas tecnologías como SOLiD de Applied Biosystems , Ion Torrent y SMRT y continúan surgiendo nuevas tecnologías (por ejemplo, secuenciación Nanopore ). A pesar de las mayores tasas de error de estas tecnologías, son importantes para el ensamblaje porque su mayor longitud de lectura ayuda a abordar el problema de la repetición. [11] Es imposible ensamblar a través de una repetición perfecta que sea más larga que la longitud de lectura máxima; sin embargo, a medida que las lecturas se hacen más largas, la posibilidad de una repetición perfecta tan grande se vuelve pequeña. Esto le da a las lecturas de secuenciación más largas una ventaja en el ensamblaje de repeticiones incluso si tienen baja precisión (~85%). [11]
La mayoría de los ensambladores de secuencias tienen incorporados algunos algoritmos para el control de calidad, como Phred . [13] Sin embargo, estas medidas no evalúan la integridad del ensamblaje en términos de contenido genético. Algunas herramientas evalúan la calidad de un ensamblaje después del hecho.
Por ejemplo, BUSCO (Benchmarking Universal Single-Copy Orthologs) es una medida de la integridad de los genes en un genoma, conjunto de genes o transcriptoma , que utiliza el hecho de que muchos genes están presentes solo como genes de copia única en la mayoría de los genomas. [14] Los conjuntos BUSCO iniciales representaban 3023 genes para vertebrados , 2675 para artrópodos , 843 para metazoos , 1438 para hongos y 429 para eucariotas . Esta tabla muestra un ejemplo para genomas humanos y de moscas de la fruta: [14]
Cada organismo tiene una región específica de mayor complejidad dentro de su genoma, por lo que se necesitan diferentes métodos computacionales. Algunos de los algoritmos más utilizados son:
Dado un conjunto de fragmentos de secuencia, el objetivo es encontrar una secuencia más larga que contenga todos los fragmentos (ver figura en Tipos de ensamblaje de secuencias ):
El resultado podría no ser una solución óptima al problema.
En general, hay tres pasos para ensamblar lecturas de secuenciación en un andamio:
Para obtener una lista de ensambladores de novo , consulte Ensambladores de secuencias de novo . Para obtener una lista de alineadores de mapeo, consulte Lista de software de alineación de secuencias § Alineación de secuencias de lectura corta .
Algunas de las herramientas comunes utilizadas en diferentes pasos de montaje se enumeran en la siguiente tabla: