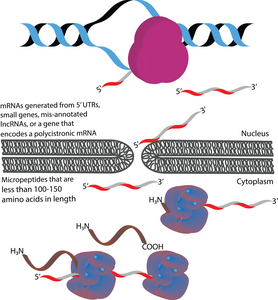
Los micropéptidos (también denominados microproteínas) son polipéptidos con una longitud de menos de 100-150 aminoácidos que están codificados por marcos de lectura abiertos cortos (sORF). [1] [2] [3] En este sentido, se diferencian de muchos otros pequeños polipéptidos activos, que se producen a través de la escisión postraduccional de polipéptidos más grandes. [1] [4] En términos de tamaño, los micropéptidos son considerablemente más cortos que las proteínas "canónicas", que tienen una longitud promedio de 330 y 449 aminoácidos en procariotas y eucariotas, respectivamente. [5] Los micropéptidos a veces se nombran de acuerdo con su ubicación genómica. Por ejemplo, el producto traducido de un marco de lectura abierto ascendente (uORF) podría llamarse péptido codificado por uORF (uPEP). [6] Los micropéptidos carecen de secuencias de señalización N-terminales, lo que sugiere que es probable que estén localizados en el citoplasma . [1] Sin embargo, algunos micropéptidos se han encontrado en otros compartimentos celulares, como lo indica la existencia de micropéptidos transmembrana . [7] [8] Se encuentran tanto en procariotas como en eucariotas. [1] [9] [10] Los sORF a partir de los cuales se traducen los micropéptidos pueden estar codificados en 5' UTR , genes pequeños o ARNm policistrónicos . Algunos genes codificadores de micropéptidos fueron originalmente anotados erróneamente como ARN largos no codificantes (lncRNA). [11]
Debido a su pequeño tamaño, los sORFs fueron pasados por alto originalmente. Sin embargo, cientos de miles de supuestos micropéptidos han sido identificados a través de varias técnicas en una multitud de organismos. Solo una pequeña fracción de estos con potencial codificante han tenido su expresión y función confirmada. Aquellos que han sido caracterizados funcionalmente, en general, tienen papeles en la señalización celular , organogénesis y fisiología celular . A medida que se descubren más micropéptidos, también se descubren más funciones de ellos. Una función reguladora es la de los peptoswitches, que inhiben la expresión de secuencias codificantes posteriores al detener los ribosomas , a través de su activación directa o indirecta por moléculas pequeñas. [11]
Existen varias técnicas experimentales para identificar posibles ORFs y sus productos de traducción. Estas técnicas sólo son útiles para la identificación de sORF que pueden producir micropéptidos y no para la caracterización funcional directa.
Un método para encontrar posibles sORFs, y por lo tanto micropéptidos, es a través de la secuenciación de ARN ( RNA-Seq ). RNA-Seq utiliza la secuenciación de próxima generación (NGS) para determinar qué ARN se expresan en una célula, tejido u organismo determinado en un momento específico. Esta colección de datos, conocida como transcriptoma , se puede utilizar como un recurso para encontrar posibles sORFs. [1] Debido a la alta probabilidad de que los sORFs de menos de 100 aa aparezcan por casualidad, es necesario realizar más estudios para determinar la validez de los datos obtenidos utilizando este método. [11]
El perfilado de ribosomas se ha utilizado para identificar posibles micropéptidos en un número cada vez mayor de organismos, incluidas moscas de la fruta, peces cebra, ratones y seres humanos. [11] Un método utiliza compuestos como harringtonina, puromicina o lactimidomicina para detener los ribosomas en los sitios de inicio de la traducción. [12] Esto indica dónde se está produciendo la traducción activa. Los inhibidores de la elongación de la traducción, como la emetina o la cicloheximida, también se pueden utilizar para obtener huellas de ribosomas que tienen más probabilidades de dar lugar a un ORF traducido. [13] Si un ribosoma está unido a un sORF o cerca de él, supuestamente codifica un micropéptido. [1] [2] [14]
La espectrometría de masas (MS) es el método de referencia para identificar y secuenciar proteínas. Mediante esta técnica, los investigadores pueden determinar si los polipéptidos se traducen de hecho a partir de un sORF.
La proteogenómica combina la proteómica, la genómica y la transcriptómica. Esto es importante cuando se buscan posibles micropéptidos. Un método para utilizar la proteogenómica implica el uso de datos de ARN-Seq para crear una base de datos personalizada de todos los polipéptidos posibles. Se realiza una cromatografía líquida seguida de espectrometría de masas en tándem (LC-MS/MS) para proporcionar información de secuencias para los productos de traducción. La comparación de los datos transcriptómicos y proteómicos se puede utilizar para confirmar la presencia de micropéptidos. [1] [2]
La conservación filogenética puede ser una herramienta útil, en particular cuando se examina una gran base de datos de sORF. La probabilidad de que un sORF resulte en un micropéptido funcional es mayor si se conserva en numerosas especies. [11] [12] Sin embargo, esto no funcionará para todos los sORF. Por ejemplo, aquellos que están codificados por lncRNA tienen menos probabilidades de conservarse dado que los propios lncRNA no tienen una alta conservación de secuencia. [2] Será necesario realizar más experimentos para determinar si de hecho se produce un micropéptido funcional.
Los anticuerpos personalizados dirigidos al micropéptido de interés pueden ser útiles para cuantificar la expresión o determinar la localización intracelular. Como sucede con la mayoría de las proteínas, una baja expresión puede dificultar la detección. El pequeño tamaño del micropéptido también puede generar dificultades a la hora de diseñar un epítopo desde el cual dirigir el anticuerpo. [2]
La edición genómica se puede utilizar para añadir FLAG/MYC u otras pequeñas etiquetas de péptidos a un sORF endógeno, creando así proteínas de fusión. En la mayoría de los casos, este método es beneficioso porque se puede realizar más rápidamente que el desarrollo de un anticuerpo personalizado. También es útil para micropéptidos para los que no se puede apuntar a ningún epítopo. [2]
Este proceso implica la clonación del ADNc del micropéptido de longitud completa en un plásmido que contiene un promotor T7 o SP6. Este método utiliza un sistema de síntesis de proteínas sin células en presencia de 35 S-metionina para producir el péptido de interés. Los productos pueden luego analizarse mediante electroforesis en gel y el péptido marcado con 35 S se visualiza mediante autorradiografía. [2]
Existen varios repositorios y bases de datos que se han creado tanto para sORF como para micropéptidos. En sORFs.org se puede encontrar un repositorio de ORF pequeños descubiertos mediante el análisis de perfiles de ribosomas. [15] [16] En ARA-PEPs se puede encontrar un repositorio de péptidos codificados por sORF putativos en Arabidopsis thaliana . [17] [18] En SmProt se puede encontrar una base de datos de proteínas pequeñas, especialmente codificadas por ARN no codificantes. [19] [20]
Hasta la fecha, la mayoría de los micropéptidos han sido identificados en organismos procariotas . Si bien la mayoría aún no se han caracterizado por completo, de los que se han estudiado, muchos parecen ser fundamentales para la supervivencia de estos organismos. Debido a su pequeño tamaño, los procariotas son particularmente susceptibles a los cambios en su entorno y, por lo tanto, han desarrollado métodos para asegurar su existencia.
Los micropéptidos expresados en E. coli ejemplifican las adaptaciones ambientales bacterianas. La mayoría de ellos se han clasificado en tres grupos: péptidos líderes, proteínas ribosómicas y proteínas tóxicas. Las proteínas líderes regulan la transcripción y/o traducción de proteínas implicadas en el metabolismo de aminoácidos cuando estos son escasos. Las proteínas ribosómicas incluyen L36 ( rpmJ ) y L34 ( rpmH ), dos componentes de la subunidad ribosómica 50S. Las proteínas tóxicas, como ldrD , son tóxicas en niveles altos y pueden matar células o inhibir el crecimiento, lo que funciona para reducir la viabilidad de la célula huésped. [21]
En S. enterica, el factor de virulencia MgtC está involucrado en la adaptación a ambientes con bajo contenido de magnesio. El péptido hidrofóbico MgrR se une a MgtC, provocando su degradación por la proteasa FtsH. [9]
El micropéptido Sda de 46 aa, expresado por B. subtilis , reprime la esporulación cuando se altera el inicio de la replicación. Al inhibir la histidina quinasa KinA, Sda previene la activación del factor de transcripción Spo0A, que es necesario para la esporulación. [10]
En S. aureus , hay un grupo de micropéptidos, de 20 a 22 aa, que se excretan durante la infección del huésped para alterar las membranas de los neutrófilos, lo que provoca la lisis celular. Estos micropéptidos permiten que la bacteria evite la degradación por parte de las principales defensas del sistema inmunológico humano. [22] [23]
Se han descubierto micropéptidos en organismos eucariotas , desde Arabidopsis thaliana hasta los humanos. Desempeñan diversas funciones en el desarrollo de tejidos y órganos, así como en su mantenimiento y funcionamiento una vez que están completamente desarrollados. Si bien muchos aún no se han caracterizado funcionalmente y es probable que queden más por descubrir, a continuación se presenta un resumen de las funciones de los micropéptidos eucariotas identificadas recientemente.
El gen POLARIS (PLS) codifica un micropéptido de 36 aa. Es necesario para la correcta formación de patrones vasculares en las hojas y la expansión celular en la raíz. Este micropéptido interactúa con las proteínas PIN del desarrollo para formar una red crítica para la comunicación hormonal entre la auxina, el etileno y la citoquinina. [24] [25] [26]
ROTUNDIFOLIA (ROT4 ) en A. thaliana codifica un péptido de 53 aa, que se localiza en la membrana plasmática de las células de las hojas. El mecanismo de la función de ROT4 no se entiende bien, pero los mutantes tienen hojas cortas y redondeadas, lo que indica que este péptido puede ser importante en la morfogénesis de las hojas. [27]
Brick1 (Brk1) codifica un micropéptido de 76 aa, que está altamente conservado tanto en plantas como en animales. En Z. mays, se descubrió que participa en la morfogénesis de los epitelios de las hojas, al promover múltiples eventos de polarización celular dependientes de actina en la epidermis de las hojas en desarrollo. [28] Zm401p10 es un micropéptido de 89 aa, que desempeña un papel en el desarrollo normal del polen en el tapete. Después de la mitosis, también es esencial en la degradación del tapete. [29] Zm908p11 es un micropéptido de 97 aa de longitud, codificado por el gen Zm908 que se expresa en los granos de polen maduros. Se localiza en el citoplasma de los tubos polínicos, donde ayuda a su crecimiento y desarrollo. [30]
El gen del arroz pulido ( pri ), conservado evolutivamente, conocido como tarsal-less (tal) en D. melanogaster , está involucrado en la diferenciación epidérmica. Esta transcripción policistrónica codifica cuatro péptidos similares, que varían entre 11 y 32 aa de longitud. Su función es truncar el factor de transcripción Shavenbaby (Svb). Esto convierte a Svb en un activador que regula directamente la expresión de los efectores objetivo, incluidos el miniatura (m) y el shavenoid (sha) , que juntos son responsables de la formación de tricomas. [31]
El gen Elabela ( Ela) (también conocido como Apela, Toddler) es importante para la embriogénesis. [32] Se expresa específicamente durante las últimas etapas de blástula y gástrula. Durante la gastrulación , es fundamental para promover la internalización y el movimiento dirigido al polo animal de las células endodérmicas del mes . Después de la gastrulación, Ela se expresa en el mesodermo lateral, el endodermo, así como en la notocorda anterior y posterior. Aunque se anotó como un lncRNA en pez cebra, ratón y humano, se encontró que el ORF de 58 aa estaba altamente conservado entre las especies de vertebrados. Ela se procesa mediante la eliminación de su péptido señal N-terminal y luego se secreta en el espacio extracelular . Su péptido maduro de 34 aa sirve como el primer ligando endógeno de un GPCR conocido como el receptor de apelina . [33] [32] La inactivación genética de Ela o Aplnr en pez cebra da como resultado fenotipos sin corazón. [34] [35]
La miorregulina (Mln) está codificada por un gen originalmente anotado como lncRNA. Mln se expresa en los tres tipos de músculo esquelético y funciona de manera similar a los micropéptidos fosfolamban (Pln) en el músculo cardíaco y sarcolipin (Sln) en el músculo esquelético lento (tipo I). Estos micropéptidos interactúan con la Ca2 + -ATPasa del retículo sarcoplásmico (SERCA), una bomba de membrana responsable de regular la captación de Ca2 + en el retículo sarcoplásmico (SR). Al inhibir la captación de Ca2 + en el SR, provocan la relajación muscular. De manera similar, los genes de la endorregulina (ELN) y otra-regulina (ALN) codifican micropéptidos transmembrana que contienen el motivo de unión de SERCA y se conservan en los mamíferos. [7]
Myomixer (Mymx) está codificado por el gen Gm7325, un péptido específico del músculo, de 84 aa de longitud, que desempeña un papel durante la embriogénesis en la fusión y la formación del músculo esquelético. Se localiza en la membrana plasmática, asociándose con una proteína de membrana fusogénica, Myomaker (Mymk). En los seres humanos, el gen que codifica Mymx está anotado como LOC101929726 no caracterizado . También se encuentran ortólogos en los genomas de tortugas, ranas y peces. [8]
En los seres humanos, NoBody (polipéptido disociado de cuerpo P no anotado), un micropéptido de 68 aa, se descubrió en el ARN no codificante de intervención larga (lincRNA) LINC01420 . Tiene una alta conservación de secuencia entre los mamíferos y se localiza en los cuerpos P. Enriquece las proteínas asociadas con el descapuchado del ARNm 5' . Se cree que interactúa directamente con Enhancer of mRNA Decapping 4 (EDC4). [36]
ELABELA ( ELA) (también conocida como APELA) es una hormona endógena que se secreta como un micropéptido de 32 aminoácidos por las células madre embrionarias humanas . [32] Es esencial para mantener la autorrenovación y pluripotencia de las células madre embrionarias humanas . Sus señales de manera autocrina a través de la vía PI3/AKT mediante un receptor de superficie celular aún no identificado. [37] En las células mesoendérmicas diferenciadoras, ELA se une a, y envía señales a través de, APLNR , un GPCR que también puede responder al péptido hormonal APLN .
Se predice que el gen CYREN , conservado en mamíferos, cuando se empalma de forma alternativa produce tres micropéptidos. Anteriormente se había descubierto que el MRI-1 era un modulador de la infección por retrovirus. El segundo micropéptido predicho, el MRI-2, puede ser importante en la unión de extremos no homólogos (NHEJ) de las roturas de doble cadena de ADN. En experimentos de co-inmunoprecipitación, el MRI-2 se unió a Ku70 y Ku80 , dos subunidades de Ku , que desempeñan un papel importante en la vía NHEJ. [38]
El micropéptido de 24 aminoácidos, Humanina (HN), interactúa con la proteína inductora de apoptosis Bcl2-associated X protein (Bax). En su estado activo, Bax sufre un cambio conformacional que expone los dominios de orientación a la membrana. Esto hace que se mueva desde el citosol a la membrana mitocondrial, donde inserta y libera proteínas apoptógenas como el citocromo c. Al interactuar con Bax, HN evita que Bax se dirija a las mitocondrias, bloqueando así la apoptosis. [39]
Se encontró que un micropéptido de 90 aa, el 'Pequeño Polipéptido Regulador de la Respuesta a los Aminoácidos' o SPAAR, estaba codificado en el lncRNA LINC00961 . Se conserva entre humanos y ratones, y se localiza en el endosoma tardío/lisosoma. SPAAR interactúa con cuatro subunidades del complejo v-ATPasa , inhibiendo la translocación de mTORC1 a la superficie lisosomal donde se activa. La regulación negativa de este micropéptido permite la activación de mTORC1 por estimulación de aminoácidos, promoviendo la regeneración muscular. [40]
![]() Este artículo fue adaptado de la siguiente fuente bajo licencia CC BY 4.0 (2018) (informes de los revisores): Maria E. Sousa; Michael H. Farkas (13 de diciembre de 2018). "Micropeptide". PLOS Genetics . 14 (12): e1007764. doi : 10.1371/JOURNAL.PGEN.1007764 . ISSN 1553-7390. PMC 6292567 . PMID 30543625. Wikidata Q60017699.
Este artículo fue adaptado de la siguiente fuente bajo licencia CC BY 4.0 (2018) (informes de los revisores): Maria E. Sousa; Michael H. Farkas (13 de diciembre de 2018). "Micropeptide". PLOS Genetics . 14 (12): e1007764. doi : 10.1371/JOURNAL.PGEN.1007764 . ISSN 1553-7390. PMC 6292567 . PMID 30543625. Wikidata Q60017699.