L1 , también conocida como L1CAM , es una proteína transmembrana miembro de la familia de proteínas L1 , codificada por el gen L1CAM . Esta proteína, de 200 a 220 kDa, es una molécula de adhesión celular neuronal con una fuerte implicación en la migración celular, adhesión, crecimiento de neuritas, mielinización y diferenciación neuronal. [5] También juega un papel clave en cánceres resistentes al tratamiento debido a su función. Fue identificada por primera vez en 1984 por M. Schachner, quien encontró la proteína en neuronas de ratones postmitóticos .
Las mutaciones en la proteína L1 son la causa del síndrome L1 , a veces conocido por el acrónimo CRASH (hipoplasia del cuerpo calloso, retraso mental, afasia, paraplejía espástica e hidrocefalia). [6]
La proteína L1 se encuentra en todo el sistema nervioso, en la superficie de las neuronas. Se ubica a lo largo de la membrana celular, de modo que un extremo de la proteína permanece dentro de la célula nerviosa, mientras que el otro extremo permanece en la superficie externa de la neurona. Esta posición permite que la proteína active señales químicas que se propagan a través de la neurona. [7]
Existe una gran variedad de células que expresan la proteína L1, no sólo las neuronales sino también algunas no neuronales. [8] Las células que hoy en día se sabe que expresan la proteína L1 son: oligodendrocitos inmaduros y células de Schwann , que son células no neuronales que dan soporte y protección a las neuronas y forman la mielina; células T que son linfocitos involucrados en la inmunidad mediada por células; otros tipos de linfocitos como las células B y los monocitos . También se expresa en células progenitoras epiteliales intestinales, neuronas del cerebelo como las células granulares del cerebelo y las células de Purkinje . Finalmente, se expresa en múltiples células tumorales por ejemplo células de melanoma y carcinoma pulmonar . [5]
L1CAM también se utiliza con frecuencia como marcador de vesículas extracelulares (VE) que se originan en células neuronales, aunque su presencia específicamente en las VE derivadas de neuronas es discutible. [9]
El gen L1CAM humano se encuentra en regiones del cromosoma X implicadas en diferentes enfermedades neuromusculares y cerca de la asociada con el retraso mental. El gen L1CAM se encuentra en el brazo largo del cromosoma X en la posición Xq28. [10] [11]


La molécula de adhesión celular L1 (L1CAM) es una glicoproteína de la superficie celular que se encuentra en los humanos (y en otras formas de vida como los ratones, por ejemplo) y que tiene una secuencia proteica de 1253 aminoácidos. La porción extracelular está formada por seis dominios de inmunoglobulina seguidos de cinco dominios de fibronectina tipo III que están conectados a un pequeño dominio intracelular por una hélice transmembrana. La proteína humana es muy similar a la que se encuentra en los ratones (son 92% idénticos a nivel de aminoácidos ), lo que permite a los científicos estudiar su estructura. Hay otras proteínas CAM como la Ng-CAM (que se encuentra en el pollo) que tiene menos similitudes con la humana (son 40% idénticos a nivel de aminoácidos). La comparación de las secuencias de humano, ratón, pollo y Drosophila y su buena conservación, indica que el dominio 2 de inmunoglobulina L1 y el dominio 2 de fibronectina tipo III probablemente sean funcionalmente importantes. [12] [13]
L1 es una proteína importante para el desarrollo del sistema nervioso afectando tanto la adhesión como la motilidad celular.
L1 tiene una función estática como molécula de adhesión celular que conecta diferentes células. Interviene en la adhesión entre neuronas y en el crecimiento y asociación de neuritas, denominada fasciculación neurítica. [14]
Las funciones que promueven la motilidad están relacionadas con la regulación del movimiento de las células nerviosas durante el desarrollo neuronal . L1 está presente en las neuronas en desarrollo y desempeña un papel importante al guiar a las nuevas neuronas hacia las posiciones correctas y ayudar a los axones a crecer y establecer conexiones con otras neuronas. L1 también está involucrada en la plasticidad sináptica , que es la capacidad de las sinapsis de fortalecerse o debilitarse, y también desempeña un papel en la regeneración después de un trauma.
Algunos estudios han demostrado que L1 tiene un papel en el crecimiento tumoral, invasión de células tumorales, metástasis de melanoma, cáncer de ovario y de colon [15] debido a una sobreexpresión de la proteína L1 que mejora el movimiento celular de las células malignas.
Los dominios de esta proteína promueven interacciones homofílicas, donde las moléculas de adhesión de una célula interactúan con moléculas idénticas en la otra célula. Y también interacciones heterófilas, donde una molécula de adhesión de una célula funciona como un receptor que se conecta con una molécula diferente en la otra célula. [16] [17] Estas interacciones promueven la adhesión celular y la regulación de la transducción de señales .
Además, L1 participa en los procesos de mielinización, que están implicados en la proliferación de la mielina a través del sistema nervioso (específicamente la mielinización progresiva de las fibras axónicas nerviosas), al mediar la elongación de las células de Schwann a lo largo del axón.
L1 está involucrado en la adhesión neurona-neurona, fasciculación de neuritas, crecimiento de neuritas, migración de células granulares cerebelosas, crecimiento de neuritas en células de Schwann e interacciones entre células epiteliales de criptas intestinales. [18] Como consecuencia, las mutaciones en el gen L1CAM causan un mal funcionamiento del sistema nervioso . Los principales trastornos vinculados a esta mutación se conocen por el acrónimo CRASH o también pueden denominarse síndrome L1 . Esto incluye trastornos como HSAS , síndrome MASA , agenesia del cuerpo calloso y paraplejía espástica . La espasticidad de las extremidades inferiores, el retraso mental , la hidrocefalia y la deformidad en flexión de los pulgares son algunos de los síntomas expresados principalmente en individuos varones que padecen esta condición. [19] [20] [21] Aunque los mecanismos patológicos que conducen al síndrome L1 aún se desconocen, se han identificado alrededor de 200 mutaciones del gen L1CAM y luego se han asociado con el síndrome. Estas mutaciones afectan principalmente a residuos clave estructuralmente importantes en la región extracelular de L1, causando alteraciones en las propiedades de unión de proteínas, que se correlacionan con el deterioro de los mecanismos fisiológicos neuronales como la adhesión celular o la interacción específica con otras moléculas. [22] La interacción de anquirina con L1CAM es un ejemplo de una unión de proteínas que falla en pacientes con CRASH [23] debido a una mutación que hace que la leucina y la histidina reemplacen a la serina y la tirosina respectivamente, en el motivo SFIGQY, donde la anquirina debería estar unida en el extremo citoplasmático de la familia L1CAM. [24] [25] La interacción anquirina-L1CAM está involucrada en la iniciación del cono de crecimiento , en consecuencia, una falla en esta interacción hace que las neuritas no alcancen el objetivo sináptico.
Además, la evidencia muestra que existe una correlación entre el trastorno del espectro alcohólico fetal y la proteína L1, ya que el etanol inhibe la adhesión mediada por L1 y el crecimiento de neuritas. [26] La enfermedad de Hirschsprung también se ha relacionado con un mal funcionamiento de L1CAM. [27]
El gen que regula la transcripción de L1CAM se encuentra en el cromosoma X. El gen L1CAM tiene una longitud de 24.657 pb y está formado por 28 exones. El empalme alternativo de este gen conduce a múltiples variantes de transcripción (hay 7 transcripciones diferentes del gen), [28] incluyendo algunas que tienen un exón alternativo que se considera específico de las neuronas . [29] Se sabe que la transcripción de L1 tiene lugar en el cerebro fetal humano y en las líneas celulares de neuroblastoma y retinoblastoma . L1 también se expresa en las líneas celulares de rabdomiosarcoma RD y A-204. Se pueden encontrar dos formas de L1 en humanos, con la diferencia de que una tiene un segmento citoplasmático de 12 pb y la otra carece de él. [30] La regulación de la expresión de L1CAM en la transcripción no se comprende completamente. Se verificaron dos sitios en líneas celulares de carcinoma endometrial y parecen usarse de manera específica dependiendo del tipo de célula. Hay dos sitios de inicio de la transcripción, ubicados en dos exones diferentes (frente a un exón 0 no traducido y al lado del primer exón 1 codificador de proteína). [31] SLUG ( SNAI2 ), un factor de transcripción, regula positivamente la expresión de L1CAM. [32]
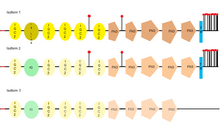
L1CAM tiene tres isoformas diferentes , que difieren en su secuencia de aminoácidos , debido al splicing alternativo (un proceso que permite obtener diferentes moléculas maduras de ARNm a partir de una transcripción primaria de ARNm ). La isoforma 1 de L1CAM se conoce como secuencia canónica . [33] La principal diferencia entre ellas es dónde se pueden encontrar, por ejemplo, la isoforma de longitud completa (isoforma 1), es la que se encuentra habitualmente en las células neuronales, mientras que la isoforma corta o no neuronal (isoforma 2), es predominante en los otros tipos de células. [34]
Se ha demostrado que L1 (proteína) interactúa con NUMB . [36]
La L1CAM es capaz de plegarse en una configuración de herradura mediante el establecimiento de interacciones homofílicas dentro de dominios similares a Ig de la misma proteína (el primer y el segundo motivo de Ig se pliegan sobre el cuarto y tercer motivo). Esta conformación es esencial para que la L1CAM pueda interactuar con otras moléculas y, posteriormente, realizar algunas de sus funciones más importantes.
Los dominios similares a Ig están implicados en muchas interacciones homofílicas con otras proteínas L1CAM ubicadas en células adyacentes. Las moléculas L1CAM interactúan a través de los dominios similares a Ig (1-4), lo que permite la adhesión entre células. También son importantes en la formación de interacciones heterofílicas con NCAM , TAG-1 , F11 y las tirosina quinasas receptoras (especialmente durante el desarrollo del sistema nervioso).
El motivo de seis Ig de la proteína L1 contiene una secuencia Arg-Gly-Asp que permite la unión con diversas integrinas de la superficie celular . Esta interacción conduce a una cascada de señalización que activa las quinasas de adhesión focal (FAK) que luego se convierten a su estado activo y forman el complejo FAK/ SRC . Este último funciona como un activador de las quinasas de proteína activadas por mitógenos . Otra función derivada de la unión de la integrina es la activación de NF-κB que da como resultado que las células sean más móviles e invasivas. [5]
Los dominios de fibronectina de la proteína L1 también son capaces de unirse a las integrinas de la superficie celular. Interactúan con el receptor 1 del factor de crecimiento de fibroblastos , lo que sugiere que puede estar vinculado a la modulación de la diferenciación neuronal. [5]
Los socios de unión más importantes de la cola citoplasmática de las proteínas L1 son las anquirinas . La interacción se lleva a cabo en sitios de unión de alta afinidad ubicados dentro de las llamadas "repeticiones ank", también conocidas como dominios de unión a la membrana. [5] Esta interacción permite que la proteína L1 se conecte con el citoesqueleto de la célula. Además, la cola citoplasmática de la proteína L1 puede unirse al adaptador 2 (ADP) , un componente clave de la endocitosis mediada por clatrina .
El hecho de que esta región contenga algunos sitios de fosforilación sugiere que L1 puede estar sujeta a regulación por quinasas. [5]
La expresión de la proteína L1CAM normalmente está restringida a las neuronas. Sin embargo, se ha observado que hay una sobreexpresión de L1CAM en todos los tipos de células cancerosas, lo que se ha asociado con un mal pronóstico, progresión tumoral y metástasis . [37] Esta regulación positiva puede no estar necesariamente asociada con mutaciones en los factores de transcripción L1. Se ha visto que esta proteína juega un papel clave en las reacciones inflamatorias como las que tienen lugar en el tejido que rodea un tumor. Esto podría explicar por qué esta proteína se produce en exceso repentinamente en las células tumorales. Las diversas funciones de L1CAM hacen que las células tumorales sean más agresivas y resistentes. Sus funciones migratorias y relacionadas con la motilidad pueden resultar clave en la transición epitelial-mesenquimal (EMT) celular, lo que permite que las células pierdan las uniones estáticas de célula a célula y la polaridad apico-basal, lo que las lleva a volverse migratorias e independientes. Además, su capacidad para formar interacciones adhesivas dentro de diferentes tipos de células puede resultar en una ventaja para las células tumorales cuando se trata de cooptar e invadir los tejidos o capilares circundantes.
Una vez que las células tumorales se vuelven independientes del anclaje y migratorias, debido a la regulación positiva de L1, abandonan el tejido al que pertenecen y migran a través de los capilares hacia otros órganos. Un destino frecuente de las células tumorales es el cerebro. Por lo tanto, para establecerse en el cerebro, las células tumorales tienen que lograr cruzar la barrera hematoencefálica (BHE) , donde quedan expuestas a la plasmina secretada por los astrocitos . La plasmina rompe la L1CAM e inhibe los poderes migratorios de la célula maligna. Sin embargo, estudios recientes han observado que estas células cancerosas producen en exceso serpinas anti-PA , que son los inhibidores habituales de la plasmina, lo que les permite cruzar la BHE y lograr la metástasis. [37]

Debido a que L1CAM se considera un factor clave en la metástasis , se ha sugerido que el bloqueo de esta proteína puede inhibir la migración de células cancerosas y la progresión tumoral. La terapia con anticuerpos dirigida contra L1CAM en modelos de cáncer de ratones bloquea el crecimiento tumoral pero mejora la EMT . [38] El ARN interferente pequeño encapsulado en liposomas también ha demostrado ser un inhibidor eficaz para la expresión de L1CAM, ya que su función es degradar un rango específico de pares de bases de ARNm (en este caso, los que codifican la secuencia de aminoácidos de L1CAM ) después de la transcripción, de modo que la proteína no pueda sintetizarse. [ cita requerida ] Sin embargo, estas posibles terapias que involucran a L1CAM como objetivo en el cáncer humano aún se encuentran en investigación preclínica. [39]
Este artículo incorpora texto de la Biblioteca Nacional de Medicina de los Estados Unidos , que se encuentra en el dominio público .
Atlas de genética y citogenética en oncología y hematología: http://atlasgeneticsoncology.org/Genes/L1CAMID44110chXq28.html