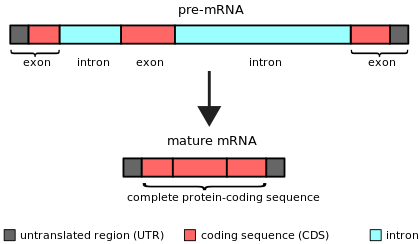
Un intrón es cualquier secuencia de nucleótidos dentro de un gen que no se expresa ni está operativa en el producto final de ARN. La palabra intrón se deriva del término región intragénica , es decir, una región dentro de un gen. [1] El término intrón se refiere tanto a la secuencia de ADN dentro de un gen como a la secuencia de ARN correspondiente en las transcripciones de ARN . [2] Las secuencias que no son intrones que se unen mediante este procesamiento del ARN para formar el ARN maduro se denominan exones . [3]
Los intrones se encuentran en los genes de la mayoría de los eucariotas y de muchos virus eucariotas y pueden estar ubicados tanto en genes codificadores de proteínas como en genes que funcionan como ARN ( genes no codificantes ). Hay cuatro tipos principales de intrones: intrones de ARNt, intrones del grupo I, intrones del grupo II e intrones espliceosómicos (ver a continuación). Los intrones son raros en bacterias y arqueas (procariotas).
Los intrones se descubrieron por primera vez en los genes codificadores de proteínas de los adenovirus [4] [5] y, posteriormente, se identificaron en los genes que codifican el ARN de transferencia y los genes del ARN ribosómico. Ahora se sabe que los intrones se encuentran en una amplia variedad de genes en organismos, bacterias [6] y virus en todos los reinos biológicos.
El hecho de que los genes se dividieran o interrumpieran por intrones fue descubierto independientemente en 1977 por Phillip Allen Sharp y Richard J. Roberts , por lo que compartieron el Premio Nobel de Fisiología o Medicina en 1993, [7] aunque se excluyó el crédito para los investigadores y colaboradores en sus laboratorios que hicieron los experimentos que resultaron en el descubrimiento, Susan Berget y Louise Chow . [8] [9] El término intrón fue introducido por el bioquímico estadounidense Walter Gilbert : [1]
"La noción de cistrón [es decir, gen]... debe ser reemplazada por la de una unidad de transcripción que contiene regiones que se perderán del mensajero maduro –y que propongo que llamemos intrones (por regiones intragénicas)– alternando con regiones que se expresarán –exones-." (Gilbert 1978)
El término intrón también se refiere a un intracistrón , es decir, un fragmento adicional de ADN que surge dentro de un cistrón . [10]
Aunque a veces a los intrones se los denomina secuencias intermedias , [11] el término "secuencia intermedia" puede referirse a cualquiera de varias familias de secuencias de ácidos nucleicos internos que no están presentes en el producto génico final, incluidas las inteínas , las regiones no traducidas (UTR) y los nucleótidos eliminados mediante la edición de ARN , además de los intrones.
Se observa que la frecuencia de intrones dentro de diferentes genomas varía ampliamente en todo el espectro de organismos biológicos. Por ejemplo, los intrones son extremadamente comunes dentro del genoma nuclear de vertebrados con mandíbulas (por ejemplo, humanos, ratones y peces globo (fugu)), donde los genes codificadores de proteínas casi siempre contienen múltiples intrones, mientras que los intrones son raros dentro de los genes nucleares de algunos microorganismos eucariotas, [12] por ejemplo, la levadura de panadería/cervecería ( Saccharomyces cerevisiae ). Por el contrario, los genomas mitocondriales de vertebrados están completamente desprovistos de intrones, mientras que los de microorganismos eucariotas pueden contener muchos intrones. [13]
Un caso particularmente extremo es el gen dhc7 de Drosophila que contiene un intrón de ≥3,6 megabases (Mb), que tarda aproximadamente tres días en transcribirse. [14] [15] En el otro extremo, un estudio de 2015 sugiere que la longitud de intrón de metazoo más corta conocida es de 30 pares de bases (pb) pertenecientes al gen humano MST1L . [16] Los intrones más cortos conocidos pertenecen a los ciliados heterotricos , como Stentor coeruleus , en el que la mayoría (> 95%) de los intrones tienen una longitud de 15 o 16 pb. [17]
El empalme de todas las moléculas de ARN que contienen intrones es superficialmente similar, como se describió anteriormente. Sin embargo, se identificaron diferentes tipos de intrones mediante el examen de la estructura de los intrones mediante análisis de secuencias de ADN, junto con el análisis genético y bioquímico de las reacciones de empalme del ARN. Se han identificado al menos cuatro clases distintas de intrones:
Se propone que los intrones del grupo III sean una quinta familia, pero se sabe poco sobre el aparato bioquímico que media su empalme. Parecen estar relacionados con los intrones del grupo II y posiblemente con los intrones espliceosomales. [18]
Los intrones nucleares de pre-ARNm (intrones spliceosomales) se caracterizan por secuencias intrónicas específicas ubicadas en los límites entre intrones y exones. [19] Estas secuencias son reconocidas por las moléculas de ARN spliceosomal cuando se inician las reacciones de empalme. [20] Además, contienen un punto de ramificación, una secuencia de nucleótidos particular cerca del extremo 3' del intrón que se une covalentemente al extremo 5' del intrón durante el proceso de empalme, generando un intrón ramificado ( lazo ) [ aclaración necesaria (jerga complicada) ] . Aparte de estos tres elementos conservados cortos, las secuencias intrónicas nucleares de pre-ARNm son muy variables. Los intrones nucleares de pre-ARNm suelen ser mucho más largos que sus exones circundantes.
Los intrones del ARN de transferencia que dependen de proteínas para su eliminación se encuentran en una ubicación específica dentro del bucle anticodón de los precursores de ARNt no empalmados y son eliminados por una endonucleasa de empalme de ARNt. Luego, los exones se unen entre sí mediante una segunda proteína, la ligasa de empalme de ARNt. [21] Cabe señalar que los intrones autoempalmados también se encuentran a veces dentro de los genes de ARNt. [22]
Los intrones del grupo I y del grupo II se encuentran en genes que codifican proteínas ( ARN mensajero ), ARN de transferencia y ARN ribosómico en una amplia gama de organismos vivos. [23] [24] Después de la transcripción en ARN, los intrones del grupo I y del grupo II también realizan amplias interacciones internas que les permiten plegarse en una arquitectura tridimensional específica y compleja . Estas arquitecturas complejas permiten que algunos intrones del grupo I y del grupo II se autoempalmen , es decir, la molécula de ARN que contiene intrones puede reorganizar su propia estructura covalente para eliminar con precisión el intrón y unir los exones en el orden correcto. En algunos casos, determinadas proteínas de unión a intrones participan en el empalme, actuando de tal manera que ayudan al intrón a plegarse en la estructura tridimensional que es necesaria para la actividad de autoempalme. Los intrones del grupo I y del grupo II se distinguen por diferentes conjuntos de secuencias internas conservadas y estructuras plegadas, y por el hecho de que el empalme de moléculas de ARN que contienen intrones del grupo II genera intrones ramificados (como los de los ARN espliceosomales), mientras que los intrones del grupo I utilizan un nucleótido de guanosina no codificado (normalmente GTP) para iniciar el empalme, añadiéndolo al extremo 5' del intrón escindido.
El espliceosoma es una estructura muy compleja que contiene hasta cien proteínas y cinco ARN diferentes. El sustrato de la reacción es una molécula larga de ARN y las reacciones de transesterificación catalizadas por el espliceosoma requieren la unión de sitios que pueden estar separados por miles de nucleótidos. [25] [26] Todas las reacciones bioquímicas están asociadas con tasas de error conocidas y cuanto más complicada sea la reacción, mayor será la tasa de error. Por lo tanto, no es sorprendente que la reacción de empalme catalizada por el espliceosoma tenga una tasa de error significativa a pesar de que existen factores accesorios del espliceosoma que suprimen la escisión accidental de sitios de empalme crípticos. [27]
En circunstancias ideales, es probable que la reacción de empalme tenga una precisión del 99,999 % (tasa de error de 10 −5 ) y se unirán los exones correctos y se eliminará el intrón correcto. [28] Sin embargo, estas condiciones ideales requieren coincidencias muy cercanas con las mejores secuencias del sitio de empalme y la ausencia de cualquier secuencia críptica del sitio de empalme competitiva dentro de los intrones y esas condiciones rara vez se cumplen en genes eucariotas grandes que pueden cubrir más de 40 pares de kilobases. Estudios recientes han demostrado que la tasa de error real puede ser considerablemente mayor que 10 −5 y puede ser tan alta como 2 % o 3 % de errores (tasa de error de 2 o 3 x 10 −2 ) por gen. [29] [30] [31] Estudios adicionales sugieren que la tasa de error no es inferior al 0,1 % por intrón. [32] [33] Este nivel relativamente alto de errores de empalme explica por qué la mayoría de las variantes de empalme se degradan rápidamente por la descomposición mediada por sinsentidos. [34] [35]
La presencia de sitios de unión descuidados dentro de los genes causa errores de empalme y puede parecer extraño que estos sitios no hayan sido eliminados por la selección natural. El argumento a favor de su persistencia es similar al argumento a favor del ADN basura. [32] [36]
Aunque las mutaciones que crean o alteran sitios de unión pueden ser ligeramente perjudiciales, la gran cantidad de posibles mutaciones de este tipo hace inevitable que algunas lleguen a fijarse en una población. Esto es particularmente relevante en especies, como los humanos, con tamaños de población efectivos a largo plazo relativamente pequeños. Es plausible, entonces, que el genoma humano contenga una carga sustancial de secuencias subóptimas que causan la generación de isoformas de transcripción aberrantes. En este estudio, presentamos evidencia directa de que esto es efectivamente así. [32]
Si bien la reacción catalítica puede ser lo suficientemente precisa para un procesamiento efectivo la mayor parte del tiempo, la tasa de error general puede estar limitada en parte por la fidelidad de la transcripción, ya que los errores de transcripción introducirán mutaciones que crearán sitios de empalme crípticos. Además, la tasa de error de transcripción de 10 −5 – 10 −6 es lo suficientemente alta como para que uno de cada 25 000 exones transcritos tenga un error de incorporación en uno de los sitios de empalme, lo que dará lugar a la omisión de un intrón o un exón. Casi todos los genes multiexónicos producirán transcripciones empalmadas incorrectamente, pero la frecuencia de este ruido de fondo dependerá del tamaño de los genes, la cantidad de intrones y la calidad de las secuencias del sitio de empalme. [30] [33]
En algunos casos, las variantes de empalme se producirán por mutaciones en el gen (ADN). Estos pueden ser polimorfismos de SNP que crean un sitio de empalme críptico o mutan un sitio funcional. También pueden ser mutaciones de células somáticas que afectan el empalme en un tejido particular o una línea celular. [37] [38] [39] Cuando el alelo mutante está en un estado heterocigoto, esto dará como resultado la producción de dos variantes de empalme abundantes; una funcional y otra no funcional. En el estado homocigoto, los alelos mutantes pueden causar una enfermedad genética como la hemofilia encontrada en los descendientes de la Reina Victoria, donde una mutación en uno de los intrones en un gen del factor de coagulación de la sangre crea un sitio de empalme 3' críptico que resulta en un empalme aberrante. [40] Una fracción significativa de las muertes humanas por enfermedad puede ser causada por mutaciones que interfieren con el empalme normal; principalmente mediante la creación de sitios de empalme crípticos. [41] [38]
Las transcripciones con empalmes incorrectos se pueden detectar fácilmente y sus secuencias se pueden introducir en las bases de datos en línea. Normalmente se las describe como transcripciones "con empalmes alternativos", lo que puede resultar confuso porque el término no distingue entre el empalme alternativo real, biológicamente relevante, y el ruido de procesamiento debido a errores de empalme. Una de las cuestiones centrales en el campo del empalme alternativo es determinar las diferencias entre estas dos posibilidades. Muchos científicos han argumentado que la hipótesis nula debería ser el ruido de empalme, lo que hace recaer la carga de la prueba sobre quienes afirman que el empalme alternativo es biológicamente relevante. Según esos científicos, la afirmación de la función debe ir acompañada de pruebas convincentes de que se producen múltiples productos funcionales a partir del mismo gen. [42] [43]
Si bien los intrones no codifican productos proteicos, son fundamentales para la regulación de la expresión génica. Algunos intrones codifican ARN funcionales mediante un procesamiento posterior después del empalme para generar moléculas de ARN no codificante . [44] El empalme alternativo se utiliza ampliamente para generar múltiples proteínas a partir de un solo gen. Además, algunos intrones desempeñan papeles esenciales en una amplia gama de funciones reguladoras de la expresión génica, como la descomposición mediada por genes sin sentido [45] y la exportación de ARNm. [46]
Tras el descubrimiento inicial de los intrones en los genes codificadores de proteínas del núcleo eucariota, se debatió mucho si los intrones de los organismos modernos se heredaron de un ancestro común antiguo (hipótesis de los intrones tempranos) o si aparecieron en los genes en un momento bastante reciente del proceso evolutivo (hipótesis de los intrones tardíos). Otra teoría sostiene que el espliceosoma y la estructura intrón-exón de los genes son una reliquia del mundo del ARN (hipótesis de los intrones primero). [47] Todavía se debate mucho sobre hasta qué punto es más correcta una de estas hipótesis, pero el consenso popular actual es que tras la formación de la primera célula eucariota, los intrones del grupo II del endosimbionte bacteriano invadieron el genoma del huésped. Al principio, estos intrones que se autoempalmaban se escindían del precursor del ARNm, pero con el tiempo algunos de ellos perdieron esa capacidad y su escisión tuvo que ser ayudada en trans por otros intrones del grupo II. Con el tiempo, se desarrolló una serie de intrones transactivos específicos que se convirtieron en los precursores de los ARNm pequeños del espliceosoma. La eficiencia del empalme mejoró mediante la asociación con proteínas estabilizadoras para formar el espliceosoma primitivo. [48] [49] [50] [51]
Los primeros estudios de secuencias de ADN genómico de una amplia gama de organismos muestran que la estructura intrón-exón de genes homólogos en diferentes organismos puede variar ampliamente. [52] Estudios más recientes de genomas eucariotas completos han demostrado que las longitudes y densidades (intrones/gen) de los intrones varían considerablemente entre especies relacionadas. Por ejemplo, mientras que el genoma humano contiene un promedio de 8,4 intrones/gen (139.418 en el genoma), el hongo unicelular Encephalitozoon cuniculi contiene solo 0,0075 intrones/gen (15 intrones en el genoma). [53] Dado que los eucariotas surgieron de un ancestro común ( descendencia común ), debe haber habido una ganancia o pérdida extensa de intrones durante el tiempo evolutivo. [54] [55] Se cree que este proceso está sujeto a la selección, con una tendencia hacia la ganancia de intrones en especies más grandes debido a sus tamaños de población más pequeños, y lo contrario en especies más pequeñas (particularmente unicelulares). [56] Los factores biológicos también influyen en qué genes de un genoma pierden o acumulan intrones. [57] [58] [59]
El empalme alternativo de exones dentro de un gen después de la escisión de un intrón actúa para introducir una mayor variabilidad de las secuencias de proteínas traducidas a partir de un solo gen, lo que permite generar múltiples proteínas relacionadas a partir de un solo gen y una única transcripción de ARNm precursora. El control del empalme alternativo de ARN se realiza mediante una red compleja de moléculas de señalización que responden a una amplia gama de señales intracelulares y extracelulares.
Los intrones contienen varias secuencias cortas que son importantes para un empalme eficiente, como sitios aceptores y donantes en cada extremo del intrón, así como un sitio de ramificación, que son necesarios para que el espliceosoma realice un empalme adecuado . Se sabe que algunos intrones mejoran la expresión del gen en el que están contenidos mediante un proceso conocido como mejora mediada por intrones (IME).
Las regiones de ADN transcritas activamente forman frecuentemente bucles R que son vulnerables al daño del ADN . En genes de levadura altamente expresados, los intrones inhiben la formación de bucles R y la aparición de daño al ADN. [60] El análisis de todo el genoma tanto en levaduras como en humanos reveló que los genes que contienen intrones tienen niveles reducidos de bucles R y daño al ADN reducido en comparación con los genes sin intrones de expresión similar. [60] La inserción de un intrón dentro de un gen propenso a bucles R también puede suprimir la formación y recombinación de bucles R. Bonnet et al. (2017) [60] especularon que la función de los intrones en el mantenimiento de la estabilidad genética puede explicar su mantenimiento evolutivo en ciertas ubicaciones, particularmente en genes altamente expresados.
La presencia física de intrones promueve la resistencia celular a la inanición a través de la represión mejorada por intrones de los genes de proteínas ribosómicas de las vías de detección de nutrientes. [61]
Los intrones pueden perderse o ganarse a lo largo del tiempo evolutivo, como lo demuestran muchos estudios comparativos de genes ortólogos . Los análisis posteriores han identificado miles de ejemplos de eventos de pérdida y ganancia de intrones, y se ha propuesto que la aparición de eucariotas, o las etapas iniciales de la evolución eucariota, involucraron una invasión de intrones. [62] Se han identificado dos mecanismos definitivos de pérdida de intrones, la pérdida de intrones mediada por transcriptasa inversa (RTMIL) y las deleciones genómicas, y se sabe que ocurren. [63] Sin embargo, los mecanismos definitivos de ganancia de intrones siguen siendo esquivos y controvertidos. Hasta ahora se han informado al menos siete mecanismos de ganancia de intrones: transposición de intrones, inserción de transposones, duplicación genómica en tándem, transferencia de intrones, ganancia de intrones durante la reparación de rotura de doble cadena (DSBR), inserción de un intrón del grupo II e intronización. En teoría, debería ser más fácil deducir el origen de los intrones obtenidos recientemente debido a la falta de mutaciones inducidas por el huésped, pero incluso los intrones obtenidos recientemente no surgieron de ninguno de los mecanismos antes mencionados. Por lo tanto, estos hallazgos plantean la pregunta de si los mecanismos propuestos de ganancia de intrones no logran describir el origen mecanicista de muchos intrones nuevos porque no son mecanismos precisos de ganancia de intrones, o si existen otros procesos, aún por descubrir, que generen intrones nuevos. [64]
En la transposición de intrones, el mecanismo de ganancia de intrones más comúnmente postulado, se cree que un intrón empalmado se empalma de manera inversa en su propio ARNm o en otro ARNm en una posición que anteriormente no tenía intrones. Luego, este ARNm que contiene intrones se transcribe de manera inversa y el ADNc que contiene intrones resultante puede causar una ganancia de intrones a través de una recombinación completa o parcial con su locus genómico original.
Se ha demostrado que las inserciones de transposones generan miles de nuevos intrones en diversas especies eucariotas. [65] Las inserciones de transposones a veces dan como resultado la duplicación de esta secuencia en cada lado del transposón. Tal inserción podría intronizar el transposón sin alterar la secuencia codificante cuando un transposón se inserta en la secuencia AGGT o codifica los sitios de empalme dentro de la secuencia del transposón. Cuando los transposones que generan intrones no crean duplicaciones del sitio diana, los elementos incluyen ambos sitios de empalme GT (5') y AG (3') y, por lo tanto, se empalman de manera precisa sin afectar la secuencia codificante de la proteína. [65] Todavía no se entiende por qué estos elementos se empalman, ya sea por casualidad o por alguna acción preferencial del transposón.
En la duplicación genómica en tándem, debido a la similitud entre los sitios de empalme de consenso del donante y el aceptor, que se parecen mucho a AGGT, la duplicación genómica en tándem de un segmento exónico que alberga una secuencia AGGT genera dos sitios de empalme potenciales. Cuando el espliceosoma lo reconoce, la secuencia entre el AGGT original y el duplicado se empalmará, lo que dará como resultado la creación de un intrón sin alterar la secuencia codificante del gen. La reparación de roturas de doble cadena a través de la unión de extremos no homólogos se identificó recientemente como una fuente de ganancia de intrones cuando los investigadores identificaron repeticiones directas cortas que flanqueaban el 43% de los intrones ganados en Daphnia. [64] Sin embargo, estos números deben compararse con el número de intrones conservados flanqueados por repeticiones en otros organismos, para que tengan relevancia estadística. Para la inserción de intrones del grupo II, se propuso que la retroubicación de un intrón del grupo II en un gen nuclear causaba una ganancia de intrones espliceosómica reciente.
Se ha planteado la hipótesis de que la transferencia de intrones da como resultado una ganancia de intrones cuando un parálogo o pseudogén gana un intrón y luego transfiere este intrón mediante recombinación a una ubicación sin intrones en su parálogo hermano. La intronización es el proceso por el cual las mutaciones crean nuevos intrones a partir de una secuencia anteriormente exónica. Por lo tanto, a diferencia de otros mecanismos propuestos de ganancia de intrones, este mecanismo no requiere la inserción o generación de ADN para crear un nuevo intrón. [64]
El único mecanismo hipotético de ganancia reciente de intrones que carece de evidencia directa es el de la inserción de intrones del grupo II, que cuando se demuestra in vivo, elimina la expresión génica. [66] Por lo tanto, es probable que los intrones del grupo II sean los supuestos ancestros de los intrones espliceosomales, que actúan como retroelementos específicos del sitio y ya no son responsables de la ganancia de intrones. [67] [68] La duplicación genómica en tándem es el único mecanismo propuesto con evidencia experimental in vivo que lo respalda: una duplicación en tándem intragénica corta puede insertar un intrón nuevo en un gen codificador de proteínas, dejando la secuencia peptídica correspondiente sin cambios. [69] Este mecanismo también tiene evidencia indirecta extensa que respalda la idea de que la duplicación genómica en tándem es un mecanismo predominante para la ganancia de intrones. Es posible probar otros mecanismos propuestos in vivo, en particular la ganancia de intrones durante DSBR, la transferencia de intrones y la intronización, aunque estos mecanismos deben demostrarse in vivo para solidificarlos como mecanismos reales de ganancia de intrones. Análisis genómicos adicionales, especialmente cuando se realizan a nivel de población, pueden cuantificar la contribución relativa de cada mecanismo, posiblemente identificando sesgos específicos de cada especie que pueden arrojar luz sobre las distintas tasas de ganancia de intrones entre diferentes especies. [64]
Estructura:
Empalme:
Función
Otros: