Los genes Hox , un subconjunto de los genes homeobox , son un grupo de genes relacionados que especifican regiones del plan corporal de un embrión a lo largo del eje cabeza-cola de los animales. Las proteínas Hox codifican y especifican las características de la "posición", asegurando que las estructuras correctas se formen en los lugares correctos del cuerpo. Por ejemplo, los genes Hox en los insectos especifican qué apéndices se forman en un segmento (por ejemplo, patas, antenas y alas en las moscas de la fruta), y los genes Hox en los vertebrados especifican los tipos y la forma de las vértebras que se formarán. En los animales segmentados, las proteínas Hox confieren identidad segmentaria o posicional, pero no forman los segmentos reales en sí.
Los estudios sobre los genes Hox en larvas ciliadas han demostrado que sólo se expresan en los futuros tejidos adultos. En larvas con metamorfosis gradual, los genes Hox se activan en los tejidos del cuerpo larvario, generalmente en la región del tronco, que se mantendrán durante la metamorfosis. En larvas con metamorfosis completa, los genes Hox se expresan principalmente en los rudimentos juveniles y están ausentes en los tejidos larvarios transitorios. Las larvas de la especie hemicordada Schizocardium californicum y la larva pilidium de Nemertea no expresan genes Hox. [1] [2]
Se puede hacer una analogía de los genes Hox con el papel del director de una obra de teatro que anuncia qué escena deben representar los actores a continuación. Si el director de la obra anuncia las escenas en el orden incorrecto, la obra en general se presentará en el orden incorrecto. De manera similar, las mutaciones en los genes Hox pueden dar lugar a que partes del cuerpo y extremidades estén en el lugar incorrecto a lo largo del cuerpo. Al igual que un director de obra de teatro, los genes Hox no actúan en la obra ni participan en la formación de las extremidades.
El producto proteico de cada gen Hox es un factor de transcripción . Cada gen Hox contiene una secuencia de ADN bien conservada conocida como homeobox, de la cual el término "Hox" era originalmente una contracción. Sin embargo, en el uso actual el término Hox ya no es equivalente a homeobox, porque los genes Hox no son los únicos genes que poseen una secuencia homeobox; por ejemplo, los humanos tienen más de 200 genes homeobox, de los cuales 39 son genes Hox. [3] [4] Los genes Hox son, por lo tanto, un subconjunto de los genes del factor de transcripción homeobox. En muchos animales, la organización de los genes Hox en el cromosoma es la misma que el orden de su expresión a lo largo del eje anteroposterior del animal en desarrollo y, por lo tanto, se dice que muestran colinealidad. [5] [6] La producción de productos de genes Hox en una ubicación incorrecta en el cuerpo está asociada con metaplasia y predispone a la enfermedad oncológica, por ejemplo, el esófago de Barrett es el resultado de la codificación Hox alterada y es un precursor del cáncer de esófago . [7]
Los productos de los genes Hox son las proteínas Hox. Las proteínas Hox son un subconjunto de los factores de transcripción, que son proteínas capaces de unirse a secuencias de nucleótidos específicas en el ADN llamadas potenciadores a través de los cuales activan o reprimen cientos de otros genes. La misma proteína Hox puede actuar como represor en un gen y activador en otro. La capacidad de las proteínas Hox para unirse al ADN se confiere por una parte de la proteína denominada homeodominio . El homeodominio es un dominio de unión al ADN de 60 aminoácidos de longitud (codificado por su secuencia de ADN correspondiente de 180 pares de bases , el homeobox). Esta secuencia de aminoácidos se pliega en un motivo de "hélice-giro-hélice" (es decir, pliegue del homeodominio ) que se estabiliza mediante una tercera hélice. La cadena polipeptídica de consenso se muestra a continuación: [8] Las proteínas Hox a menudo actúan en asociación con cofactores, como las proteínas PBC y Meis codificadas por tipos muy diferentes de genes homeobox. [9]
Hélice 1 Hélice 2 Hélice 3/4 ______________ __________ _________________RRRKRTAYTRYQLLELEKEFLFNRYLTRRRRIELAHSLNLTERHIKIWFQNRRMKWKKEN....|....|....|....|....|....|....|....|....|....| ....|....| 10 20 30 40 50 60

Los genes homeobox, y por lo tanto el motivo proteico homeodominio, se encuentran en la mayoría de los eucariotas . Los genes Hox, al ser un subconjunto de los genes homeobox, surgieron más recientemente en la evolución dentro del reino animal o Metazoa . Dentro del reino animal, los genes Hox están presentes en los bilaterales [10] (animales con un eje cabeza-cola claro), y también se han encontrado en Cnidaria como las anémonas de mar . [11] Esto implica que los genes Hox surgieron hace más de 550 millones de años. En los bilaterales, los genes Hox a menudo se organizan en grupos de genes, aunque hay muchas excepciones en las que los genes se han separado por reordenamientos cromosómicos. [12] La comparación de secuencias de homeodominio entre proteínas Hox a menudo revela una mayor similitud entre especies que dentro de una especie; Esta observación condujo a la conclusión de que los grupos de genes Hox evolucionaron temprano en la evolución animal a partir de un único gen Hox a través de duplicación en tándem y divergencia posterior, y que un grupo de genes Hox prototípico que contenía al menos siete genes Hox diferentes estaba presente en el ancestro común de todos los animales bilaterales. [10] [13]
En la mayoría de los animales bilaterales , los genes Hox se expresan en dominios escalonados a lo largo del eje de la cabeza a la cola del embrión, lo que sugiere que su papel en la especificación de la posición es una característica antigua compartida. [14] La conservación funcional de las proteínas Hox se puede demostrar por el hecho de que una mosca puede funcionar en gran medida con una proteína Hox de pollo en lugar de la suya propia. [15] Entonces, a pesar de tener un último ancestro común que vivió hace más de 550 millones de años, [16] la versión de pollo y mosca del mismo gen Hox son lo suficientemente similares como para apuntar a los mismos genes posteriores en las moscas.

Drosophila melanogaster es un modelo importante para comprender la generación y evolución del plan corporal. Los principios generales de la función y la lógica del gen Hox dilucidados en las moscas se aplicarán a todos los organismos bilaterales, incluidos los humanos. Drosophila , como todos los insectos, tiene ocho genes Hox. Estos se agrupan en dos complejos, ambos ubicados en el cromosoma 3. El complejo Antennapedia (que no debe confundirse con el gen Antp ) consta de cinco genes: labial ( lab ), proboscipedia ( pb ), deformado ( Dfd ), peines sexuales reducidos ( Scr ) y Antennapedia ( Antp ). El complejo Bithorax, llamado así por el gen Ultrabithorax, consta de los tres genes restantes: Ultrabithorax ( Ubx ), abdominal-A ( abd-A ) y abdominal-B ( abd-B ).
El gen lab es el gen que se expresa más anteriormente. Se expresa en la cabeza, principalmente en el segmento intercalar (un segmento sin apéndices entre la antena y la mandíbula), y también en el intestino medio. La pérdida de la función del lab da como resultado que el embrión de Drosophila no pueda internalizar las estructuras de la boca y la cabeza que inicialmente se desarrollan en el exterior de su cuerpo (un proceso llamado involución de la cabeza). La falla de la involución de la cabeza altera o elimina las glándulas salivales y la faringe. El gen lab se llamó así inicialmente porque altera el apéndice labial; sin embargo, el gen lab no se expresa en el segmento labial, y el fenotipo del apéndice labial es probablemente el resultado de la amplia desorganización resultante de la falla de la involución de la cabeza. [17]
El gen pb es responsable de la formación de los palpos labiales y maxilares. Algunas evidencias muestran que pb interactúa con Scr . [18]
El gen Dfd es responsable de la formación de los segmentos maxilar y mandibular en la cabeza larvaria. [19] Los fenotipos mutantes de Dfd son similares a los de labial. La pérdida de la función de Dfd en el embrión da como resultado una falla en la involución de la cabeza (ver gen labial), con una pérdida de las estructuras de la cabeza larvaria. Las mutaciones en el adulto tienen como resultado la eliminación de partes de la cabeza o transformaciones de la cabeza a identidad torácica. [17]
El gen Scr es responsable del desarrollo cefálico y torácico en el embrión y el adulto de Drosophila . [20]
El segundo segmento torácico, o T2, desarrolla un par de patas y un par de alas. El gen Antp especifica esta identidad al promover la formación de patas y permitir (pero no activar directamente) la formación de alas. Una mutación dominante de Antp , causada por una inversión cromosómica , hace que Antp se exprese en el disco imaginal antenal, de modo que, en lugar de formar una antena, el disco forma una pata, lo que da como resultado una pata que sale de la cabeza de la mosca. [ cita requerida ]

El tercer segmento torácico, o T3, contiene un par de patas y un par de halterios (alas muy reducidas que funcionan para mantener el equilibrio durante el vuelo). Ubx modela T3 en gran medida reprimiendo genes involucrados en la formación del ala. La lámina del ala está compuesta por dos capas de células que se adhieren firmemente entre sí y reciben nutrientes de varias venas del ala. Uno de los muchos genes que Ubx reprime es el de ampollas, que activa proteínas involucradas en la adhesión célula-célula, y el de spalt, que modela la ubicación de las venas del ala. En mutantes con pérdida de función de Ubx , Ubx ya no reprime los genes del ala y los halterios se desarrollan como un segundo par de alas, lo que da lugar a las famosas moscas de cuatro alas. Cuando Ubx se expresa incorrectamente en el segundo segmento torácico, como ocurre en moscas con la mutación potenciadora "Cbx", reprime los genes del ala y las alas se desarrollan como halterios, lo que da lugar a una mosca de cuatro cabestros. [21]
En Drosophila , abd-A se expresa a lo largo de la mayor parte del abdomen, desde los segmentos abdominales 1 (A1) hasta A8. La expresión de abd-A es necesaria para especificar la identidad de la mayoría de los segmentos abdominales. Una función principal de abd-A en insectos es reprimir la formación de extremidades. En mutantes con pérdida de función de abd-A , los segmentos abdominales A2 a A8 se transforman en una identidad más parecida a A1. Cuando abd-A se expresa ectópicamente en todo el embrión, todos los segmentos anteriores de A4 se transforman en una identidad abdominal similar a A4. [17] El gen abd-A también afecta al patrón de generación de cutícula en el ectodermo y al patrón de generación de músculo en el mesodermo . [18]
El gen abd-B se transcribe en dos formas diferentes: una proteína reguladora y una proteína morfogénica. El gen regulador abd-B suprime las estructuras epidérmicas ventrales embrionarias en los segmentos octavo y noveno del abdomen de Drosophila . Tanto la proteína reguladora como la proteína morfogénica están involucradas en el desarrollo del segmento de la cola. [18]
Las proteínas con un alto grado de similitud de secuencia también se asume generalmente que exhiben un alto grado de similitud funcional, es decir, se supone que las proteínas Hox con homeodominios idénticos tienen propiedades de unión al ADN idénticas (a menos que se sepa que secuencias adicionales influyen en la unión al ADN). Para identificar el conjunto de proteínas entre dos especies diferentes que tienen más probabilidades de ser más similares en función, se utilizan esquemas de clasificación. Para las proteínas Hox, existen tres esquemas de clasificación diferentes: basado en inferencia filogenética, basado en sintenia y basado en similitud de secuencia. [22] Los tres esquemas de clasificación proporcionan información conflictiva para las proteínas Hox expresadas en la mitad del eje corporal ( Hox6-8 y Antp, Ubx y abd-A ). Un enfoque combinado utilizó información basada en inferencia filogenética de las diferentes especies y trazó los tipos de secuencia de proteínas en el árbol filogenético de la especie. El enfoque identificó las proteínas que mejor representan las formas ancestrales ( Hox7 y Antp ) y las proteínas que representan versiones nuevas derivadas (o que se perdieron en un ancestro y ahora faltan en numerosas especies). [23]
Los genes Hox actúan en muchos niveles dentro de las jerarquías de genes del desarrollo: en el nivel "ejecutivo" regulan genes que a su vez regulan grandes redes de otros genes (como la vía genética que forma un apéndice). También regulan directamente los llamados genes realizadores o genes efectores que actúan en la base de dichas jerarquías para formar en última instancia los tejidos, estructuras y órganos de cada segmento. La segmentación implica procesos como la morfogénesis (diferenciación de células precursoras en sus células especializadas terminales), la asociación estrecha de grupos de células con destinos similares, la escultura de estructuras y límites de segmentos mediante la muerte celular programada y el movimiento de células desde donde nacen hasta donde funcionarán en última instancia, por lo que no es sorprendente que los genes diana de los genes Hox promuevan la división celular, la adhesión celular, la apoptosis y la migración celular. [5]
La secuencia de ADN unida por la proteína homeodominio contiene la secuencia de nucleótidos TAAT, siendo la T terminal 5' la más importante para la unión. [28] Esta secuencia se conserva en casi todos los sitios reconocidos por los homeodominios, y probablemente distingue dichas ubicaciones como sitios de unión al ADN. Los pares de bases que siguen a esta secuencia inicial se utilizan para distinguir entre las proteínas homeodominio, todas las cuales tienen sitios de reconocimiento similares. Por ejemplo, el nucleótido que sigue a la secuencia TAAT es reconocido por el aminoácido en la posición 9 de la proteína homeodominio. En la proteína materna Bicoid, esta posición está ocupada por la lisina , que reconoce y se une al nucleótido guanina . En Antennapedia, esta posición está ocupada por la glutamina , que reconoce y se une a la adenina . Si la lisina en Bicoid se reemplaza por glutamina, la proteína resultante reconocerá los sitios potenciadores de unión de Antennapedia. [29] [30]
Sin embargo, todos los factores de transcripción que contienen homeodominio se unen esencialmente a la misma secuencia de ADN. La secuencia unida por el homeodominio de una proteína Hox tiene sólo seis nucleótidos de longitud, y una secuencia tan corta se encontraría al azar muchas veces a lo largo del genoma, mucho más que el número de sitios funcionales reales. Especialmente para las proteínas Hox, que producen cambios tan dramáticos en la morfología cuando se expresan incorrectamente, esto plantea la pregunta de cómo cada factor de transcripción puede producir resultados tan específicos y diferentes si todos se unen a la misma secuencia. Un mecanismo que introduce una mayor especificidad de la secuencia de ADN a las proteínas Hox es la unión de cofactores proteicos. Dos de estos cofactores Hox son Extradenticle (Exd) y Homothorax (Hth). Exd y Hth se unen a las proteínas Hox y parecen inducir cambios conformacionales en la proteína Hox que aumentan su especificidad. [31]
Así como los genes Hox regulan a los genes realizadores, a su vez son regulados por otros genes. En Drosophila y algunos insectos (pero no en la mayoría de los animales), los genes Hox son regulados por genes gap y genes pair-rule , que a su vez son regulados por el ARNm suministrado por la madre . Esto da como resultado una cascada de factores de transcripción: los factores maternos activan los genes gap o pair-rule; los genes gap y pair-rule activan los genes Hox; luego, finalmente, los genes Hox activan los genes realizadores que hacen que los segmentos en el embrión en desarrollo se diferencien.
La regulación se logra a través de gradientes de concentración de proteínas, llamados campos morfogénicos . Por ejemplo, altas concentraciones de una proteína materna y bajas concentraciones de otras activarán un conjunto específico de genes gap o de regla de pares. En las moscas, la franja 2 en el embrión es activada por las proteínas maternas Bicoid y Hunchback, pero reprimida por las proteínas gap Giant y Kruppel. Por lo tanto, la franja 2 solo se formará donde haya Bicoid y Hunchback, pero no donde haya Giant y Kruppel. [32]
Se ha demostrado que las cadenas de microARN ubicadas en grupos Hox inhiben más genes hox anteriores ("fenómeno de prevalencia posterior"), posiblemente para ajustar mejor su patrón de expresión. [33]
Se ha demostrado que el ARN no codificante (ARNnc) es abundante en los grupos Hox. En los seres humanos, puede haber 231 ARNnc presentes. Uno de ellos, HOTAIR , silencia en trans (se transcribe a partir del grupo HOXC e inhibe los genes HOXD tardíos) al unirse a las proteínas del grupo Polycomb (PRC2). [34]
La estructura de la cromatina es esencial para la transcripción, pero también requiere que el grupo forme un bucle fuera del territorio cromosómico . [35]
En animales superiores, incluidos los humanos, el ácido retinoico regula la expresión diferencial de los genes Hox a lo largo del eje anteroposterior. [36] Los genes en los extremos 3' de los grupos Hox son inducidos por el ácido retinoico, lo que da como resultado dominios de expresión que se extienden más anteriormente en el cuerpo en comparación con los genes Hox 5' que no son inducidos por el ácido retinoico, lo que da como resultado dominios de expresión que permanecen más posteriores.
La PCR cuantitativa ha mostrado varias tendencias con respecto a la colinealidad: el sistema está en equilibrio y el número total de transcripciones depende del número de genes presentes según una relación lineal. [37]
En algunos organismos, especialmente en los vertebrados, los distintos genes Hox están situados muy cerca unos de otros en el cromosoma, en grupos o racimos. El orden de los genes en el cromosoma es el mismo que el de la expresión de los genes en el embrión en desarrollo, siendo el primer gen el que se expresa en el extremo anterior del organismo en desarrollo. La razón de esta colinealidad aún no se entiende por completo, pero podría estar relacionada con la activación de los genes Hox en una secuencia temporal mediante la descompresión gradual de la cromatina a lo largo de un racimos de genes.
Los genes Hox reciben su nombre de los fenotipos homeóticos que resultan cuando se altera su función, en los que un segmento se desarrolla con la identidad de otro (por ejemplo, patas en lugar de antenas). A los genes Hox de diferentes filos se les han dado nombres diferentes, lo que ha provocado confusión sobre la nomenclatura. El conjunto de genes Hox en Drosophila está formado por dos grupos, el complejo Antennapedia y el complejo Bithorax, que juntos se denominaban históricamente HOM-C (por complejo homeótico). Aunque históricamente los genes HOM-C se han referido a homólogos de Drosophila , mientras que los genes Hox se referían a homólogos de vertebrados, esta distinción ya no se hace, y tanto los genes HOM-C como los Hox se denominan genes Hox. [ cita requerida ]
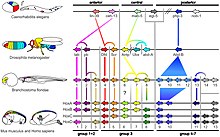
Los ratones y los humanos tienen 39 genes Hox en cuatro grupos: [38] [39]
Los ancestros de los vertebrados tenían un único grupo de genes Hox, [40] [41] [ cita requerida ] que se duplicó (dos veces) temprano en la evolución de los vertebrados por duplicaciones del genoma completo para dar cuatro grupos de genes Hox: Hoxa, Hoxb, Hoxc y Hoxd. Actualmente no está claro si estas duplicaciones ocurrieron antes o después de la divergencia de las lampreas y los mixinos de otros vertebrados. [42] La mayoría de los tetrápodos tienen cuatro grupos HOX, mientras que la mayoría de los peces teleósteos , incluidos el pez cebra y el medaka , tienen siete u ocho grupos de genes Hox debido a una duplicación adicional del genoma que ocurrió en su historia evolutiva. [43] [38] En el pez cebra, uno de los ocho grupos de genes Hox (un grupo Hoxd) ha perdido todos los genes codificadores de proteínas, y solo un único gen de microARN marca la ubicación del grupo original. [44] En algunos peces teleósteos, como el salmón , se produjo una duplicación genómica aún más reciente, duplicando los siete u ocho grupos de genes Hox para dar al menos 13 grupos. [45] Otro teleósteo, el pez mariposa de agua dulce , ha experimentado en cambio una pérdida significativa de grupos de genes HOX, con solo 5 grupos presentes. [46]
Los cuerpos de los vertebrados no están segmentados de la misma manera que los insectos; en promedio, son mucho más complejos, lo que lleva a una mayor infraestructura en su plan corporal en comparación con los insectos. Los genes HOX controlan la regulación y el desarrollo de muchas estructuras clave en el cuerpo, como los somitas , que forman las vértebras y las costillas, la dermis de la piel dorsal, los músculos esqueléticos de la espalda y los músculos esqueléticos de la pared corporal y las extremidades. Los genes HOX ayudan a diferenciar las células somitas en identidades más específicas y las dirigen a desarrollarse de manera diferente dependiendo de dónde se encuentren en el cuerpo. [47] Una gran diferencia entre vertebrados e invertebrados es la ubicación y la estratificación de los genes HOX. Los mecanismos fundamentales del desarrollo están fuertemente conservados entre los vertebrados, desde los peces hasta los mamíferos.
Debido al hecho de que los genes HOX están tan altamente conservados, la mayoría de las investigaciones se han realizado en organismos modelo mucho más simples, como los ratones. Una de las principales diferencias que se notó al comparar ratones y drosophila , en particular, tiene que ver con la ubicación y la estratificación de los genes HOX dentro del genoma . Los vertebrados tienen genes HOX que son homólogos a los de la mosca, ya que es uno de los genes más altamente conservados , pero la ubicación es diferente. Por ejemplo, hay más genes HOX en el lado 5' del segmento del ratón en comparación con los invertebrados. [48] Estos genes corresponden a la expresión en la cola, lo que tendría sentido ya que las moscas no tendrían nada similar a la cola que tienen todos los vertebrados. Además, en la mayoría de los vertebrados hay 39 miembros segregados en cuatro matrices de genes separados y estrechamente agrupados (A–D) en cuatro cromosomas separados , mientras que hay ocho genes HOX en total para la Drosophila. [49] Los grupos son mucho más redundantes y es menos probable que generen mutaciones . En las moscas, un gen puede mutar, lo que da como resultado que un halterio , algo fundamental para que puedan volar, se transforme en un ala, o que una antena se convierta en una pata; en el ratón, se deben eliminar simultáneamente de dos a cuatro genes para obtener una transformación completa similar. Algunos investigadores creen que, debido a la redundancia del plan de cúmulos HOX de vertebrados y más restringido en comparación con los cúmulos HOX de invertebrados, la capacidad de evolución de los cúmulos HOX de vertebrados es, por alguna razón estructural o funcional, mucho menor que la de sus contrapartes invertebradas. [50] Esta rápida capacidad de evolución se debe en parte a que los invertebrados experimentaron episodios mucho más dramáticos de radiación adaptativa y mutaciones. Más de 20 clados principales de invertebrados difieren tan radicalmente en la organización corporal, en parte debido a una mayor tasa de mutación, que se clasificaron formalmente como filos diferentes . [51] Todos los genes parálogos deben eliminarse para que haya cambios fenotípicos en su mayor parte. Esta es también una razón por la que las mutaciones homeóticas en vertebrados se observan tan raramente.
En los embriones de ratón, los genes HOX10, que son uno de los genes que se encuentran en la porción de la cola del animal, desactivan el sistema de "construcción de costillas" cuando el gen se activa. Los genes están activos en la parte inferior de la espalda, donde las vértebras no desarrollan costillas, e inactivos en la parte media de la espalda, lo que permite que se formen las costillas. Cuando los parálogos de HOX10 se inactivan experimentalmente, las vértebras de la parte inferior de la espalda desarrollan costillas. [52] Esta investigación impulsó una búsqueda evolutiva de estas mutaciones en todos los animales. Un ejemplo de esto es en lagartos y serpientes. En las serpientes, los genes HOX10 han perdido su capacidad de bloquear las costillas de esa manera. [53]
Los amphioxus como Branchiostoma floridae tienen un solo grupo Hox con 15 genes, conocidos como AmphiHox1 a AmphiHox15 . [54]
Seis genes Hox están dispersos en el genoma de Caenorhabditis elegans , un gusano redondo . [10] : fig. 3 Hydra y Nematostella vectensis , ambas en el filo Cnidaria , tienen algunos genes homeobox similares a Hox/ParaHox. [55] [11]
La expresión del gen Hox también se ha estudiado en braquiópodos , [56] anélidos , [57] y un conjunto de moluscos . [58]
Los genes Hox se denominan así porque las mutaciones en ellos causan transformaciones homeóticas . Las transformaciones homeóticas fueron identificadas y estudiadas por primera vez por William Bateson en 1894, quien acuñó el término "homeosis". Después del redescubrimiento de los principios genéticos de Mendel , Bateson y otros se dieron cuenta de que algunos ejemplos de homeosis en órganos florales y esqueletos de animales podían atribuirse a la variación en los genes.
La evidencia definitiva de una base genética de algunas transformaciones homeóticas se obtuvo aislando mutantes homeóticos. El primer mutante homeótico fue encontrado por Calvin Bridges en el laboratorio de Thomas Hunt Morgan en 1915. Este mutante muestra una duplicación parcial del tórax y por lo tanto se lo denominó Bithorax ( bx ). Transforma el tercer segmento torácico (T3) hacia el segundo (T2). Bithorax surgió espontáneamente en el laboratorio y se ha mantenido continuamente como stock de laboratorio desde entonces. [59]
Los estudios genéticos de Morgan y otros proporcionaron las bases para los análisis sistemáticos de Edward B. Lewis y Thomas Kaufman, que proporcionaron definiciones preliminares de los numerosos genes homeóticos de los complejos Bithorax y Antennapedia, y también demostraron que los fenotipos mutantes de la mayoría de estos genes podían rastrearse hasta defectos de patrón en el plan corporal embrionario.
Ed Lewis , Christiane Nüsslein-Volhard y Eric F. Wieschaus identificaron y clasificaron 15 genes de importancia clave para determinar el plan corporal y la formación de segmentos corporales de la mosca de la fruta D. melanogaster en 1980. [60] Por su trabajo, Lewis, Nüsslein-Volhard y Wieschaus recibieron el Premio Nobel de Fisiología o Medicina en 1995. [61]
En 1983, la homeobox fue descubierta independientemente por investigadores de dos laboratorios: Ernst Hafen, Michael Levine y William McGinnis (en el laboratorio de Walter Gehring en la Universidad de Basilea , Suiza) y Matthew P. Scott y Amy Weiner (en el laboratorio de Thomas Kaufman en la Universidad de Indiana en Bloomington ).
Los genes Hox desempeñan papeles fundamentales en el desarrollo de estructuras como las extremidades, los pulmones, el sistema nervioso y los ojos. Como observaron TR Lappin y sus colegas en 2006, "la conservación evolutiva ofrece un alcance ilimitado para la investigación experimental del control funcional de la red de genes Hox, lo que está proporcionando información importante sobre las enfermedades humanas". En el futuro, se pueden realizar más investigaciones para investigar los papeles de los genes Hox en la leucemia y el cáncer (como el cáncer de ovario epitelial). [38]