Los análisis de genes knockout CRISPR-Cas9 a escala de todo el genoma tienen como objetivo dilucidar la relación entre el genotipo y el fenotipo mediante la ablación de la expresión génica a escala de todo el genoma y el estudio de las alteraciones fenotípicas resultantes. El enfoque utiliza el sistema de edición genética CRISPR-Cas9 , junto con bibliotecas de ARN guía individuales (sgRNA) , que están diseñadas para dirigirse a cada gen del genoma. En los últimos años, el análisis CRISPR a escala de todo el genoma ha surgido como una herramienta poderosa para realizar análisis de pérdida de función a gran escala, con poco ruido, alta eficiencia de knockout y efectos mínimos fuera del objetivo.

Los primeros estudios en Caenorhabditis elegans [1] y Drosophila melanogaster [2] [3] mostraron pruebas sistemáticas de pérdida de función (LOF) a gran escala realizadas mediante mutagénesis de saturación , lo que demostró el potencial de este enfoque para caracterizar las vías genéticas e identificar genes con funciones únicas y esenciales. La técnica de mutagénesis de saturación se aplicó más tarde en otros organismos, por ejemplo, el pez cebra [4] [5] y los ratones. [6] [7]
Los enfoques específicos para la eliminación de genes surgieron en la década de 1980 con técnicas como la recombinación homóloga , [8] [9] la transescisión de ribozimas , [10] [11] y las tecnologías antisentido . [12] [13]
En el año 2000, la tecnología de interferencia de ARN (ARNi) había surgido como una técnica rápida, simple y económica para la eliminación selectiva de genes , y se utilizaba rutinariamente para estudiar la función genética in vivo en C. elegans . [14] [15] [16] [17] De hecho, en el lapso de solo unos pocos años después de su descubrimiento por Fire et al . (1998), [18] casi todos los ~19.000 genes en C. elegans habían sido analizados utilizando la eliminación basada en ARNi . [19]
La producción de bibliotecas de ARNi facilitó la aplicación de esta tecnología a escala del genoma, y los métodos basados en ARNi se convirtieron en el enfoque predominante para las pruebas de derribo de todo el genoma. [ cita requerida ]
Sin embargo, los métodos basados en RNAi para el cribado de genes knockdown en todo el genoma tienen sus limitaciones. Por un lado, los elevados efectos fuera del objetivo causan problemas con las observaciones de falsos positivos. [20] [21] Además, debido a que el RNAi reduce la expresión génica a nivel postranscripcional al dirigirse al ARN, los cribados basados en RNAi solo dan como resultado una supresión parcial y a corto plazo de los genes. Si bien el knockdown parcial puede ser deseable en ciertas situaciones, se necesitaba una tecnología con una eficiencia de focalización mejorada y menos efectos fuera del objetivo. [ cita requerida ]
Desde su identificación inicial como un sistema inmunológico adaptativo procariota, [22] el sistema CRISPR / Cas9 de tipo II bacteriano de repeticiones cortas de palíndromo agrupadas y regularmente interespaciadas (agrupadas y regularmente interespaciadas) se ha convertido en una herramienta simple y eficiente para generar mutaciones LOF dirigidas. [23] Se ha aplicado con éxito para editar genomas humanos y ha comenzado a desplazar al RNAi como la herramienta dominante en estudios de mamíferos. [24] En el contexto de los cribados de knockout de todo el genoma, estudios recientes han demostrado que los cribados CRISPR/Cas9 pueden lograr un agotamiento de proteínas altamente eficiente y completo, y superar los problemas fuera del objetivo observados con los cribados de RNAi. [25] [26] En resumen, la reciente aparición de CRISPR-Cas9 ha aumentado drásticamente nuestra capacidad para realizar cribados LOF a gran escala. La versatilidad y programabilidad de Cas9, junto con el bajo ruido, la alta eficiencia de knockout y los efectos mínimos fuera del objetivo, han hecho de CRISPR la plataforma elegida por muchos investigadores que se dedican a la selección y edición de genes. [24] [27]
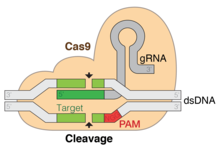
El sistema Cas9 / CRISPR (repeticiones cortas de palíndromo agrupadas y regularmente interespaciadas ) es una tecnología de edición genética que puede introducir roturas de doble cadena (DSB) en un locus genómico objetivo. Al utilizar un único ARN guía (sgRNA) , la endonucleasa Cas9 puede administrarse a una secuencia de ADN específica donde escinde la cadena de nucleótidos. [28] La especificidad del sgRNA está determinada por una secuencia de 20 nt, homóloga al locus genómico de interés, y la unión a Cas9 está mediada por una región de andamiaje constante del sgRNA. El sitio objetivo deseado debe ser seguido inmediatamente (5' a 3') por un motivo adyacente al protoespaciador (PAM) conservado de 3 nucleótidos. [29] [30] Para reparar las DSB, la célula puede utilizar la unión de extremos no homólogos altamente propensa a errores , o recombinación homóloga . Mediante el diseño de sgRNAs adecuados, se pueden introducir inserciones o deleciones planificadas en el genoma. En el contexto de los análisis de LOF en todo el genoma, el objetivo es provocar la alteración y la inactivación de genes. [ cita requerida ]
Para realizar knockouts CRISPR a escala de todo el genoma, se deben generar colecciones de sgRNA conocidas como bibliotecas de sgRNA o bibliotecas de knockouts CRISPR. El primer paso para crear una biblioteca de sgRNA es identificar las regiones genómicas de interés basándose en reglas conocidas de selección de sgRNA. [31] Por ejemplo, los sgRNA son más eficientes cuando se dirigen a las regiones codificantes de los genes y no a los UTR 5' y 3' . Los exones conservados se presentan como objetivos atractivos y se debe considerar la posición relativa al sitio de inicio de la transcripción. [31] En segundo lugar, se identifican y seleccionan todos los posibles sitios PAM. [31] Se debe analizar la actividad dentro y fuera del objetivo, al igual que el contenido de GC, y se deben evitar los tramos de homopolímero. [31] La endonucleasa Cas9 más utilizada, derivada de Streptococcus pyogenes , reconoce una secuencia PAM de NGG. [32]
Además, parece que ciertos nucleótidos se ven favorecidos en lugares específicos. La guanina se ve fuertemente favorecida sobre la citosina en la posición 20, justo al lado del motivo PAM, y en la posición 16 se prefiere la citosina sobre la guanina. [33] Para el nucleótido variable en el motivo PAM NGG, se ha demostrado que se prefiere la citosina y se desfavorece la timina. [33] Teniendo en cuenta estos criterios, la biblioteca de sgRNA se diseña computacionalmente en torno a los sitios PAM seleccionados. [31] [33] [34]
Se deben crear múltiples sgRNA (al menos 4 a 6) contra cada gen para limitar la detección de falsos positivos, y se deben incluir sgRNA de control negativo sin objetivos conocidos. [31] [33] Luego, los sgRNA se crean mediante síntesis in situ , se amplifican por PCR y se clonan en un sistema de administración de vectores.
El desarrollo de una nueva biblioteca de sgRNA es un proceso laborioso y que requiere mucho tiempo. En la práctica, los investigadores pueden seleccionar una biblioteca existente según su propósito experimental y las líneas celulares de interés. A febrero de 2020, los recursos más utilizados para los análisis de knockout CRISPR de todo el genoma han sido las dos bibliotecas Genome-Scale CRISPR Knock-Out (GeCKO) creadas por el laboratorio de Zhang. [35] Disponibles a través de Addgene, estas bibliotecas lentivirales se dirigen respectivamente a exones humanos y de ratón, y ambas están disponibles como un sistema de un vector (donde los sgRNA y Cas9 están presentes en el mismo plásmido) o como un sistema de dos vectores (donde los sgRNA y Cas9 están presentes en plásmidos separados). Cada biblioteca se entrega como dos medias bibliotecas, lo que permite a los investigadores realizar el análisis con 3 o 6 sgRNA/gen. [36]
Además de GeCKO, se han generado y puesto a disposición a través de Addgene otras bibliotecas CRISPR. Los laboratorios Sabatini y Lander cuentan actualmente con 7 bibliotecas humanas y de ratón independientes, incluidas subbibliotecas específicas para subgrupos distintos, como quinasas y genes ribosomales (Addgene #51043–51048). Además, las mejoras en la especificidad de los sgRNA han dado lugar a bibliotecas de "segunda generación", como las bibliotecas Brie (Addgene #73632) y Brunello (Addgene #73178) generadas por los laboratorios Doench y Root, y la biblioteca Toronto knockout (TKO) (Addgene #1000000069) generada por el laboratorio Moffat. [36]

La eliminación selectiva de genes mediante CRISPR/Cas9 requiere el uso de un sistema de administración para introducir el sgRNA y el Cas9 en la célula. Aunque hay varios sistemas de administración diferentes disponibles para CRISPR, [37] [38] los análisis de pérdida de función en todo el genoma se llevan a cabo predominantemente utilizando vectores lentivirales de tercera generación. [35] [39] [40] Estos vectores lentivirales pueden transducir de manera eficiente una amplia gama de tipos de células e integrarse de manera estable en el genoma de células en división y no en división. [41] [42] Las partículas lentivirales de tercera generación se producen mediante la cotransfección de células de riñón embrionario humano (HEK) 293T con:
El sobrenadante que contiene partículas lentivirales se recolecta, se concentra y luego se usa para infectar las células objetivo. [45] El protocolo exacto para la producción lentiviral variará según el objetivo de la investigación y la biblioteca aplicada. [35] [43] [44] Si se utiliza un sistema de dos vectores, por ejemplo, las células se transducen secuencialmente con Cas9 y sgRNA en un procedimiento de dos pasos. [35] [44] Aunque es más complejo, esto tiene la ventaja de un título más alto para el virus de la biblioteca sgRNA. [35]
En general, existen dos formatos diferentes de cribado de knockout CRISPR de todo el genoma: en matriz y en agrupación. En un cribado en matriz, cada pocillo contiene un sgRNA específico y conocido que se dirige a un gen específico. [46] Dado que el sgRNA responsable de cada fenotipo se conoce en función de la ubicación del pocillo, los fenotipos se pueden identificar y analizar sin necesidad de secuenciación genética. Este formato permite la medición de fenotipos celulares más específicos, tal vez por fluorescencia o luminiscencia, y permite a los investigadores utilizar más tipos de bibliotecas y métodos de administración. [46] Sin embargo, para los cribados LOF a gran escala, los formatos en matriz se consideran de baja eficiencia y costosos en términos de recursos financieros y materiales porque las poblaciones de células tienen que aislarse y cultivarse individualmente. [46]
En una prueba agrupada, las células cultivadas en un solo recipiente se transducen en masa con vectores virales que contienen colectivamente toda la biblioteca de sgRNA. Para garantizar que la cantidad de células infectadas por más de una partícula que contiene sgRNA sea limitada, se utiliza una multiplicidad de infección (MOI) baja (normalmente 0,3-0,6). [46] [47] La evidencia hasta ahora ha sugerido que cada sgRNA debería estar representado en un mínimo de 200 células. [48] [23] Se seleccionarán las células transducidas, seguidas de una selección positiva o negativa para el fenotipo de interés, y será necesaria la secuenciación genética para identificar los sgRNA integrados. [46]
Tras la selección fenotípica, se extrae el ADN genómico de los clones seleccionados, junto con una población de células de control. [23] [46] [49] En los protocolos más comunes para knockouts de todo el genoma, se crea una "biblioteca de secuenciación de próxima generación (NGS)" mediante una reacción en cadena de la polimerasa (PCR) de dos pasos. [23] [46] El primer paso amplifica la región sgRNA, utilizando cebadores específicos para la secuencia de integración lentiviral, y el segundo paso agrega secuencias i5 e i7 de Illumina. [23] La NGS de los productos de PCR permite identificar los sgRNA recuperados, y se puede utilizar un paso de cuantificación para determinar la abundancia relativa de cada sgRNA. [23]
El paso final en la pantalla es evaluar computacionalmente los sgRNA significativamente enriquecidos o empobrecidos, rastrearlos hasta sus genes correspondientes y, a su vez, determinar qué genes y vías podrían ser responsables del fenotipo observado. Actualmente hay varios algoritmos disponibles para este propósito, siendo el más popular el método Model-based Analysis of Genome-wide CRISPR/Cas9 Knockout (MAGeCK). [50] Desarrollado específicamente para pantallas de knockout CRISPR/Cas9 en 2014, MAGeCK demostró un mejor rendimiento en comparación con algoritmos alternativos en ese momento, [50] y desde entonces ha demostrado resultados sólidos y alta sensibilidad en diferentes condiciones experimentales. [51] A partir de 2015, el algoritmo MAGeCK se ha ampliado para introducir mediciones de control de calidad y dar cuenta de la eficiencia de knockout de sgRNA que anteriormente se pasaba por alto. [51] También se integró una herramienta de visualización basada en la web (VISPR), que permite a los usuarios explorar de forma interactiva los resultados, el análisis y los controles de calidad. [51]
En los últimos años, la prueba CRISPR de todo el genoma ha surgido como una herramienta poderosa para estudiar las intrincadas redes de señalización celular. [52] La señalización celular es esencial para una serie de procesos biológicos fundamentales, incluidos el crecimiento celular, la proliferación, la diferenciación y la apoptosis .
Un ejemplo práctico es la identificación de genes necesarios para la señalización proliferativa en células cancerosas. Las células se transducen con una biblioteca de sgRNA CRISPR y se estudia su crecimiento a lo largo del tiempo. Al comparar la abundancia de sgRNA en células seleccionadas con un control, se puede identificar qué sgRNA se agotan y, a su vez, qué genes pueden ser responsables del defecto de proliferación. Estas pruebas se han utilizado para identificar genes esenciales para el cáncer en la leucemia mieloide aguda [53] y el neuroblastoma [54] y para describir diferencias específicas de tumores entre líneas de células cancerosas [55] .
Las terapias dirigidas contra el cáncer están diseñadas para atacar genes, proteínas o entornos específicos que contribuyen al crecimiento o supervivencia de las células tumorales. Sin embargo, después de un período de tratamiento prolongado con estas terapias, las células tumorales pueden desarrollar resistencia. Aunque los mecanismos detrás de la resistencia a los fármacos contra el cáncer son poco conocidos, las posibles causas incluyen: alteración del objetivo, degradación del fármaco, escape de la apoptosis y alteraciones epigenéticas. [56] La resistencia es bien conocida y plantea un problema grave en el tratamiento del cáncer. [ cita requerida ]
Para superar este problema, se puede identificar un socio letal sintético . Se pueden utilizar exámenes LOF de todo el genoma utilizando CRISPR-Cas9 para detectar socios letales sintéticos. [57] Para esto, una línea celular de tipo salvaje y una línea celular tumoral que contiene la mutación que causa resistencia se transducen con una biblioteca de sgRNA CRISPR. Las dos líneas celulares se cultivan y se analizan las células muertas o subrepresentadas para identificar posibles genes socios letales sintéticos. Un estudio reciente de Hinze et al. (2019) [58] utilizó este método para identificar una interacción letal sintética entre el fármaco de quimioterapia asparaginasa y dos genes en la vía de señalización de Wnt NKD2 y LGR6.
Debido a sus pequeños genomas y al número limitado de proteínas codificadas, los virus aprovechan las proteínas del huésped para su entrada, replicación y transmisión. La identificación de dichas proteínas, también denominadas factores de dependencia del huésped (HDF), es particularmente importante para identificar objetivos terapéuticos. En los últimos años, muchos grupos han utilizado con éxito la tecnología CRISPR/Cas9 en todo el genoma como estrategia de detección de HDF en infecciones virales. [59]
Marceau et al. (2017) [60] ofrecen un ejemplo que se propuso analizar los factores del huésped asociados con la infección por dengue y hepatitis C (VHC) (dos virus de la familia Flaviviridae ). Se descubrió que ELAVL1 , una proteína de unión al ARN codificada por el gen ELAVL1, era un receptor crítico para la entrada del VHC, y se demostró una divergencia notable en los factores de dependencia del huésped entre los dos flaviviridae. [60]
Otras aplicaciones reportadas de las pruebas CRISPR de todo el genoma incluyen el estudio de: metabolismo mitocondrial, [61] resistencia a toxinas bacterianas, [62] impulsores genéticos de la metástasis, [63] resistencia a medicamentos contra el cáncer, [64] muerte celular inducida por el virus del Nilo Occidental, [65] y redes de genes de células inmunes. [66] [67]
Esta sección se ocupará específicamente de los análisis CRISPR de todo el genoma. Para una revisión de las limitaciones de CRISPR, consulte Lino et al. (2018) [38].
En última instancia, los análisis CRISPR de todo el genoma estarán limitados por las propiedades de la biblioteca de sgRNA elegida. Cada biblioteca contendrá un conjunto diferente de sgRNA y la cobertura promedio por gen puede variar. Las bibliotecas disponibles actualmente tienden a estar sesgadas hacia los sgRNA que se dirigen a los exones codificadores de proteínas tempranos (5'), en lugar de los que se dirigen a los dominios proteicos más funcionales. [58] Este problema fue destacado por Hinze et al. (2019), [58] quienes observaron que los genes asociados con la sensibilidad a la asparaginasa no obtuvieron puntaje en su análisis de todo el genoma de células de leucemia resistentes a la asparaginasa.
Si no se dispone de una biblioteca adecuada, la creación y amplificación de una nueva biblioteca de sgRNA es un proceso largo que puede llevar muchos meses. Los desafíos potenciales incluyen: (i) diseño eficaz de sgRNA; (ii) asegurar una cobertura completa de sgRNA en todo el genoma; (iii) diseño de la estructura principal del vector lentiviral; (iv) producción de cantidades suficientes de lentivirus de alta calidad; (v) superar la baja eficiencia de transformación; (vi) escalamiento adecuado del cultivo bacteriano. [68]
Uno de los mayores obstáculos para la detección de CRISPR en todo el genoma es garantizar una cobertura adecuada de la biblioteca de sgRNA en toda la población celular. [23] La evidencia hasta el momento ha sugerido que cada sgRNA debería estar representado y mantenido en un mínimo de 200 a 300 células. [23] [48]
Teniendo en cuenta que el protocolo estándar utiliza una multiplicidad de infección de ~0,3 y una eficiencia de transducción del 30-40 % [44] [23], la cantidad de células necesarias para producir y mantener una cobertura adecuada se vuelve muy grande. A modo de ejemplo, la biblioteca de sgRNA humana más popular es la biblioteca GeCKO v2 creada por el laboratorio de Zhang; [30] contiene 123 411 sgRNA. Los estudios que utilizan esta biblioteca transducen comúnmente más de 1x10 8 células [58] [59] [69]
Como CRISPR sigue mostrando poco ruido y efectos mínimos fuera del objetivo, una estrategia alternativa es reducir la cantidad de sgRNA por gen para una selección primaria. Se utilizan puntos de corte menos estrictos para la selección de aciertos y luego se utilizan sgRNA adicionales en una selección secundaria más específica. Este enfoque lo demuestran Doench et al . (2016), [33] quienes encontraron que >92% de los genes recuperados utilizando el protocolo estándar también se recuperaron utilizando menos sgRNA por gen. Sugieren que esta estrategia podría ser útil en estudios donde la ampliación es prohibitivamente costosa. [ cita requerida ]
Los vectores lentivirales tienen ciertas limitaciones generales. Por un lado, es imposible controlar dónde se integra el genoma viral en el genoma del huésped, y esto puede afectar a funciones importantes de la célula. Vannucci et al. [70] proporcionan una excelente revisión de los vectores virales junto con sus ventajas y desventajas generales. En el contexto específico de los análisis CRISPR de todo el genoma, producir y transducir las partículas lentivirales es relativamente laborioso y lleva mucho tiempo, y lleva aproximadamente dos semanas en total. [44] Además, debido a que el ADN se integra en el genoma del huésped, la administración lentiviral conduce a la expresión a largo plazo de Cas9, lo que puede provocar efectos fuera del objetivo. [ cita requerida ]
En un análisis en matriz, cada pocillo contiene un sgRNA específico y conocido que se dirige a un gen específico. Por lo tanto, los análisis en matriz permiten obtener un perfil detallado de una sola célula, pero están limitados por los altos costos y la mano de obra necesaria para aislar y cultivar la gran cantidad de poblaciones de células individuales. [46] Los análisis CRISPR agrupados convencionales son relativamente simples y rentables de realizar, pero se limitan al estudio de toda la población celular. Esto significa que los fenotipos raros pueden ser más difíciles de identificar y solo se pueden seleccionar fenotipos crudos para, por ejemplo, la supervivencia celular, la proliferación o la expresión del gen reportero. [ cita requerida ]
La elección del medio de cultivo podría afectar la relevancia fisiológica de los hallazgos de los experimentos de cultivo celular debido a las diferencias en la composición y las concentraciones de nutrientes. [71] Recientemente se mostró un sesgo sistemático en los conjuntos de datos generados para las pruebas de silenciamiento de genes CRISPR y RNAi (especialmente para genes metabólicos), [72] y para el perfil metabólico de líneas celulares cancerosas . [71] Por ejemplo, se encontró una dependencia más fuerte de ASNS (asparagina sintetasa) en líneas celulares cultivadas en DMEM , que carece de asparagina, en comparación con las líneas celulares cultivadas en RPMI o F12 (que contienen asparagina). [72] Se podría evitar dicho sesgo utilizando un medio uniforme para todas las líneas celulares examinadas e idealmente, utilizando un medio de crecimiento que represente mejor los niveles fisiológicos de nutrientes. Recientemente, se desarrollaron tipos de medios como Plasmax [73] y Human Plasma Like Medium (HPLM), [74] .
Las tecnologías emergentes apuntan a combinar los análisis CRISPR agrupados con la resolución detallada de la secuenciación masiva y paralela de ARN de una sola célula (RNA-seq) . Los estudios que utilizan “CRISP-seq”, [75] “CROP-seq”, [76] y “PERTURB-seq” [77] han demostrado lecturas genómicas ricas, que identifican con precisión las firmas de expresión génica para genes knockouts individuales en un grupo complejo de células. Estos métodos tienen el beneficio adicional de producir perfiles transcripcionales de las células inducidas por sgRNA. [78]
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ){{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ){{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ){{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace )