AlphaFold es un programa de inteligencia artificial (IA) desarrollado por DeepMind , una filial de Alphabet , que realiza predicciones de la estructura de las proteínas . [1] El programa está diseñado como un sistema de aprendizaje profundo . [2]
El software AlphaFold ha tenido dos versiones principales. Un equipo de investigadores que utilizó AlphaFold 1 (2018) obtuvo el primer lugar en la clasificación general de la 13.ª Evaluación Crítica de Predicción de Estructuras (CASP) en diciembre de 2018. El programa fue particularmente exitoso en predecir la estructura más precisa para objetivos calificados como los más difíciles. por los organizadores del concurso, donde no existían estructuras plantilla disponibles a partir de proteínas con una secuencia parcialmente similar. Un equipo que utilizó AlphaFold 2 (2020) repitió su colocación en la competencia CASP14 en noviembre de 2020. [3] El equipo logró un nivel de precisión mucho más alto que cualquier otro grupo. [2] [4] Obtuvo una puntuación superior a 90 para alrededor de dos tercios de las proteínas en la prueba de distancia global (GDT) de CASP , una prueba que mide el grado en que la estructura predicha de un programa computacional es similar a la estructura determinada por un experimento de laboratorio, con 100 es una coincidencia completa, dentro del límite de distancia utilizado para calcular GDT. [2] [5]
Los resultados de AlphaFold 2 en CASP14 se describieron como "asombrosos" [6] y "transformacionales". [7] Algunos investigadores observaron que la precisión no es lo suficientemente alta para un tercio de sus predicciones, y que no revela el mecanismo o las reglas del plegamiento de proteínas para que el problema del plegamiento de proteínas se considere resuelto. [8] [9] Sin embargo, ha habido un respeto generalizado por el logro técnico, y el análisis sugiere que AlphaFold 2 es lo suficientemente preciso como para predecir incluso los efectos de una sola mutación. [10] El 15 de julio de 2021, el artículo AlphaFold 2 se publicó en Nature como una publicación de acceso avanzado junto con software de código abierto y una base de datos con capacidad de búsqueda de proteomas de especies . [11] [12] [13] Actualmente se está desarrollando una versión más avanzada de AlphaFold. Permite modelar complejos proteicos con ácidos nucleicos, pequeños ligandos, iones y residuos modificados. [14]

Las proteínas consisten en cadenas de aminoácidos que se pliegan espontáneamente, en un proceso llamado plegamiento de proteínas , para formar estructuras tridimensionales (3-D) de las proteínas. La estructura tridimensional es crucial para la función biológica de la proteína. Sin embargo, comprender cómo la secuencia de aminoácidos puede determinar la estructura tridimensional es un gran desafío, y esto se denomina "problema de plegamiento de proteínas". [15] El "problema del plegamiento de proteínas" implica comprender la termodinámica de las fuerzas interatómicas que determinan la estructura estable plegada, el mecanismo y la vía a través de los cuales una proteína puede alcanzar su estado final plegado con extrema rapidez, y cómo se modifica la estructura nativa de una proteína. puede predecirse a partir de su secuencia de aminoácidos. [dieciséis]
Actualmente las estructuras de las proteínas se determinan experimentalmente mediante técnicas como la cristalografía de rayos X , la criomicroscopía electrónica y la resonancia magnética nuclear , técnicas que son a la vez costosas y requieren mucho tiempo. [15] Tales esfuerzos han identificado las estructuras de alrededor de 170.000 proteínas en los últimos 60 años, mientras que hay más de 200 millones de proteínas conocidas en todas las formas de vida. [5] Si es posible predecir la estructura de las proteínas únicamente a partir de la secuencia de aminoácidos, sería de gran ayuda para avanzar en la investigación científica. Sin embargo, la paradoja de Levinthal muestra que si bien una proteína puede plegarse en milisegundos, el tiempo que lleva calcular todas las estructuras posibles al azar para determinar la verdadera estructura nativa es mayor que la edad del universo conocido, lo que hizo que predecir las estructuras de las proteínas fuera un gran desafío. en biología para científicos. [15]
A lo largo de los años, los investigadores han aplicado numerosos métodos computacionales para resolver el problema de la predicción de la estructura de las proteínas , pero su precisión no se ha acercado a las técnicas experimentales, excepto para proteínas pequeñas y simples, lo que limita su valor. CASP , que se lanzó en 1994 para desafiar a la comunidad científica a producir sus mejores predicciones de la estructura de las proteínas, descubrió que en 2016 se pueden lograr puntuaciones GDT de aproximadamente 40 sobre 100 para las proteínas más difíciles. [5] AlphaFold comenzó a competir en el 2018 CASP utilizando una técnica de aprendizaje profundo de inteligencia artificial (IA) . [15]
Se sabe que DeepMind ha entrenado el programa en más de 170.000 proteínas de un depósito público de secuencias y estructuras de proteínas. El programa utiliza una forma de red de atención , una técnica de aprendizaje profundo que se centra en hacer que la IA identifique partes de un problema mayor y luego las una para obtener la solución general. [2] La capacitación general se llevó a cabo con una potencia de procesamiento de entre 100 y 200 GPU . [2] Entrenar el sistema en este hardware tomó "unas pocas semanas", después de lo cual el programa tardaría "una cuestión de días" en converger para cada estructura. [17]
AlphaFold 1 (2018) se basó en el trabajo desarrollado por varios equipos en la década de 2010, un trabajo que analizó los grandes bancos de datos de secuencias de ADN relacionadas ahora disponibles de muchos organismos diferentes (la mayoría sin estructuras 3D conocidas), para tratar de encontrar cambios en diferentes residuos . eso parecía estar correlacionado, aunque los residuos no eran consecutivos en la cadena principal. Tales correlaciones sugieren que los residuos pueden estar físicamente cerca unos de otros, aunque no cerca en la secuencia, lo que permite estimar un mapa de contactos . Sobre la base de un trabajo reciente anterior a 2018, AlphaFold 1 amplió esto para estimar una distribución de probabilidad de qué tan cerca podrían estar los residuos, convirtiendo el mapa de contacto en un mapa de distancia probable. También utilizó métodos de aprendizaje más avanzados que antes para desarrollar la inferencia. Combinando un potencial estadístico basado en esta distribución de probabilidad con la energía libre local calculada de la configuración, el equipo pudo utilizar el descenso de gradiente hasta una solución que se adaptaba mejor a ambas. [ se necesita aclaración ] [18] [19]
Más técnicamente, Torrisi et al resumieron en 2019 el enfoque de AlphaFold versión 1 de la siguiente manera: [20]
Lo fundamental de AlphaFold es un predictor de mapas de distancia implementado como una red neuronal residual muy profunda con 220 bloques residuales que procesan una representación de dimensionalidad 64 × 64 × 128, correspondiente a características de entrada calculadas a partir de dos fragmentos de 64 aminoácidos. Cada bloque residual tiene tres capas, incluida una capa convolucional dilatada de 3 × 3: los bloques pasan por la dilatación de los valores 1, 2, 4 y 8. En total, el modelo tiene 21 millones de parámetros. La red utiliza una combinación de entradas 1D y 2D, incluidos perfiles evolutivos de diferentes fuentes y características de coevolución. Junto con un mapa de distancias en forma de un histograma de distancias muy fino, AlphaFold predice los ángulos Φ y Ψ para cada residuo que se utilizan para crear la estructura 3D predicha inicial. Los autores de AlphaFold concluyeron que la profundidad del modelo, su gran tamaño de cultivo, el gran conjunto de entrenamiento de aproximadamente 29.000 proteínas, las técnicas modernas de aprendizaje profundo y la riqueza de información del histograma de distancias predichas ayudaron a AlphaFold a lograr una alta precisión de predicción del mapa de contacto. .
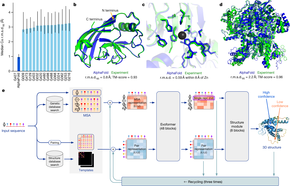

La versión 2020 del programa ( AlphaFold 2 , 2020) es significativamente diferente de la versión original que ganó CASP 13 en 2018, según el equipo de DeepMind. [22] [23]
El equipo de DeepMind había identificado que su enfoque anterior, que combinaba la física local con un potencial guía derivado del reconocimiento de patrones, tenía una tendencia a sobreestimar las interacciones entre residuos que estaban cerca en la secuencia en comparación con las interacciones entre residuos más alejados a lo largo de la cadena. Como resultado, AlphaFold 1 tendía a preferir modelos con una estructura ligeramente más secundaria ( hélices alfa y láminas beta ) de lo que era en realidad (una forma de sobreajuste ). [24]
El diseño del software utilizado en AlphaFold 1 contenía una serie de módulos, cada uno entrenado por separado, que se utilizaron para producir el potencial guía que luego se combinó con el potencial energético basado en la física. AlphaFold 2 reemplazó esto con un sistema de subredes acopladas en un único modelo diferenciable de extremo a extremo, basado completamente en el reconocimiento de patrones, que fue entrenado de manera integrada como una única estructura integrada. [23] [25] La física local, en forma de refinamiento de energía basado en el modelo AMBER , se aplica solo como un paso de refinamiento final una vez que la predicción de la red neuronal ha convergido, y solo ajusta ligeramente la estructura predicha. [24]
Una parte clave del sistema 2020 son dos módulos, que se cree que están basados en un diseño de transformador , que se utilizan para refinar progresivamente un vector de información para cada relación (o " borde " en la terminología de la teoría de grafos) entre un residuo de aminoácido de la proteína y otro residuo de aminoácido (estas relaciones están representadas por la matriz que se muestra en verde); y entre cada posición de aminoácido y cada secuencia diferente en la alineación de la secuencia de entrada (estas relaciones están representadas por la matriz que se muestra en rojo). [25] Internamente, estas transformaciones de refinamiento contienen capas que tienen el efecto de reunir datos relevantes y filtrar datos irrelevantes (el "mecanismo de atención") para estas relaciones, de una manera dependiente del contexto, aprendido de los datos de entrenamiento. Estas transformaciones se repiten, la información actualizada producida en un paso se convierte en la entrada del siguiente, con la información mejorada de residuo/residuo alimentando la actualización de la información de residuo/secuencia, y luego la información mejorada de residuo/secuencia alimentando la actualización de la información sobre residuos/residuos. [25] A medida que avanza la iteración, según un informe, el "algoritmo de atención... imita la forma en que una persona podría armar un rompecabezas: primero conectando piezas en pequeños grupos, en este caso grupos de aminoácidos, y luego buscando formas de unir los grupos en un todo más grande". [5]
La salida de estas iteraciones luego informa al módulo de predicción de estructura final, [25] que también usa transformadores, [26] y luego se itera a su vez. En un ejemplo presentado por DeepMind, el módulo de predicción de estructura logró una topología correcta para la proteína objetivo en su primera iteración, con un GDT_TS de 78, pero con una gran cantidad (90%) de violaciones estereoquímicas, es decir, ángulos de enlace no físicos o longitudes. En iteraciones posteriores, el número de violaciones estereoquímicas disminuyó. En la tercera iteración, el GDT_TS de la predicción se acercaba a 90, y en la octava iteración, el número de violaciones estereoquímicas se acercaba a cero. [27]
El equipo de AlphaFold declaró en noviembre de 2020 que cree que AlphaFold se puede desarrollar aún más, con margen para seguir mejorando la precisión. [22] Un análisis reciente sugiere que la versión actual de AlphaFold2 ya es lo suficientemente precisa como para predecir incluso los efectos de una sola mutación. [10]
Los datos de entrenamiento se restringieron originalmente a cadenas peptídicas individuales. Sin embargo, la actualización de octubre de 2021, denominada AlphaFold-Multimer, incluyó complejos de proteínas en sus datos de entrenamiento. DeepMind afirmó que esta actualización logró aproximadamente el 70% de las veces predecir con precisión las interacciones proteína-proteína. [28]

En diciembre de 2018, AlphaFold de DeepMind ocupó el primer lugar en la clasificación general de la 13.ª Evaluación crítica de técnicas para la predicción de la estructura de proteínas (CASP). [29] [30]
El programa predijo con especial éxito la estructura más precisa para objetivos calificados como los más difíciles por los organizadores de la competición, donde no había estructuras plantilla disponibles a partir de proteínas con una secuencia parcialmente similar. AlphaFold dio la mejor predicción para 25 de 43 objetivos proteicos en esta clase, [30] [31] [32] logrando una puntuación media de 58,9 en la puntuación de la prueba de distancia global (GDT) del CASP , por delante de 52,5 y 52,4 de los dos Los siguientes equipos mejor ubicados, [33] que también estaban utilizando el aprendizaje profundo para estimar las distancias de contacto. [34] [35] En general, en todos los objetivos, el programa logró una puntuación GDT de 68,5. [36]
En enero de 2020, las implementaciones y el código ilustrativo de AlphaFold 1 se lanzaron de código abierto en GitHub . [37] [15] pero, como se indica en el archivo "Léame" en ese sitio web: "Este código no se puede usar para predecir la estructura de una secuencia de proteínas arbitraria. Se puede usar para predecir la estructura solo en el conjunto de datos CASP13 (enlaces a continuación). El código de generación de funciones está estrechamente vinculado a nuestra infraestructura interna y a las herramientas externas, por lo que no podemos abrirlo". Por lo tanto, en esencia, el código depositado no es adecuado para uso general sino sólo para las proteínas CASP13. La compañía no ha anunciado planes para hacer que su código esté disponible públicamente a partir del 5 de marzo de 2021.
En noviembre de 2020, la nueva versión de DeepMind, AlphaFold 2, ganó CASP14. [17] [38] En general, AlphaFold 2 hizo la mejor predicción para 88 de los 97 objetivos. [6]
En la medida de precisión de la prueba de distancia global (GDT) preferida de la competencia , el programa logró una puntuación media de 92,4 (sobre 100), lo que significa que más de la mitad de sus predicciones obtuvieron una puntuación superior al 92,4% por tener sus átomos en más de 100 puntos. o menos en el lugar correcto, [39] [40] un nivel de precisión que, según se informa , es comparable a técnicas experimentales como la cristalografía de rayos X. [22] [7] [36] En 2018, AlphaFold 1 solo había alcanzado este nivel de precisión en dos de todas sus predicciones. [6] El 88% de las predicciones en la competición de 2020 tuvieron una puntuación GDT_TS superior a 80. En el grupo de objetivos clasificados como los más difíciles, AlphaFold 2 logró una puntuación media de 87.
Medido por la desviación cuadrática media (RMS-D) de la ubicación de los átomos de carbono alfa de la cadena principal de la proteína, que tiende a estar dominada por el desempeño de los valores atípicos peor ajustados, el 88% de las predicciones de AlphaFold 2 tenía una desviación RMS de menos de 4 Å para el conjunto de átomos de C-alfa superpuestos. [6] El 76% de las predicciones lograron mejores que 3 Å, y el 46% tuvo una precisión RMS del átomo de C-alfa mejor que 2 Å, [6] con una desviación RMS mediana en sus predicciones de 2,1 Å para un conjunto de átomos de CA superpuestos. . [6] AlphaFold 2 también logró una precisión en el modelado de cadenas laterales de superficie descrita como "realmente extraordinaria".
Para verificar adicionalmente AlphaFold-2, los organizadores de la conferencia se acercaron a cuatro grupos experimentales líderes en busca de estructuras que encontraban particularmente desafiantes y que no habían podido determinar. En los cuatro casos, los modelos tridimensionales producidos por AlphaFold 2 fueron lo suficientemente precisos como para determinar las estructuras de estas proteínas mediante reemplazo molecular . Entre ellos se encontraba la diana T1100 (Af1503), una pequeña proteína de membrana estudiada por experimentadores durante diez años. [5]
De las tres estructuras en las que AlphaFold 2 tuvo menos éxito en predecir, dos se obtuvieron mediante métodos de RMN de proteínas , que definen la estructura de las proteínas directamente en una solución acuosa, mientras que AlphaFold se entrenó principalmente en estructuras de proteínas en cristales . El tercero existe en la naturaleza como un complejo multidominio que consta de 52 copias idénticas del mismo dominio , una situación que AlphaFold no fue programado para considerar. Para todos los objetivos con un solo dominio, excluyendo solo una proteína muy grande y las dos estructuras determinadas por RMN, AlphaFold 2 logró una puntuación GDT_TS superior a 80.
En 2022, DeepMind no ingresó a CASP15, pero la mayoría de los participantes utilizaron AlphaFold o herramientas que incorporaban AlphaFold. [41]
AlphaFold 2 con una puntuación de más de 90 en la prueba de distancia global (GDT) de CASP se considera un logro significativo en biología computacional [5] y un gran progreso hacia un gran desafío de la biología que ya lleva décadas. [7] El ganador del Premio Nobel y biólogo estructural Venki Ramakrishnan calificó el resultado como "un avance sorprendente en el problema del plegamiento de proteínas", [5] y agregó que "Ha ocurrido décadas antes de que muchas personas en el campo hubieran predicho. Será emocionante "Vemos las muchas maneras en que cambiará fundamentalmente la investigación biológica". [17]
Impulsado por comunicados de prensa de CASP y DeepMind, [42] [17] El éxito de AlphaFold 2 recibió una amplia atención de los medios. [43] Además de artículos periodísticos en la prensa científica especializada, como Nature , [7] Science , [5] MIT Technology Review , [2] y New Scientist , [44] [45] la historia fue ampliamente cubierta por los principales medios de comunicación. periódicos nacionales, [46] [47] [48] [49] así como servicios de noticias generales y publicaciones semanales, como Fortune , [50] [23] The Economist , [22] Bloomberg , [36] Der Spiegel , [51] y El espectador . [52] En Londres, The Times hizo de la historia su portada fotográfica, con dos páginas más de cobertura interna y un editorial. [53] [54] Un tema frecuente fue que se espera que la capacidad de predecir estructuras de proteínas con precisión basándose en la secuencia de aminoácidos constituyentes tenga una amplia variedad de beneficios en el espacio de las ciencias biológicas, incluida la aceleración del descubrimiento de fármacos avanzados y la posibilidad de una mejor comprensión de las enfermedades. [7] [55] Al escribir sobre el evento, MIT Technology Review señaló que la IA había "resuelto un gran desafío de la biología de cincuenta años de antigüedad". [2] El mismo artículo continuó señalando que el algoritmo de IA podría "predecir la forma de las proteínas dentro del ancho de un átomo". [2]
Como resume Der Spiegel, las reservas sobre esta cobertura se centran principalmente en dos áreas: "Aún queda mucho por hacer" y: "Ni siquiera sabemos cómo lo hacen". [56]
Aunque el líder del proyecto, John Jumper, realizó una presentación de 30 minutos sobre AlphaFold 2 el segundo día de la conferencia CASP (1 de diciembre), [57] se ha descrito como "de un nivel extremadamente alto, llena de ideas e insinuaciones, pero casi totalmente carente de detalles". [58] [ ¿ fuente poco confiable? ] A diferencia de otros grupos de investigación que se presentaron en CASP14, la presentación de DeepMind no fue grabada y no está disponible públicamente. Se espera que DeepMind publique un artículo científico que describa AlphaFold 2 en el volumen de actas [ ¿cuándo? ] de la conferencia CASP; pero no se sabe si irá más allá de lo dicho en la presentación.
En declaraciones a El País , el investigador Alfonso Valencia afirma: "Lo más importante que nos deja este avance es saber que este problema tiene solución, que es posible solucionarlo... Sólo sabemos el resultado. Google no proporciona el software". Y esta es la parte frustrante del logro porque no beneficiará directamente a la ciencia". [49] Sin embargo, por mucho que Google y DeepMind publiquen, puede ayudar a otros equipos a desarrollar sistemas de inteligencia artificial similares, un beneficio "indirecto". [49] A finales de 2019, DeepMind lanzó gran parte del código de la primera versión de AlphaFold como código abierto; pero sólo cuando el trabajo ya estaba en marcha en el mucho más radical AlphaFold 2. Otra opción que podría tomarse podría ser hacer que la predicción de estructuras AlphaFold 2 esté disponible como un servicio de suscripción en línea de caja negra. Se ha estimado que la convergencia de una única secuencia requiere del orden de 10.000 dólares en tiempo de cálculo al por mayor . [59] Pero esto negaría a los investigadores el acceso a los estados internos del sistema, la oportunidad de aprender de manera más cualitativa qué da lugar al éxito de AlphaFold 2 y el potencial de nuevos algoritmos que podrían ser más livianos y eficientes y aun así lograr tales resultados. Los temores de una posible falta de transparencia por parte de DeepMind se contrastan con cinco décadas de fuertes inversiones públicas en el banco de datos de proteínas abierto y luego también en repositorios abiertos de secuencias de ADN , sin los cuales los datos para entrenar AlphaFold 2 no habrían existido. [60] [61] [62]
Es de destacar que el 18 de junio de 2021, Demis Hassabis tuiteó: "¡Breve actualización sobre algunos avances interesantes en #AlphaFold! Hemos estado trabajando a fondo en nuestro documento de métodos completo (actualmente bajo revisión) con el código fuente abierto adjunto y en proporcionando amplio acceso gratuito a AlphaFold para la comunidad científica. ¡Más muy pronto!" [63]
Sin embargo, todavía no está claro hasta qué punto las predicciones estructurales realizadas por AlphaFold 2 se mantendrán para las proteínas unidas en complejos con otras proteínas y otras moléculas. [64] Esto no fue parte de la competencia CASP en la que participó AlphaFold, y no fue una eventualidad para la que fue diseñado internamente. Mientras que las estructuras que AlphaFold 2 predijo eran para proteínas que tenían fuertes interacciones con otras copias de sí mismas o con otras estructuras, estos fueron los casos en los que las predicciones de AlphaFold 2 tendieron a ser menos refinadas y menos confiables. Como una gran fracción de las máquinas biológicas más importantes de una célula comprende tales complejos, o se relacionan con cómo las estructuras de las proteínas se modifican cuando entran en contacto con otras moléculas, esta es un área que seguirá siendo el foco de considerable atención experimental. [64]
Con tan poco conocimiento aún sobre los patrones internos que AlphaFold 2 aprende a hacer sus predicciones, aún no está claro hasta qué punto el programa puede verse afectado en su capacidad para identificar nuevos pliegues, si dichos pliegues no están bien representados en la proteína existente. estructuras conocidas en bases de datos de estructuras. [8] [64] Tampoco se sabe bien hasta qué punto las estructuras de proteínas en tales bases de datos, en su abrumadora mayoría de proteínas que han sido posibles cristalizar en rayos X, son representativas de proteínas típicas que aún no han sido cristalizadas. Y tampoco está claro qué tan representativas son las estructuras de proteínas congeladas en los cristales de las estructuras dinámicas encontradas en las células in vivo . Las dificultades de AlphaFold 2 con las estructuras obtenidas mediante métodos de RMN de proteínas pueden no ser una buena señal.
Por lo tanto, las estructuras de AlphaFold 2 pueden ser sólo una ayuda limitada en tales contextos. [8] [64] Además, según el columnista de Science Derek Lowe , debido a que la predicción de la unión de moléculas pequeñas incluso entonces todavía no es muy buena, la predicción computacional de los objetivos farmacológicos simplemente no está en condiciones de asumir el papel de "columna vertebral". del descubrimiento de fármacos corporativo, por lo que "la determinación de la estructura de las proteínas simplemente no es un paso limitante en el descubrimiento de fármacos en general". [65] También se ha observado que incluso con una estructura para una proteína, comprender cómo funciona, qué hace y cómo encaja dentro de procesos biológicos más amplios todavía puede ser un gran desafío. [66] Sin embargo, si un mejor conocimiento de la estructura de las proteínas pudiera conducir a una mejor comprensión de los mecanismos de las enfermedades individuales y, en última instancia, a mejores objetivos farmacológicos, o a una mejor comprensión de las diferencias entre los modelos humanos y animales, en última instancia, eso podría conducir a mejoras. [67]
Además, debido a que AlphaFold procesa secuencias exclusivas de proteínas por diseño, no se consideran otras biomoléculas asociadas. Sobre el impacto de la ausencia de metales, cofactores y, más visiblemente, modificaciones cotraduccionales y postraduccionales como la glicosilación de proteínas de los modelos AlphaFold, Elisa Fadda (Universidad de Maynooth, Irlanda) y Jon Agirre (Universidad de York, Reino Unido) destacaron la Es necesario que los científicos revisen bases de datos como UniProt-KB para detectar posibles componentes faltantes, ya que estos pueden desempeñar un papel importante no solo en el plegamiento sino también en la función de las proteínas. [68] Sin embargo, los autores destacaron que muchos modelos AlphaFold eran lo suficientemente precisos como para permitir la introducción de modificaciones posteriores a la predicción . [68]
Finalmente, algunos han observado que incluso una respuesta perfecta al problema de la predicción de proteínas aún dejaría preguntas sobre el problema del plegamiento de las proteínas : comprender en detalle cómo ocurre realmente el proceso de plegamiento en la naturaleza (y cómo a veces también pueden plegarse mal ). [69]
Pero incluso con tales advertencias, AlphaFold 2 fue descrito como un gran paso técnico y un logro intelectual. [70] [71]
La base de datos de estructura de proteínas AlphaFold se lanzó el 22 de julio de 2021, como un esfuerzo conjunto entre AlphaFold y EMBL-EBI . En el momento del lanzamiento, la base de datos contiene modelos predichos por AlphaFold de estructuras proteicas de casi todo el proteoma UniProt de humanos y 20 organismos modelo , lo que suma más de 365.000 proteínas. La base de datos no incluye proteínas con menos de 16 o más de 2700 residuos de aminoácidos , [72] pero para los humanos están disponibles en el archivo por lotes completo. [73] AlphaFold planeó agregar más secuencias a la colección, siendo el objetivo inicial (a principios de 2022) cubrir la mayor parte del conjunto UniRef90 de más de 100 millones de proteínas. Al 15 de mayo de 2022, estaban disponibles 992.316 predicciones. [74]
En julio de 2021, UniProt-KB e InterPro [75] se actualizaron para mostrar las predicciones de AlphaFold cuando estén disponibles. [76]
El 28 de julio de 2022, el equipo subió a la base de datos las estructuras de alrededor de 200 millones de proteínas de 1 millón de especies, cubriendo casi todas las proteínas conocidas del planeta. [77]
AlphaFold DB utiliza un modelo monomérico similar a la versión CASP14. Como resultado, se esperan muchas de las mismas limitaciones: [78]
AlphaFold se ha utilizado para predecir estructuras de proteínas del SARS-CoV-2 , el agente causante del COVID-19 . Las estructuras de estas proteínas estaban pendientes de detección experimental a principios de 2020. [83] [7] Los científicos del Instituto Francis Crick del Reino Unido examinaron los resultados antes de publicarlos en la comunidad de investigación en general. El equipo también confirmó una predicción precisa contra la proteína de pico del SARS-CoV-2 determinada experimentalmente que se compartió en el Protein Data Bank , una base de datos internacional de acceso abierto, antes de publicar las estructuras determinadas computacionalmente de las moléculas de proteína poco estudiadas. [84] El equipo reconoció que, aunque estas estructuras proteicas podrían no ser objeto de esfuerzos de investigación terapéutica en curso, contribuirán a la comprensión de la comunidad sobre el virus SARS-CoV-2. [84] Específicamente, la predicción de AlphaFold 2 de la estructura de la proteína ORF3a fue muy similar a la estructura determinada por investigadores de la Universidad de California, Berkeley, utilizando microscopía crioelectrónica . Se cree que esta proteína específica ayuda al virus a salir de la célula huésped una vez que se replica. También se cree que esta proteína desempeña un papel en el desencadenamiento de la respuesta inflamatoria a la infección. [85]