La succinato deshidrogenasa ( SDH ) o succinato-coenzima Q reductasa ( SQR ) o complejo respiratorio II es un complejo enzimático , que se encuentra en muchas células bacterianas y en la membrana mitocondrial interna de los eucariotas . Es la única enzima que participa tanto en el ciclo del ácido cítrico como en la cadena de transporte de electrones . [1] El análisis histoquímico que muestra un nivel alto de succinato deshidrogenasa en el músculo demuestra un alto contenido mitocondrial y un alto potencial oxidativo. [2]
En el paso 6 del ciclo del ácido cítrico , SQR cataliza la oxidación de succinato a fumarato con la reducción de ubiquinona a ubiquinol . Esto ocurre en la membrana mitocondrial interna al acoplar las dos reacciones.

Los SQR mitocondriales y muchos bacterianos se componen de cuatro subunidades estructuralmente diferentes : dos hidrofílicas y dos hidrofóbicas . Las dos primeras subunidades, una flavoproteína (SdhA) y una proteína hierro-azufre (SdhB), forman una cabeza hidrófila donde tiene lugar la actividad enzimática del complejo. SdhA contiene un cofactor de flavina adenina dinucleótido (FAD) unido covalentemente y el sitio de unión succinato y SdhB contiene tres grupos de hierro-azufre: [2Fe-2S], [4Fe-4S] y [3Fe-4S]. Las dos segundas subunidades son subunidades de anclaje de membrana hidrofóbicas, SdhC y SdhD. Las mitocondrias humanas contienen dos isoformas distintas de SdhA (subunidades Fp tipo I y tipo II); estas isoformas también se encuentran en Ascaris suum y Caenorhabditis elegans . [3] Las subunidades forman un complejo de citocromo b unido a una membrana con seis hélices transmembrana que contienen un grupo hemo b y un sitio de unión de ubiquinona . También se encuentran dos moléculas de fosfolípidos , una cardiolipina y una fosfatidiletanolamina , en las subunidades SdhC y SdhD (no se muestran en la imagen). Sirven para ocupar el espacio hidrofóbico debajo del hemo b. Estas subunidades se muestran en la imagen adjunta. SdhA es verde, SdhB es verde azulado, SdhC es fucsia y SdhD es amarillo. Alrededor de SdhC y SdhD hay una membrana de fosfolípidos con el espacio intermembrana en la parte superior de la imagen. [4]
Se pueden reconocer dos sitios de unión de ubiquinona distintivos en la SDH de mamíferos: Q P proximal a la matriz y Q D distal a la matriz . El sitio de unión de ubiquinona Qp, que muestra una mayor afinidad por la ubiquinona, se encuentra en un espacio compuesto por SdhB, SdhC y SdhD. La ubiquinona está estabilizada por las cadenas laterales de His207 de la subunidad B, Ser27 y Arg31 de la subunidad C y Tyr83 de la subunidad D. El anillo de quinona está rodeado por Ile28 de la subunidad C y Pro160 de la subunidad B. Estos residuos , junto con Il209, Trp163 , y Trp164 de la subunidad B, y Ser27 (átomo de C) de la subunidad C, forman el entorno hidrófobo del bolsillo de unión de quinona Qp. [6] Por el contrario, el sitio de unión de ubiquinona QD , que se encuentra más cerca del espacio intermembrana, está compuesto únicamente de SdhD y tiene menor afinidad por la ubiquinona. [7]
SdhA proporciona el sitio de unión para la oxidación del succinato . Las cadenas laterales Thr254, His354 y Arg399 de la subunidad A estabilizan la molécula mientras que FAD oxida y transporta los electrones al primero de los grupos hierro-azufre , [2Fe-2S]. [8] Esto se puede ver en la imagen 5.
El sitio de unión del succinato y el sitio de unión de la ubiquinona están conectados por una cadena de centros redox que incluyen FAD y los grupos hierro - azufre . Esta cadena se extiende a lo largo de 40 Å a través del monómero enzimático . Todas las distancias de borde a borde entre los centros son menores que el límite sugerido de 14 Å para la transferencia fisiológica de electrones . [4] Esta transferencia de electrones se demuestra en la imagen 8.
Todas las subunidades de la SDH mitocondrial humana están codificadas nuclearmente. Después de la traducción, la subunidad SDHA se transloca como apoproteína a la matriz mitocondrial. Posteriormente, uno de los primeros pasos es la unión covalente del cofactor FAD (flavinilación covalente). Este proceso se ve reforzado por el factor 2 de ensamblaje de succinato deshidrogenasa ( SDHAF2 ; [9] también llamado Sdh5 en levaduras y SdhE en bacterias) y por algunos de los intermediarios del ciclo de Krebs. El fumarato estimula más fuertemente la flavinilación covalente de SDHA. [10] A través de estudios del sistema bacteriano, se ha demostrado que el mecanismo de unión de FAD implica un intermediario quinona:métido. [11] En el ensamblaje mitocondrial, pero no bacteriano, SDHA interactúa con un segundo factor de ensamblaje llamado factor de ensamblaje de succinato deshidrogenasa 4 (SDHAF4; llamado Sdh8 en levadura) antes de insertarse en el complejo final. [7]
Los grupos protésicos Fe-S de la subunidad SDHB se están formando en la matriz mitocondrial mediante el complejo proteico ISU. También se cree que el complejo es capaz de insertar los grupos de hierro y azufre en SDHB durante su maduración. Los estudios sugieren que la inserción del grupo Fe-S precede a la formación del dímero SDHA-SDHB. Tal incorporación requiere la reducción de los residuos de cisteína dentro del sitio activo de SDHB. Tanto los residuos de cisteína reducidos como los grupos de Fe-S ya incorporados son altamente susceptibles al daño de ROS . Dos factores de ensamblaje de SDH más, SDHAF1 (Sdh6) y SDHAF3 (Sdh7 en levadura), parecen estar involucrados en la maduración de SDHB en una forma de proteger la subunidad o dímero SDHA-SDHB del daño del grupo Fe-S causado por ROS. [7]
El ensamblaje del ancla hidrofóbica que consta de las subunidades SDHC y SDHD aún no está claro. Especialmente en caso de inserción de hemo b e incluso de su función. El grupo protésico hemo b no parece ser parte de la vía de transporte de electrones dentro del complejo II. [5] El cofactor más bien mantiene la estabilidad del anclaje.
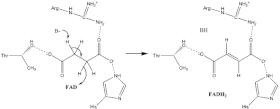
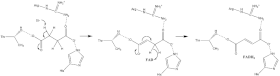
Se sabe mucho sobre el mecanismo de oxidación del succinato , que implica la transferencia de un protón y un hidruro. Una combinación de mutagénesis y análisis estructural identifica a Arg-286 de la subunidad SDHA ( numeración de E. coli ) como la lanzadera de protones. Las estructuras cristalinas de las enzimas de múltiples organismos muestran que están bien preparadas para el paso de transferencia de protones. A partir de entonces, existen dos posibles mecanismos de eliminación: E2 o E1cb. En la eliminación de E2, el mecanismo es concertado. El residuo básico o cofactor desprotona el carbono alfa y FAD acepta el hidruro del carbono beta , oxidando el succinato unido a fumarato ; consulte la imagen 6. En E1cb, se forma un enolato intermedio, como se muestra en la imagen 7, antes de que FAD acepte el carbono alfa. hidruro . Se requieren más investigaciones para determinar qué mecanismo de eliminación sufre el succinato en la succinato deshidrogenasa. El fumarato oxidado , ahora débilmente unido al sitio activo , puede salir libremente de la proteína .
Después de que los electrones se derivan de la oxidación del succinato a través de FAD , hacen un túnel a lo largo del relé [Fe-S] hasta llegar al grupo [3Fe-4S]. Estos electrones se transfieren posteriormente a una molécula de ubiquinona en espera dentro del sitio activo . El sistema de túnel de electrones Hierro - Azufre se muestra en la imagen 9.


El oxígeno carbonilo O1 de la ubiquinona se orienta en el sitio activo (imagen 4) mediante interacciones de enlaces de hidrógeno con Tyr83 de la subunidad D. La presencia de electrones en el grupo de hierro y azufre [3Fe-4S] induce el movimiento de la ubiquinona hacia una segunda orientación. Esto facilita una segunda interacción de enlace de hidrógeno entre el grupo carbonilo O4 de la ubiquinona y Ser27 de la subunidad C. Después del primer paso de reducción de un solo electrón , se forma una especie de radical semiquinona . El segundo electrón llega del grupo [3Fe-4S] para proporcionar la reducción completa de la ubiquinona a ubiquinol . Este mecanismo de reducción de ubiquinona se muestra en la imagen 8.
Aunque todavía se está investigando la funcionalidad del hemo en la succinato deshidrogenasa, algunos estudios [ ¿por quién? ] han afirmado que el primer electrón entregado a la ubiquinona a través de [3Fe-4S] puede hacer un túnel de ida y vuelta entre el hemo y el intermediario de ubiquinona . De esta forma, el cofactor hemo actúa como sumidero de electrones . Su función es prevenir la interacción del intermediario con el oxígeno molecular para producir especies reactivas de oxígeno (ROS). El grupo hemo , en relación con la ubiquinona , se muestra en la imagen 4.
También se ha propuesto que puede existir un mecanismo de activación para evitar que los electrones hagan un túnel directamente al hemo desde el grupo [3Fe-4S]. Un candidato potencial es el residuo His207, que se encuentra directamente entre el grupo y el hemo . His207 de la subunidad B está en proximidad directa del grupo [3Fe-4S], la ubiquinona unida y el hemo ; y podría modular el flujo de electrones entre estos centros redox. [12]
Para reducir completamente la quinona en SQR, se necesitan dos electrones y dos protones . Se ha argumentado que una molécula de agua (HOH39) llega al sitio activo y está coordinada por His207 de la subunidad B, Arg31 de la subunidad C y Asp82 de la subunidad D. La especie de semiquinona es protonada por protones liberados desde HOH39, completando la ubiquinona. reducción a ubiquinol . Lo más probable es que His207 y Asp82 faciliten este proceso. Otros estudios afirman que Tyr83 de la subunidad D está coordinado con una histidina cercana , así como con el oxígeno carbonilo O1 de la ubiquinona . El residuo de histidina disminuye el pKa de la tirosina , haciéndola más adecuada para donar su protón al intermedio reducido de ubiquinona .
Hay dos clases distintas de inhibidores (SDHI) del complejo II: los que se unen en la bolsa de succinato y los que se unen en la bolsa de ubiquinona. Los inhibidores de tipo ubiquinona incluyen carboxina y tenoiltrifluoroacetona . Los inhibidores de análogos de succinato incluyen el compuesto sintético malonato , así como los intermedios del ciclo del TCA, malato y oxaloacetato . De hecho, el oxaloacetato es uno de los inhibidores más potentes del Complejo II. No se comprende del todo por qué un intermediario común del ciclo del TCA inhibiría el Complejo II, aunque puede ejercer un papel protector al minimizar la producción de superóxido mediada por transferencia de electrones inversos por parte del Complejo I. [13] La atpenina 5a es un inhibidor muy potente del Complejo II que imita la unión de ubiquinona .
Los inhibidores de tipo ubiquinona se han utilizado como fungicidas en la agricultura desde los años 1960. La carboxina se utilizó principalmente para controlar enfermedades causadas por basidiomicetos como la roya del tallo y las enfermedades por Rhizoctonia . En la década de 1980 se descubrió que las benzanilides simples tenían una actividad comparable a la carboxina y se comercializaron varias de ellas, entre ellas benodanil , flutolanil y mepronil . [14] Más recientemente, se han desarrollado otros compuestos con un espectro más amplio contra una variedad de patógenos vegetales, incluidos boscalid , fluopiram , fluxapiroxad , pydiflumetofeno y sedaxano . [15] [14] Algunos hongos importantes desde el punto de vista agrícola no son sensibles a los miembros de la nueva generación de inhibidores de tipo ubiquinona. [dieciséis]
FRAC tiene un grupo de trabajo [17] para SDHI y recomienda prácticas de manejo de resistencia . [18]
El papel fundamental de la succinato-coenzima Q reductasa en la cadena de transferencia de electrones de las mitocondrias la hace vital en la mayoría de los organismos multicelulares ; también se ha demostrado que la eliminación de esta enzima del genoma es letal en la etapa embrionaria en ratones.
Todas las mutaciones de SDHx pueden provocar tumorogénesis en células cromafines , provocando tumores neuroendocrinos como paraganglioma , feocromocitoma , carcinoma renal y tumor del estroma gastrointestinal (GIST). [19] SDHA, SDHB, SDHD y SDHAF1 pueden causar deficiencia del complejo mitocondrial II que puede provocar síndrome de Leigh , encefalopatía mitocondrial y atrofia óptica . Las mutaciones SDHB son las más penetrantes en paragangliomas y feocromocitos [20] y tanto las mutaciones SDHD como SDHAF2 tienen algunos efectos de impronta materna. [21]
La succinato deshidrogenasa de mamíferos funciona no solo en la generación de energía mitocondrial , sino que también desempeña un papel en la detección de oxígeno y la supresión de tumores ; y, por tanto, es objeto de investigación continua.
Se observan niveles reducidos de la enzima mitocondrial succinato deshidrogenasa (SDH), el elemento principal del complejo II, post mortem en el cerebro de pacientes con enfermedad de Huntington, y se han identificado defectos en el metabolismo energético en pacientes con EH tanto presintomáticos como sintomáticos. [22]