La visualización de fagos es una técnica de laboratorio para el estudio de las interacciones proteína-proteína , proteína - péptido y proteína- ADN que utiliza bacteriófagos ( virus que infectan bacterias ) para conectar las proteínas con la información genética que las codifica . [1] En esta técnica, un gen que codifica una proteína de interés se inserta en un gen de proteína de cubierta de fago , lo que hace que el fago "muestre" la proteína en su exterior mientras contiene el gen de la proteína en su interior, lo que resulta en una conexión entre genotipo y fenotipo . Las proteínas que muestran los fagos pueden luego analizarse frente a otras proteínas, péptidos o secuencias de ADN, para detectar la interacción entre la proteína mostrada y las de otras moléculas. De esta manera, se pueden analizar y amplificar grandes bibliotecas de proteínas en un proceso llamado selección in vitro , que es análogo a la selección natural .
Los bacteriófagos más comunes utilizados en la presentación de fagos son el fago filamentoso M13 y fd , [2] [3] aunque también se han utilizado los fagos T4 , [4] T7 y λ .
La visualización de fagos fue descrita por primera vez por George P. Smith en 1985, cuando demostró la visualización de péptidos en fagos filamentosos (virus largos y delgados que infectan bacterias) fusionando la proteína de la cápside del virus a un péptido de una colección de secuencias de péptidos. [1] Esto mostró los diferentes péptidos en las superficies externas de la colección de clones virales, donde el paso de selección del proceso aisló los péptidos con la mayor afinidad de unión. En 1988, Stephen Parmley y George Smith describieron la biopanning para la selección por afinidad y demostraron que las rondas recursivas de selección podían enriquecer los clones presentes en una proporción de 1 en mil millones o menos. [5] En 1990, Jamie Scott y George Smith describieron la creación de grandes bibliotecas de péptidos aleatorios que se muestran en fagos filamentosos. [6] La tecnología de visualización de fagos fue desarrollada y mejorada por grupos del Laboratorio de Biología Molecular con Greg Winter y John McCafferty , el Instituto de Investigación Scripps con Richard Lerner y Carlos Barbas y el Centro Alemán de Investigación del Cáncer con Frank Breitling y Stefan Dübel para la visualización de proteínas como anticuerpos para la ingeniería de proteínas terapéuticas . Smith y Winter recibieron la mitad del Premio Nobel de Química de 2018 por su contribución al desarrollo de la visualización de fagos. [7] Una patente de George Pieczenik que reclama prioridad desde 1985 también describe la generación de bibliotecas de péptidos. [8]
Al igual que el sistema de dos híbridos , la visualización de fagos se utiliza para la detección de alto rendimiento de interacciones de proteínas. En el caso de la visualización de fagos filamentosos M13 , el ADN que codifica la proteína o el péptido de interés se liga al gen pIII o pVIII, que codifica la proteína de la capa menor o mayor , respectivamente. A veces se utilizan sitios de clonación múltiples para garantizar que los fragmentos se inserten en los tres marcos de lectura posibles para que el fragmento de ADNc se traduzca en el marco adecuado. A continuación, el híbrido de gen de fago y ADN insertado se inserta (un proceso conocido como " transducción ") en células bacterianas de E. coli como TG1, SS320, ER2738 o XL1-Blue E. coli . Si se utiliza un vector " fagémido " (un vector de construcción de visualización simplificado), las partículas de fago no se liberarán de las células de E. coli hasta que se infecten con fagos auxiliares , lo que permite el empaquetamiento del ADN del fago y el ensamblaje de los viriones maduros con el fragmento de proteína relevante como parte de su capa externa en la proteína de la capa menor (pIII) o mayor (pVIII). Al inmovilizar un objetivo de ADN o proteína relevante en la superficie de un pocillo de placa de microtitulación , permanecerá un fago que muestre una proteína que se une a uno de esos objetivos en su superficie, mientras que los demás se eliminarán mediante lavado. Los que permanezcan se pueden eluir , utilizar para producir más fagos (por infección bacteriana con fagos auxiliares) y para producir una mezcla de fagos que esté enriquecida con fagos relevantes (es decir, de unión). El ciclo repetido de estos pasos se conoce como "panning" , en referencia al enriquecimiento de una muestra de oro mediante la eliminación de materiales indeseables. El fago eluido en el paso final se puede utilizar para infectar un huésped bacteriano adecuado, del cual se pueden recolectar los fagémidos y extraer y secuenciar la secuencia de ADN relevante para identificar las proteínas o fragmentos de proteínas relevantes que interactúan. [ cita requerida ]
El uso de un fago auxiliar se puede eliminar mediante el uso de la tecnología de "línea celular de empaquetamiento bacteriano". [9]
La elución se puede realizar combinando un tampón de elución de bajo pH con sonificación, que, además de aflojar la interacción péptido-diana, también sirve para separar la molécula diana de la superficie de inmovilización. Este método basado en ultrasonidos permite la selección en un solo paso de un péptido de alta afinidad. [10]
Las aplicaciones de la tecnología de visualización de fagos incluyen la determinación de los socios de interacción de una proteína (que se usaría como el "cebo" del fago inmovilizado con una biblioteca de ADN que consiste en todas las secuencias codificantes de una célula, tejido u organismo) de modo que se pueda determinar la función o el mecanismo de la función de esa proteína. [11] La visualización de fagos también es un método ampliamente utilizado para la evolución de proteínas in vitro (también llamada ingeniería de proteínas ). Como tal, la visualización de fagos es una herramienta útil en el descubrimiento de fármacos . Se utiliza para encontrar nuevos ligandos (inhibidores de enzimas, agonistas y antagonistas de receptores) para las proteínas diana. [12] [13] [14] La técnica también se utiliza para determinar antígenos tumorales (para su uso en el diagnóstico y la orientación terapéutica) [15] y en la búsqueda de interacciones proteína-ADN [16] utilizando bibliotecas de ADN especialmente construidas con segmentos aleatorios. Recientemente, la visualización de fagos también se ha utilizado en el contexto de los tratamientos contra el cáncer, como el enfoque de transferencia celular adoptiva. [17] En estos casos, se utiliza la visualización de fagos para crear y seleccionar anticuerpos sintéticos que se dirigen a las proteínas de la superficie del tumor. [17] Estos se convierten en receptores sintéticos para células T recolectadas del paciente que se utilizan para combatir la enfermedad. [18]
Los métodos competitivos para la evolución de proteínas in vitro incluyen la visualización en levaduras , la visualización en bacterias , la visualización en ribosomas y la visualización en ARNm . [ cita requerida ]
La invención de la visualización de anticuerpos en fagos revolucionó el descubrimiento de fármacos basados en anticuerpos. El trabajo inicial fue realizado por laboratorios del Laboratorio de Biología Molecular del MRC ( Greg Winter y John McCafferty ), el Instituto de Investigación Scripps (Richard Lerner y Carlos F. Barbas) y el Centro Alemán de Investigación del Cáncer (Frank Breitling y Stefan Dübel). [19] [20] [21] En 1991, el grupo Scripps informó sobre la primera visualización y selección de anticuerpos humanos en fagos. [22] Este estudio inicial describió el aislamiento rápido del anticuerpo humano Fab que se unía a la toxina del tétanos y el método se amplió luego para clonar rápidamente anticuerpos humanos anti-VIH-1 para el diseño y la terapia de vacunas. [23] [24] [25] [26] [27]
La visualización de fagos de bibliotecas de anticuerpos se ha convertido en un método poderoso tanto para estudiar la respuesta inmune como para seleccionar y desarrollar rápidamente anticuerpos humanos para terapia. La visualización de fagos de anticuerpos fue utilizada posteriormente por Carlos F. Barbas en el Instituto de Investigación Scripps para crear bibliotecas de anticuerpos humanos sintéticos, un principio patentado por primera vez en 1990 por Breitling y colaboradores (Patente CA 2035384), lo que permitió crear anticuerpos humanos in vitro a partir de elementos de diversidad sintéticos. [28] [29] [30] [31]
Las bibliotecas de anticuerpos que muestran millones de anticuerpos diferentes en fagos se utilizan a menudo en la industria farmacéutica para aislar anticuerpos terapéuticos altamente específicos, para su desarrollo en fármacos de anticuerpos principalmente como terapias contra el cáncer o antiinflamatorias. Uno de los más exitosos fue el adalimumab , descubierto por Cambridge Antibody Technology como D2E7 y desarrollado y comercializado por Abbott Laboratories . El adalimumab, un anticuerpo contra el TNF alfa , fue el primer anticuerpo completamente humano del mundo [32] en alcanzar ventas anuales superiores a los mil millones de dólares. [33]
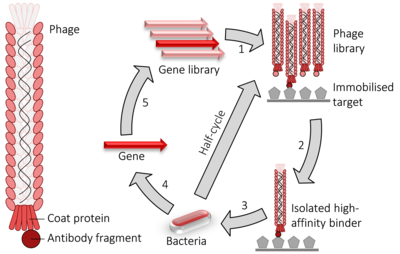
A continuación se muestra la secuencia de eventos que se siguen en la detección mediante visualización de fagos para identificar polipéptidos que se unen con alta afinidad a la proteína objetivo o secuencia de ADN deseada: [ cita requerida ]
pIII es la proteína que determina la infectividad del virión. pIII está compuesta por tres dominios (N1, N2 y CT) conectados por enlaces ricos en glicina. [34] El dominio N2 se une al pilus F durante la infección del virión liberando el dominio N1 que luego interactúa con una proteína TolA en la superficie de la bacteria. [34] Las inserciones dentro de esta proteína generalmente se agregan en la posición 249 (dentro de una región de enlace entre CT y N2), la posición 198 (dentro del dominio N2) y en el extremo N (insertado entre la secuencia de secreción N-terminal y el extremo N-terminal de pIII). [34] Sin embargo, cuando se utiliza el sitio BamHI ubicado en la posición 198, se debe tener cuidado con el residuo de cisteína desapareado (C201) que podría causar problemas durante la visualización del fago si se utiliza una versión no truncada de pIII. [34]
Una ventaja de utilizar pIII en lugar de pVIII es que pIII permite la expresión monovalente cuando se utiliza un fagémido (plásmido derivado de fagos Ff ) combinado con un fago auxiliar. Además, pIII permite la inserción de secuencias proteicas más grandes (>100 aminoácidos) [35] y es más tolerante a él que pVIII. Sin embargo, el uso de pIII como socio de fusión puede conducir a una disminución de la infectividad del fago que conduce a problemas como el sesgo de selección causado por la diferencia en la tasa de crecimiento del fago [36] o peor aún, la incapacidad del fago de infectar a su huésped. [34] La pérdida de la infectividad del fago se puede evitar utilizando un plásmido fagémido y un fago auxiliar de modo que el fago resultante contenga tanto pIII de tipo salvaje como de fusión. [34]
El ADNc también se ha analizado utilizando pIII a través de un sistema de dos cremalleras de leucina complementarias, [37] Direct Interaction Rescue [38] o añadiendo un enlazador de 8-10 aminoácidos entre el ADNc y pIII en el extremo C. [39]
pVIII es la proteína de cubierta principal de los fagos Ff. Los péptidos se fusionan generalmente al extremo N de pVIII. [34] Por lo general, los péptidos que se pueden fusionar a pVIII tienen una longitud de 6 a 8 aminoácidos. [34] La restricción de tamaño parece tener menos que ver con el impedimento estructural causado por la sección agregada [40] y más con la exclusión de tamaño causada por pIV durante la exportación de la proteína de cubierta. [40] Dado que hay alrededor de 2700 copias de la proteína en un fago típico, es más probable que la proteína de interés se exprese de forma polivalente incluso si se utiliza un fagémido. [34] Esto hace que el uso de esta proteína sea desfavorable para el descubrimiento de socios de unión de alta afinidad. [34]
Para superar el problema de tamaño de pVIII, se han diseñado proteínas de cubierta artificiales. [41] Un ejemplo es la proteína de cubierta artificial invertida (ACP) de Weiss y Sidhu, que permite la visualización de proteínas grandes en el extremo C. [41] Las ACP podrían mostrar una proteína de 20 kDa, sin embargo, solo en niveles bajos (en su mayoría solo de forma monovalente). [41]
pVI se ha utilizado ampliamente para la visualización de bibliotecas de ADNc. [34] La visualización de bibliotecas de ADNc mediante visualización de fagos es una alternativa atractiva al método de levadura-2-híbrido para el descubrimiento de proteínas y péptidos interactuantes debido a su alta capacidad de rendimiento. [34] pVI se ha utilizado preferentemente a pVIII y pIII para la expresión de bibliotecas de ADNc porque se puede agregar la proteína de interés al extremo C de pVI sin afectar en gran medida el papel de pVI en el ensamblaje de fagos. Esto significa que el codón de parada en el ADNc ya no es un problema. [42] Sin embargo, la visualización de ADNc mediante fagos siempre está limitada por la incapacidad de la mayoría de los procariotas de producir modificaciones postraduccionales presentes en las células eucariotas o por el plegamiento incorrecto de proteínas multidominio.
Si bien pVI ha sido útil para el análisis de bibliotecas de ADNc, pIII y pVIII siguen siendo las proteínas de cubierta más utilizadas para la presentación de fagos. [34]
En un experimento realizado en 1995, se intentó la visualización de la glutatión S-transferasa tanto en pVII como en pIX y fracasó. [43] Sin embargo, la visualización de esta proteína en fagos se completó con éxito después de la adición de una secuencia de señal periplásmica (pelB o ompA) en el extremo N. [44] En un estudio reciente, se ha demostrado que AviTag, FLAG e His se podían mostrar en pVII sin la necesidad de una secuencia de señal. Luego, la expresión de Fv de cadena sencilla (scFv) y receptores de células T de cadena sencilla (scTCR) se expresaron tanto con como sin la secuencia de señal. [45]
PelB (una secuencia de señal de aminoácidos que dirige la proteína al periplasma, donde una peptidasa señal escinde PelB) mejoró el nivel de presentación de fagos en comparación con las fusiones pVII y pIX sin la secuencia de señal. Sin embargo, esto condujo a la incorporación de más genomas de fagos auxiliares en lugar de genomas de fagémidos. En todos los casos, los niveles de presentación de fagos fueron inferiores a los obtenidos con la fusión pIII. Sin embargo, una presentación inferior podría ser más favorable para la selección de ligantes debido a que una presentación inferior está más cerca de la presentación monovalente verdadera. En cinco de seis ocasiones, las fusiones pVII y pIX sin pelB fueron más eficientes que las fusiones pIII en los ensayos de selección por afinidad. El artículo incluso continúa afirmando que las plataformas de presentación pVII y pIX pueden superar a pIII a largo plazo. [45]
El uso de pVII y pIX en lugar de pIII también podría ser una ventaja porque el rescate del virión puede llevarse a cabo sin romper el enlace virión-antígeno si el pIII utilizado es de tipo salvaje. En cambio, se podría cortar en una sección entre la perla y el antígeno para eluir. Dado que el pIII está intacto, no importa si el antígeno permanece unido al fago. [45]
El problema de utilizar fagos Ff para la visualización de fagos es que requieren que la proteína de interés se transloque a través de la membrana interna bacteriana antes de que se ensamble en el fago. [46] Algunas proteínas no pueden experimentar este proceso y, por lo tanto, no se pueden mostrar en la superficie de los fagos Ff. En estos casos, se utiliza en su lugar la visualización de fagos T7. [46] En la visualización de fagos T7, la proteína que se va a mostrar se une al extremo C de la proteína de la cápside del gen 10 de T7. [46]
La desventaja de utilizar T7 es que el tamaño de la proteína que se puede expresar en la superficie está limitado a péptidos más cortos porque no se pueden acomodar grandes cambios en el genoma de T7 como ocurre en M13, donde el fago simplemente alarga su capa para que quepa el genoma más grande dentro de ella. Sin embargo, puede ser útil para la producción de una gran biblioteca de proteínas para la selección de scFV, donde el scFV se expresa en un fago M13 y los antígenos se expresan en la superficie del fago T7. [47]
Las bases de datos y las herramientas computacionales para mimotopos han sido una parte importante del estudio de visualización de fagos. [48] Las bases de datos, [49] programas y servidores web [50] se han utilizado ampliamente para excluir péptidos no relacionados con el objetivo, [51] caracterizar interacciones proteína-moléculas pequeñas y mapear interacciones proteína-proteína. Los usuarios pueden usar la estructura tridimensional de una proteína y los péptidos seleccionados del experimento de visualización de fagos para mapear epítopos conformacionales. Algunos de los métodos computacionales rápidos y eficientes están disponibles en línea. [50]
Técnicas competitivas: