La teoría del gen dividido es una teoría del origen de los intrones , secuencias largas no codificantes en genes eucariotas entre los exones . [1] [2] [3] La teoría sostiene que la aleatoriedad de las secuencias de ADN primordiales solo permitiría marcos de lectura abiertos (ORF) pequeños (<600 pb), y que las estructuras de intrones y las secuencias reguladoras importantes se derivan de codones de terminación . En este marco de intrones primero, la maquinaria espliceosómica y el núcleo evolucionaron debido a la necesidad de unir estos ORF (ahora "exones") en proteínas más grandes, y que los genes bacterianos sin intrones son menos ancestrales que los genes eucariotas divididos. La teoría se originó con Periannan Senapathy .
La teoría proporciona soluciones a cuestiones clave relacionadas con la arquitectura de los genes escindidos, incluidos los genes eucariotas escindidos, los exones, los intrones, las uniones de empalme y los puntos de ramificación, basándose en el origen de los genes escindidos a partir de secuencias genéticas aleatorias. También proporciona posibles soluciones al origen de la maquinaria espliceosómica, el límite nuclear y la célula eucariota.
Esta teoría condujo al algoritmo Shapiro-Senapathy , que proporciona la metodología para detectar los sitios de empalme, exones y genes divididos en el ADN eucariota, y que es el método principal para detectar mutaciones en el sitio de empalme en genes que causan cientos de enfermedades.
La teoría de los genes divididos requiere un origen separado de todas las especies eucariotas. También requiere que los procariotas más simples evolucionen a partir de eucariotas. Esto contradice completamente el consenso científico sobre la formación de células eucariotas por endosimbiosis de bacterias. En 1994, Senapathy escribió un libro sobre este aspecto de su teoría: El nacimiento independiente de los organismos. Proponía que todos los genomas eucariotas se formaron por separado en un grupo primordial. El biólogo holandés Gert Korthoff criticó la teoría planteando varios problemas que no pueden explicarse mediante una teoría de orígenes independientes. Señaló que varios eucariotas necesitan cuidados y lo llamó el "problema de la bota", ya que incluso el eucariota inicial necesitaba el cuidado de los padres. Korthoff señala que una gran fracción de eucariotas son parásitos. La teoría de Senapathy requeriría una coincidencia para explicar su existencia. [4] [5] La teoría de Senapathy no puede explicar la fuerte evidencia de descendencia común ( homología , código genético universal, embriología , registro fósil ). [6]

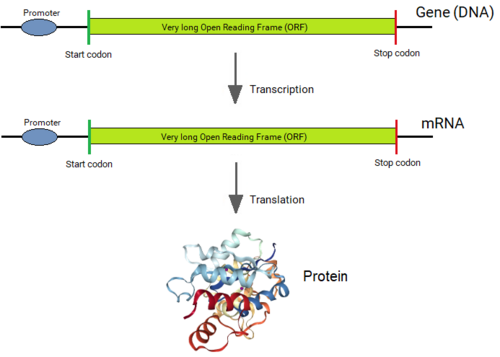
Los genes de todos los organismos, excepto las bacterias, consisten en regiones cortas codificantes de proteínas ( exones ) interrumpidas por secuencias largas ( intrones ). [1] [2] Cuando se expresa un gen, su secuencia de ADN se copia en una secuencia de "ARN primario" por la enzima ARN polimerasa . Luego, la maquinaria del "spliceosoma" elimina físicamente los intrones de la copia de ARN del gen mediante el proceso de empalme, dejando solo una serie de exones conectados contiguamente, que se convierte en ARN mensajero (ARNm). Este ARNm ahora es leído por el ribosoma , que produce la proteína codificada. Por lo tanto, aunque los intrones no se eliminan físicamente de un gen, la secuencia de un gen se lee como si los intrones no estuvieran presentes.
Los exones suelen ser cortos, con una longitud media de unas 120 bases (por ejemplo, en los genes humanos). Las longitudes de los intrones varían ampliamente, de 10 a 500.000, pero las longitudes de los exones tienen un límite superior de unas 600 bases en la mayoría de los eucariotas. Debido a que los exones codifican secuencias de proteínas, son importantes para la célula, pero constituyen solo el ~2% de las secuencias. Los intrones, en cambio, constituyen el 98% de las secuencias, pero parecen tener pocas funciones cruciales, excepto secuencias potenciadoras y reguladoras del desarrollo en casos raros. [7] [8]
Hasta que Philip Sharp [9] [10] y Richard Roberts [11] descubrieron los intrones [12] en los genes eucariotas en 1977, se creía que la secuencia codificante de todos los genes se encontraba siempre en un único tramo, delimitado por un único y largo ORF. El descubrimiento de los intrones fue una profunda sorpresa, que inmediatamente planteó las preguntas de cómo, por qué y cuándo los intrones entraron en los genes eucariotas.
Pronto se hizo evidente que un gen eucariota típico estaba interrumpido en muchos lugares por intrones, lo que dividía la secuencia codificante en muchos exones cortos. También fue sorprendente que los intrones fueran largos, de cientos de miles de bases. Estos hallazgos dieron lugar a la pregunta de por qué hay muchos intrones dentro de un gen (por ejemplo, hay ~312 intrones en el gen humano TTN), por qué son largos y por qué los exones son cortos.
También se descubrió que la maquinaria del espliceosoma era grande y compleja, con unas 300 proteínas y varias moléculas de ARNmSn . Las preguntas se extendieron al origen del espliceosoma. Poco después del descubrimiento de los intrones, se hizo evidente que las uniones entre exones e intrones a ambos lados exhibían secuencias específicas que dirigían la maquinaria del espliceosoma a la posición de base exacta para el empalme. Cómo y por qué surgieron estas señales de unión de empalme fue otra pregunta importante.
El descubrimiento de los intrones y la arquitectura génica fragmentada de los genes eucariotas dio inicio a una nueva era en la biología eucariota. La pregunta de por qué los genes eucariotas tenían genes fragmentados provocó especulaciones y debates casi de inmediato.
En 1978, Ford Doolittle publicó un artículo en el que afirmaba que la mayoría de los biólogos moleculares asumían que el genoma eucariota surgió de un genoma procariota «más simple» y «primitivo», muy parecido al de Escherichia coli . [13] Sin embargo, este tipo de evolución requeriría que se introdujeran intrones en las secuencias codificantes de los genes bacterianos. Con respecto a este requisito, Doolittle dijo: «Es extraordinariamente difícil imaginar cómo se podrían introducir secuencias irrelevantes desde el punto de vista informativo en genes estructurales preexistentes sin efectos nocivos». Afirmó: «Me gustaría argumentar que el genoma eucariota, al menos en ese aspecto de su estructura que se manifiesta como «genes en pedazos», es de hecho la forma original primitiva».
James Darnell expresó puntos de vista similares en 1978. Afirmó: “Las diferencias en la bioquímica de la formación del ARN mensajero en eucariotas en comparación con procariotas son tan profundas que sugieren que la evolución secuencial de células procariotas a eucariotas parece improbable. Las secuencias no contiguas descubiertas recientemente en el ADN eucariota que codifican el ARN mensajero pueden reflejar una distribución antigua, en lugar de una nueva, de la información en el ADN y que los eucariotas evolucionaron independientemente de los procariotas”. [14]
Sin embargo, en un aparente intento de reconciliarse con la idea de que el ARN precedió al ADN en la evolución, y con el concepto de los tres linajes evolutivos de archea , bacteria y eukarya, tanto Doolittle como Darnell se desviaron de su especulación original en un artículo conjunto en 1985. [15] Sugirieron que el ancestro de los tres grupos de organismos, el " progenote ", tenía una estructura de genes en pedazos, de la cual evolucionaron los tres linajes. Especularon que la etapa precelular tenía genes de ARN primitivos que tenían intrones, que se transcribieron de manera inversa en ADN y formaron el progenote. Las bacterias y archea evolucionaron a partir del progenote al perder intrones, y el "urcariota" evolucionó a partir de él al retener intrones. Más tarde, el eucariota evolucionó a partir del urcariota al desarrollar un núcleo y absorber mitocondrias de las bacterias. Luego, los organismos multicelulares evolucionaron a partir del eucariota.
Estos autores predijeron que las distinciones entre procariotas y eucariotas eran tan profundas que la evolución de procariotas a eucariotas no era sostenible y tenía orígenes diferentes. Sin embargo, aparte de las especulaciones de que los genes de ARN precelulares debían tener intrones, no abordaron las cuestiones clave del origen de los intrones. No explicaron por qué los exones eran cortos y los intrones largos, cómo se originaron las uniones de empalme, qué significaban la estructura y la secuencia de las uniones de empalme y por qué los genomas eucariotas eran grandes.
Casi al mismo tiempo que Doolittle y Darnell sugirieron que los intrones en los genes eucariotas podían ser antiguos, Colin Blake [16] y Walter Gilbert [17] [18] publicaron sus puntos de vista sobre los orígenes de los intrones de forma independiente. En su opinión, los intrones se originaron como secuencias espaciadoras que permitieron la recombinación y la reorganización conveniente de exones que codificaban dominios funcionales distintos para desarrollar nuevos genes. De este modo, los nuevos genes se ensamblaban a partir de módulos de exones que codificaban dominios funcionales, regiones de plegamiento o elementos estructurales de genes preexistentes en el genoma de un organismo ancestral, evolucionando así genes con nuevas funciones. No especificaron cómo se originaron los exones o los intrones. Además, incluso después de muchos años, un análisis exhaustivo de miles de proteínas y genes mostró que solo en muy raras ocasiones los genes exhiben el supuesto fenómeno de reorganización de exones. [19] [20] Además, los biólogos moleculares cuestionaron la propuesta de reordenamiento de exones, desde un punto de vista puramente evolutivo, por razones tanto metodológicas como conceptuales, y, a largo plazo, esta teoría no sobrevivió.
En la época en que se descubrieron los intrones, Senapathy se preguntaba cómo podrían haberse originado los genes. Supuso que para que cualquier gen se formara, las secuencias genéticas (ARN o ADN) debían haber estado presentes en el entorno prebiótico. Una pregunta básica que se planteó fue cómo las secuencias codificadoras de proteínas podrían haberse originado a partir de secuencias de ADN primordiales en el origen de las primeras células.
Para responder a esto, hizo dos suposiciones básicas:
También supuso que los codones debían haberse establecido antes del origen de los primeros genes. Si el ADN primordial contenía secuencias de nucleótidos aleatorias, se preguntó: ¿existía un límite superior en la longitud de las secuencias codificantes y, de ser así, este límite desempeñaba un papel crucial en la formación de las características estructurales de los genes en el origen de los mismos?
Su lógica era la siguiente: la longitud media de las proteínas en los organismos vivos, incluidos los organismos eucariotas y bacterianos, era de unos 400 aminoácidos. Sin embargo, existían proteínas mucho más largas, incluso más largas que 10.000-30.000 aminoácidos tanto en eucariotas como en bacterias. [21] Por tanto, la secuencia codificante de miles de bases existía en un único tramo en los genes bacterianos. Por el contrario, la secuencia codificante de los eucariotas existía solo en segmentos cortos de exones de unas 120 bases, independientemente de la longitud de la proteína. Si las longitudes de los ORF de la secuencia codificante en secuencias de ADN aleatorias eran tan largas como las de los organismos bacterianos, entonces eran posibles genes codificantes largos y contiguos en el ADN aleatorio. Esto no se sabía, ya que nunca se había estudiado la distribución de las longitudes de los ORF en una secuencia de ADN aleatoria.
Como las secuencias de ADN aleatorias se podían generar en la computadora, Senapathy pensó que podría plantear estas preguntas y realizar sus experimentos in silico . Además, cuando comenzó a estudiar esta cuestión, ya existía suficiente información sobre secuencias de ADN y proteínas en la base de datos de la National Biomedical Research Foundation (NBRF) a principios de los años 1980.

Senapathy analizó primero la distribución de las longitudes de los ORF en secuencias de ADN aleatorias generadas por computadora. Sorprendentemente, este estudio reveló que alrededor de 200 codones (600 bases) era el límite superior en longitudes de ORF. El ORF más corto (cero bases de longitud) era el más frecuente. A medida que aumentaban las longitudes de los ORF, su frecuencia disminuía logarítmicamente, acercándose a cero en aproximadamente 600 bases. Cuando se trazó la probabilidad de longitudes de ORF en una secuencia aleatoria, se reveló que la probabilidad de longitudes crecientes de ORF disminuía exponencialmente y se reducía en un máximo de aproximadamente 600 bases. A partir de esta distribución "exponencial negativa" de longitudes de ORF, se descubrió que la mayoría de los ORF eran mucho más cortos que el máximo.

Este hallazgo fue sorprendente porque la secuencia codificante para la longitud promedio de una proteína de 400 AA (con ~1200 bases de secuencia codificante) y proteínas más largas de miles de AA (que requieren >10 000 bases de secuencia codificante) no ocurriría de golpe en una secuencia aleatoria. Si esto fuera cierto, un gen típico con una secuencia codificante contigua no podría originarse en una secuencia aleatoria. Por lo tanto, la única forma posible de que cualquier gen pudiera originarse a partir de una secuencia aleatoria era dividir la secuencia codificante en segmentos más cortos y seleccionar estos segmentos de los ORF cortos disponibles en la secuencia aleatoria, en lugar de aumentar la longitud del ORF eliminando codones de terminación consecutivos. Este proceso de elegir segmentos cortos de secuencias codificantes de los ORF disponibles para hacer un ORF largo conduciría a una estructura dividida.
Si esta hipótesis fuera cierta, las secuencias de ADN eucariota deberían reflejarla. Cuando Senapathy trazó la distribución de longitudes de ORF en secuencias de ADN eucariota, el gráfico era notablemente similar al de secuencias de ADN aleatorias. Este gráfico también era una distribución exponencial negativa que se reducía a un máximo de aproximadamente 600 bases, como en el caso de los genes eucariotas, [1] [22] [3] que coincidía exactamente con la longitud máxima de ORF observada tanto en secuencias de ADN aleatorias como de ADN eucariota.
Los genes divididos se originaron a partir de secuencias de ADN aleatorias mediante la elección de los mejores segmentos codificantes cortos (exones) y su empalme. Las secuencias de intrones intermedias eran vestigios sobrantes de las secuencias aleatorias y, por lo tanto, estaban destinadas a ser eliminadas por el espliceosoma. Estos hallazgos indicaron que los genes divididos podrían haberse originado a partir de secuencias de ADN aleatorias con exones e intrones tal como aparecen en los organismos eucariotas actuales. El premio Nobel Marshall Nirenberg , que descifró los codones, afirmó que estos hallazgos demostraban firmemente que la teoría de los genes divididos para el origen de los intrones y la estructura dividida de los genes debe ser válida. [1] [23]
Blake propuso la hipótesis de Gilbert-Blake en 1979 para el origen de los intrones y afirmó que la teoría del gen dividido de Senapathy explicaba de manera exhaustiva el origen de la estructura del gen dividido. Además, afirmó que explicaba varias cuestiones clave, incluido el origen del mecanismo de empalme: [16]
Un trabajo reciente de Senapathy, aplicado al ARN, explica de forma exhaustiva el origen de la forma segregada del ARN en regiones codificantes y no codificantes. También sugiere por qué se desarrolló un mecanismo de empalme al comienzo de la evolución primordial. Descubrió que la distribución de las longitudes de los marcos de lectura en una secuencia aleatoria de nucleótidos se correspondía exactamente con la distribución observada de los tamaños de los exones eucariotas. Estos estaban delimitados por regiones que contenían señales de parada, los mensajes para terminar la construcción de la cadena polipeptídica, y eran, por tanto, regiones no codificantes o intrones. Por tanto, la presencia de una secuencia aleatoria fue suficiente para crear en el ancestro primordial la forma segregada del ARN observada en la estructura del gen eucariota. Además, la distribución aleatoria también muestra un límite a los 600 nucleótidos, lo que sugiere que el tamaño máximo de un polipéptido temprano era de 200 residuos, de nuevo como se observa en el tamaño máximo del exón eucariota. Así, en respuesta a las presiones evolutivas para crear genes más grandes y complejos, los fragmentos de ARN se unieron mediante un mecanismo de empalme que eliminó los intrones. Por lo tanto, la existencia temprana tanto de intrones como de empalme de ARN en eucariotas parece ser muy probable a partir de una base estadística simple. Estos resultados también concuerdan con la relación lineal encontrada entre el número de exones en el gen de una proteína particular y la longitud de la cadena polipeptídica”.

Según la teoría de los genes divididos, un exón se define por un ORF. Se requiere un mecanismo para reconocer que un ORF se ha originado. Como un ORF se define por una secuencia codificante contigua delimitada por codones de terminación, estos extremos de codones de terminación tenían que ser reconocidos por el sistema de reconocimiento de genes exón-intrón. Este sistema podría haber definido los exones por la presencia de un codón de terminación en los extremos de los ORF, que debería estar incluido dentro de los extremos de los intrones y eliminado por el proceso de empalme. Por lo tanto, los intrones deberían contener un codón de terminación en sus extremos, que sería parte de las secuencias de unión de empalme.
Si esta hipótesis fuera cierta, los genes divididos de los organismos vivos actuales deberían contener codones de terminación exactamente en los extremos de los intrones. Cuando Senapathy puso a prueba esta hipótesis en las uniones de empalme de genes eucariotas, descubrió que la gran mayoría de las uniones de empalme contenían un codón de terminación al final de cada intrón, fuera de los exones. De hecho, se descubrió que estos codones de terminación formaban la secuencia de empalme GT:AG “canónica”, y los tres codones de terminación se encontraban como parte de las fuertes señales de consenso. Por lo tanto, la teoría básica de los genes divididos para el origen de los intrones y la estructura de los genes divididos condujo a la comprensión de que las uniones de empalme se originaban a partir de los codones de terminación. [2]
Cuando Senapathy pensó en esta cuestión, sólo se disponía de datos de secuencias de unas 1.000 uniones exón-intrón. Tomó los datos de 1.030 secuencias de uniones de empalme (donantes y aceptores) y contó los codones que se producían en cada una de las posiciones de 7 bases de la secuencia de la señal donante [CAG:GTGAGT] y en cada una de las posibles posiciones de 2 bases de la señal aceptora [CAG:G] de la base de datos GenBank. Descubrió que los codones de parada se producían con alta frecuencia sólo en la posición de la 5.ª base de la señal donante y en la primera posición de la base de la señal aceptora. Estas posiciones son el* inicio del intrón (de hecho, una base después del inicio) y el final del intrón, como Senapathy había predicho. Se muestran los recuentos de codones sólo en estas posiciones. Incluso cuando los codones de estas posiciones no eran codones de parada, el 70% de ellos empezaban con las dos primeras bases de los codones de parada TA y TG [TAT = 75; [TAC = 59; TGT = 70].
Los tres codones de terminación (TGA, TAA y TAG) se encontraron después de una base (G) al comienzo de los intrones. Estos codones de terminación se muestran en la unión de empalme donante canónica de consenso como AG:GT(A/G)GGT, donde TAA y TGA son los codones de terminación, y el TAG adicional también está presente en esta posición. Además del codón CAG, solo TAG, que es un codón de terminación, se encontró en los extremos de los intrones. La unión de empalme aceptor canónica se muestra como (C/T)AG:GT, en la que TAG es el codón de terminación. Estas secuencias de consenso muestran claramente la presencia de los codones de terminación en los extremos de los intrones que bordean los exones en todos los genes eucariotas, lo que proporciona una fuerte corroboración de la teoría del gen dividido. Nirenberg afirmó nuevamente que estas observaciones respaldaban completamente la teoría del gen dividido para el origen de las secuencias de unión de empalme a partir de codones de terminación. [2] [24]
Poco después del descubrimiento de los intrones por parte de Philip Sharp y Richard Roberts , se supo que las mutaciones dentro de las uniones de empalme podían provocar enfermedades. Senapathy demostró que las mutaciones en las bases del codón de terminación (bases canónicas) causaban más enfermedades que las mutaciones en bases no canónicas. [1]
Una etapa intermedia en el proceso de empalme del ARN eucariota es la formación de una estructura en forma de lazo. Está anclada en un residuo de adenosina en el intrón entre 10 y 50 nucleótidos aguas arriba del sitio de empalme 3'. Una secuencia corta conservada (la secuencia del punto de ramificación) funciona como la señal de reconocimiento para el sitio de formación del lazo. Durante el proceso de empalme, esta secuencia conservada hacia el final del intrón forma una estructura en forma de lazo con el comienzo del intrón. [25] El paso final del proceso de empalme ocurre cuando los dos exones se unen y el intrón se libera como un ARN en forma de lazo. [26]
Varios investigadores encontraron secuencias de puntos de ramificación en diferentes organismos [25] , entre ellos levaduras, humanos, moscas de la fruta, ratas y plantas. Senapathy descubrió que, en todas estas secuencias, el codón que termina en la adenosina del punto de ramificación es consistentemente un codón de terminación. Lo interesante es que dos de los tres codones de terminación (TAA y TGA) aparecen casi siempre en esta posición.
Estos hallazgos llevaron a Senapathy a proponer que la señal del punto de ramificación se originó a partir de codones de terminación. El hallazgo de que dos codones de terminación diferentes (TAA y TGA) ocurren dentro de la señal del lazo con el punto de ramificación como la tercera base de los codones de terminación corrobora esta propuesta. Como el punto de ramificación del lazo se produce en la última adenina del codón de terminación, es posible que la maquinaria del espliceosoma que se originó para la eliminación de los codones de terminación de la secuencia primaria de ARN creara una señal de secuencia de codón de terminación auxiliar como la secuencia del lazo para ayudar a su función de empalme. [2]
Se cree que el pequeño ARN nuclear U2 que se encuentra en los complejos de empalme ayuda al proceso de empalme al interactuar con la secuencia lariat. [27] Las secuencias complementarias tanto para la secuencia lariat como para la señal aceptora están presentes en un segmento de solo 15 nucleótidos en el ARN U2. Además, se ha propuesto que el ARN U1 funcione como una guía en el proceso de empalme para identificar la unión de empalme donante precisa mediante el apareamiento de bases complementarias. Las regiones conservadas del ARN U1 incluyen, por lo tanto, secuencias complementarias a los codones de terminación. Estas observaciones permitieron a Senapathy predecir que los codones de terminación habían operado en el origen no solo de las señales de unión de empalme y la señal lariat, sino también de algunos ARN nucleares pequeños.
Senapathy propuso que las secuencias reguladoras de la expresión génica (secuencias promotoras y del sitio de adición de poli-A) también podrían haberse originado a partir de codones de terminación. Una secuencia conservada, AATAAA, existe en casi todos los genes a poca distancia del final del mensaje codificador de proteínas y sirve como señal para la adición de poli(A) en la copia de ARNm del gen. [28] Esta señal de secuencia de poli(A) contiene un codón de terminación, TAA. Una secuencia que se encuentra poco después de esta señal, que se cree que forma parte de la señal de poli(A) completa, también contiene los codones de terminación TAG y TGA.
Los promotores eucariotas dependientes de la ARN-polimerasa II pueden contener una caja TATA (secuencia de consenso TATAAA), que contiene el codón de terminación TAA. Los elementos promotores bacterianos en ~10 bases exhiben una caja TATA con un consenso de TATAAT (que contiene el codón de terminación TAA), y en -35 bases exhiben un consenso de TTGACA (que contiene el codón de terminación TGA). Por lo tanto, la evolución de todo el mecanismo de procesamiento del ARN parece haber sido influenciada por la aparición demasiado frecuente de codones de terminación, lo que hace que los codones de terminación sean los puntos focales para el procesamiento del ARN.

Senapathy descubrió que los codones de terminación son partes clave de cada elemento genético en los genes eucariotas. La tabla y la figura muestran que las partes clave de los elementos promotores centrales, la señal de lazo, las señales de empalme del donante y el aceptor y la señal de adición de poli-A consisten en uno o más codones de terminación. Este hallazgo corrobora la afirmación de la teoría del gen dividido de que la razón subyacente para el paradigma del gen dividido completo es el origen de los genes divididos a partir de secuencias de ADN aleatorias, en las que la naturaleza utilizó la distribución aleatoria de una frecuencia extremadamente alta de codones de terminación para definir estos elementos genéticos.
Las investigaciones basadas en la teoría de los genes divididos arrojan luz sobre otras cuestiones básicas de los exones y los intrones. Los exones de los eucariotas son generalmente cortos (los exones humanos tienen una media de ~120 bases, y pueden tener hasta 10 bases) y los intrones suelen ser largos (una media de ~3.000 bases, y pueden tener varios cientos de miles de bases de longitud), por ejemplo los genes RBFOX1, CNTNAP2, PTPRD y DLG2. Senapathy proporcionó una respuesta plausible a estas preguntas, la única explicación hasta la fecha. Si los genes eucariotas se originaron a partir de secuencias de ADN aleatorias, tienen que coincidir con las longitudes de los ORF de secuencias aleatorias, y posiblemente deberían tener alrededor de 100 bases (cerca de la longitud media de los ORF en secuencias aleatorias). Las secuencias del genoma de los organismos vivos exhiben exactamente las mismas longitudes promedio de 120 bases para los exones, y los exones más largos de 600 bases (con pocas excepciones), que es la misma longitud que la de los ORFs aleatorios más largos. [1] [2] [3] [22]
Si los genes divididos se originaran en secuencias de ADN aleatorias, los intrones serían largos por varias razones. Los codones de terminación se encuentran en grupos que dan lugar a numerosos ORF cortos consecutivos: los ORF más largos que podrían definirse como exones serían más raros. Además, los mejores parámetros de secuencia codificante para proteínas funcionales se elegirían de los ORF largos en una secuencia aleatoria, lo que puede ocurrir raramente. Además, la combinación de secuencias de unión de empalme de donantes y aceptores dentro de longitudes cortas de segmentos de secuencia codificante que definirían los límites de los exones ocurriría raramente en una secuencia aleatoria. Estas razones combinadas harían que los intrones fueran largos en comparación con los exones.
Este trabajo también explica por qué genomas como el genoma humano tienen miles de millones de bases, y por qué solo una pequeña fracción (~2%) codifica proteínas y otros elementos reguladores. [29] [30] Si los genes divididos se originaron a partir de secuencias de ADN primordiales aleatorias, contendrían una cantidad significativa de ADN representado por intrones. Además, un genoma ensamblado a partir de ADN aleatorio que contenga genes divididos también incluiría ADN aleatorio intergénico. Por lo tanto, los genomas que se originaron a partir de secuencias de ADN aleatorias tenían que ser grandes, independientemente de la complejidad del organismo.
La observación de que varios organismos como la cebolla (~16 mil millones de bases [31] ) y la salamandra (~32 mil millones de bases [32] ) tienen genomas mucho más grandes que los humanos (~3 mil millones de bases [33] [34] ) mientras que los organismos no son más complejos que los humanos concuerda con la teoría. Además, el hecho de que varios organismos con genomas más pequeños tengan una cantidad similar de genes que los humanos, como C. elegans (tamaño del genoma ~100 millones de bases, ~19.000 genes) [35] y Arabidopsis thaliana (tamaño del genoma ~125 millones de bases, ~25.000 genes), [36] apoya la teoría. La teoría predice que los intrones en los genes divididos en estos genomas podrían ser la forma "reducida" (o eliminada) en comparación con los genes más grandes con intrones largos, lo que conduce a genomas reducidos. [1] [22] De hecho, los investigadores han propuesto recientemente que estos genomas más pequeños son en realidad genomas reducidos. [37]
Senapathy abordó el origen de la maquinaria espliceosómica que elimina los intrones de las transcripciones de ARN. Si los genes divididos se hubieran originado a partir de ADN aleatorio, entonces los intrones se habrían convertido en una parte innecesaria pero integral de los genes eucariotas junto con las uniones de empalme. La maquinaria espliceosómica sería necesaria para eliminarlos y permitir que los exones cortos se empalmen linealmente entre sí como un ARNm codificante contiguo que se puede traducir en una proteína completa. Por lo tanto, la teoría del gen dividido sostiene que la maquinaria espliceosómica existe para eliminar los intrones innecesarios. [1] [2]
Blake afirma: “El trabajo de Senapathy, cuando se aplica al ARN, explica de manera exhaustiva el origen de la forma segregada del ARN en regiones codificantes y no codificantes. También sugiere por qué se desarrolló un mecanismo de empalme al comienzo de la evolución primordial”. [16]
Senapathy propuso una lógica funcional y mecanicista plausible para explicar el origen del núcleo eucariota, una cuestión de gran importancia en biología. [1] [2] Si las transcripciones de los genes divididos y los ARNm empalmados estuvieran presentes en una célula sin núcleo, los ribosomas intentarían unirse tanto al transcrito de ARN primario no empalmado como al ARNm empalmado, lo que provocaría un caos. Un límite que separa el proceso de empalme de ARN de la traducción de ARNm evita este problema. El límite nuclear proporciona una separación clara del empalme de ARN primario y la traducción de ARNm.
Estas investigaciones llevaron a la posibilidad de que el ADN primordial con una secuencia esencialmente aleatoria diera lugar a la estructura compleja de los genes divididos con exones, intrones y uniones de empalme. Las células que albergaban genes divididos tenían que ser complejas con un límite citoplasmático nuclear y debían tener una maquinaria espliceosómica. Por lo tanto, era posible que la célula más antigua fuera compleja y eucariota. [1] [2] [3] [22] Sorprendentemente, los hallazgos de una extensa investigación genómica comparativa de varios organismos desde 2007 muestran abrumadoramente que los primeros organismos podrían haber sido altamente complejos y eucariotas, y podrían haber contenido proteínas complejas, [38] [39] [40] [41] [42] [43] [44] como predijo la teoría de Senapathy.
El espliceosoma es un mecanismo altamente complejo, que contiene ~200 proteínas y varios SnRNP . Collins y Penny afirmaron: “Partimos de la hipótesis de que... el espliceosoma ha aumentado en complejidad a lo largo de la evolución eucariota. Sin embargo, el examen de la distribución de los componentes espliceosomales indica que no solo había un espliceosoma presente en el ancestro eucariota, sino que también contenía la mayoría de los componentes clave que se encuentran en los eucariotas actuales... el último ancestro común de los eucariotas actuales parece mostrar gran parte de la complejidad molecular que se observa hoy en día”. Esto sugiere que los primeros organismos eucariotas eran complejos y contenían genes y proteínas sofisticados. [45]
Era prácticamente imposible que se hubieran producido genes con secuencias codificantes ininterrumpidas de miles de bases de longitud (hasta 90.000 bases) que se encuentran en muchos organismos bacterianos [21] . Sin embargo, los genes bacterianos podrían haberse originado a partir de genes divididos mediante la pérdida de intrones, la única forma propuesta para llegar a secuencias codificantes largas. También es una forma mejor [ aclaración necesaria ] que aumentar la longitud de los ORF de ORF aleatorios cortos a ORF largos mediante la eliminación específica de los codones de terminación mediante mutación. [1] [2] [3]
Según la teoría de los genes divididos, este proceso de pérdida de intrones podría haberse producido a partir de ADN aleatorio prebiótico. Estos genes codificantes contiguos podrían estar organizados de forma compacta en los genomas bacterianos sin ningún intrón y ser más aerodinámicos. Según Senapathy, el límite nuclear que se requiere para una célula que contiene genes divididos no sería necesario para una célula que contiene solo genes ininterrumpidos. Por lo tanto, las células bacterianas no desarrollaron un núcleo. Según la teoría de los genes divididos, los genomas eucariotas y bacterianos podrían haberse originado independientemente a partir de los genes divididos en secuencias de ADN aleatorias primordiales.
Senapathy desarrolló algoritmos para detectar sitios de empalme de donantes y aceptores, exones y un gen dividido completo en una secuencia genómica. Desarrolló el método de la matriz de peso de posición (PWM) basado en la frecuencia de las cuatro bases en las secuencias de consenso del donante y el aceptor en diferentes organismos para identificar los sitios de empalme en una secuencia dada. Además, formuló el primer algoritmo para encontrar los exones basado en el requisito de que los exones contengan una secuencia donante (en el extremo 5') y una secuencia aceptora (en el extremo 3'), y un ORF en el que debe aparecer el exón, y otro algoritmo para encontrar un gen dividido completo. Estos algoritmos se conocen colectivamente como el algoritmo Shapiro-Senapathy (S&S). [46] [47]
Este algoritmo ayuda a identificar mutaciones de empalme que causan enfermedades y reacciones adversas a los medicamentos. [46] [47] Los científicos utilizaron el algoritmo para identificar mutaciones y genes que causan cánceres, trastornos hereditarios, enfermedades de inmunodeficiencia y trastornos neurológicos. Se utiliza cada vez más en la práctica clínica y la investigación para encontrar mutaciones en genes causantes de enfermedades conocidos en pacientes y para descubrir genes nuevos que sean causales de diferentes enfermedades. Además, se utiliza para definir los sitios de empalme crípticos y deducir los mecanismos por los cuales las mutaciones pueden afectar el empalme normal y conducir a diferentes enfermedades. También se emplea en la investigación básica.
Los hallazgos basados en S&S han tenido un impacto en cuestiones importantes en la biología eucariota y en la medicina humana. [48]
La teoría de los genes divididos implica que las características estructurales de los genes divididos predichas a partir de secuencias aleatorias simuladas por computadora ocurren en genes divididos eucariotas. Esto se confirma en la mayoría de los genes divididos conocidos. Las secuencias muestran una distribución exponencial negativa casi perfecta de longitudes de ORF. [1] [2] [22] [3] Con raras excepciones, los exones de genes eucariotas caen dentro del máximo predicho de 600 bases.
La teoría predice correctamente que los exones están delimitados por codones de terminación, especialmente en los extremos 3' de los exones. En realidad, están precisamente delimitados con mayor fuerza en los extremos 3' de los exones y con menor fuerza en los extremos 5' en la mayoría de los genes conocidos, como se predijo. [1] [2] [22] [3] Estos codones de terminación son las partes funcionales más importantes de ambas uniones de empalme. Por lo tanto, la teoría proporciona una explicación para las uniones de empalme "conservadas" en los extremos de los exones y para la pérdida de estos codones de terminación junto con los intrones cuando se eliminan. La teoría predice correctamente que las uniones de empalme se distribuyen aleatoriamente en las secuencias de ADN eucariota. [3] [25] [46] [47] La teoría predice correctamente que las uniones de empalme presentes en los genes de ARN de transferencia y los genes de ARN ribosómico no contienen codones de terminación. La señal de lazo, otra secuencia involucrada en el proceso de empalme, también contiene codones de terminación. [1] [2] [3] [22] [25] [46] [47]
La teoría predice correctamente que los intrones no son codificantes y que en su mayoría no son funcionales. A excepción de algunas secuencias de intrones, incluidas las secuencias de señal de empalme del donante y el aceptor y las secuencias de punto de ramificación, y posiblemente los potenciadores de empalme de intrones que se encuentran en los extremos de los intrones, que ayudan a la eliminación de intrones, la gran mayoría de los intrones carecen de cualquier función. La teoría no excluye secuencias raras dentro de los intrones que podrían ser utilizadas por el genoma y la célula, especialmente porque los intrones son muy largos.
De este modo, las predicciones de la teoría quedan corroboradas con precisión por los principales elementos de los genomas eucariotas modernos.
Un análisis comparativo de los datos del genoma moderno de varios organismos vivos reveló que las características de los genes divididos se remontan a los organismos más antiguos. Estos organismos podrían haber contenido los genes divididos y las proteínas complejas que se encuentran en los organismos vivos actuales. [49] [ 50] [51] [52 ] [53] [54] [55] [56] [57]
Los estudios que emplean el análisis de máxima verosimilitud encontraron que los primeros organismos eucariotas contenían los mismos genes que los organismos modernos con una densidad de intrones aún mayor. [58] La genómica comparativa de muchos organismos, incluidos los eucariotas basales [59] (considerados organismos eucariotas primitivos como Amoeboflagellata, Diplomonadida y Parabasalia ) mostró que los genes divididos ricos en intrones acompañaban y el espliceosoma de los organismos modernos estaban presentes en sus primeros antepasados, y que los primeros organismos venían con todos los componentes celulares eucariotas. [60] [49] [61] [62] [63] [58]
{{cite journal}}: Requiere citar revista |journal=( ayuda )