
En genética , el término sintenia se refiere a dos conceptos relacionados:
La Enciclopedia Británica da la siguiente descripción de la sintenia, utilizando la definición moderna: [2]
La secuenciación y el mapeo genómico han permitido comparar las estructuras generales de los genomas de muchas especies diferentes. El hallazgo general es que los organismos de divergencia relativamente reciente muestran bloques similares de genes en las mismas posiciones relativas en el genoma. Esta situación se llama sintenia, que se traduce aproximadamente como poseer secuencias cromosómicas comunes. Por ejemplo, muchos de los genes de los humanos son sinténicos con los de otros mamíferos, no solo simios sino también vacas, ratones, etc. El estudio de la sintenia puede mostrar cómo se corta y pega el genoma en el curso de la evolución.
Synteny es un neologismo que significa "en la misma cinta"; del griego σύν , syn "junto con" + ταινία , tainiā "banda". Esto se puede interpretar clásicamente como "en el mismo cromosoma", o en el sentido moderno de tener el mismo orden de genes en dos cadenas (homólogas) de ADN (o cromosomas).
El concepto clásico está relacionado con el ligamiento genético : el ligamiento entre dos loci se establece mediante la observación de frecuencias de recombinación menores a las esperadas entre ellos. Por el contrario, cualquier loci en el mismo cromosoma es por definición sinténico, incluso si su frecuencia de recombinación no se puede distinguir de los loci no ligados mediante experimentos prácticos. Por lo tanto, en teoría, todos los loci ligados son sinténicos, pero no todos los loci sinténicos están necesariamente ligados. De manera similar, en genómica , los loci genéticos en un cromosoma son sinténicos independientemente de si esta relación se puede establecer mediante métodos experimentales como la secuenciación /ensamblaje de ADN , el recorrido del genoma , la localización física o el mapeo hap .
Los estudiantes de genética (clásica) emplean el término sintenia para describir la situación en la que dos loci genéticos han sido asignados al mismo cromosoma pero aún así pueden estar separados por una distancia suficientemente grande en unidades de mapa como para que no se haya demostrado el ligamiento genético.
La sintenia compartida (también conocida como sintenia conservada) describe la co-localización preservada de genes en cromosomas de diferentes especies. Durante la evolución , los reordenamientos del genoma, como las translocaciones cromosómicas, pueden separar dos loci, lo que resulta en la pérdida de sintenia entre ellos. Por el contrario, las translocaciones también pueden unir dos fragmentos de cromosomas previamente separados, lo que resulta en una ganancia de sintenia entre loci. Una sintenia compartida más fuerte de lo esperado puede reflejar la selección de relaciones funcionales entre genes sinténicos, como combinaciones de alelos que son ventajosas cuando se heredan juntos, o mecanismos reguladores compartidos. [3]
A la luz del cambio más reciente en el significado de sintenia , esta conservación del contenido genético y el enlace sin preservación del orden también se ha denominado mesosintenia . [4]
En la actualidad (desde ~2000) el término se utiliza más comúnmente para describir la preservación del orden preciso de los genes en un cromosoma transmitido de un ancestro común, [5] [6] [7] [8] a pesar de que los genetistas más "de la vieja escuela" rechazan lo que perciben como una apropiación indebida del término, [9] prefiriendo en su lugar la colinealidad . [10]
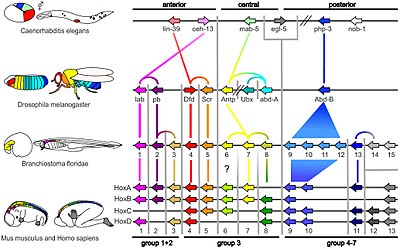
El análisis de la sintenia en el sentido del orden genético tiene varias aplicaciones en genómica. La sintenia compartida es uno de los criterios más fiables para establecer la ortología de las regiones genómicas en diferentes especies. Además, la conservación excepcional de la sintenia puede reflejar importantes relaciones funcionales entre genes. Por ejemplo, el orden de los genes en el " grupo Hox ", que son determinantes clave del plan corporal animal y que interactúan entre sí de manera crítica, se conserva esencialmente en todo el reino animal. [11]
La sintenia se utiliza ampliamente en el estudio de genomas complejos, ya que la genómica comparativa permite la presencia y posible función de genes en un organismo modelo más simple para inferir los de uno más complejo. Por ejemplo, el trigo tiene un genoma muy grande y complejo que es difícil de estudiar. En 1994, una investigación del Centro John Innes en Inglaterra y el Instituto Nacional de Investigación Agrobiológica en Japón demostró que el genoma del arroz, mucho más pequeño, tenía una estructura y un orden genético similar al del trigo. [12] Estudios posteriores descubrieron que muchos cereales son sinténicos [13] y, por lo tanto, plantas como el arroz o la gramínea Brachypodium podrían usarse como modelo para encontrar genes o marcadores genéticos de interés que podrían usarse en el mejoramiento y la investigación del trigo. En este contexto, la sintenia también fue esencial para identificar una región muy importante en el trigo, el locus Ph1 involucrado en la estabilidad y fertilidad del genoma, que se localizó utilizando información de regiones sinténicas en el arroz y Brachypodium. [14]
La sintenia también se utiliza ampliamente en la genómica microbiana. En Hyphomicrobiales y Enterobacteriales , los genes sinténicos codifican una gran cantidad de funciones celulares esenciales y representan un alto nivel de relaciones funcionales. [15]
Los patrones de sintenia compartida o de rupturas de sintenia también pueden utilizarse como caracteres para inferir las relaciones filogenéticas entre varias especies, e incluso para inferir la organización del genoma de especies ancestrales extintas. A veces se establece una distinción cualitativa entre macrosintnia , la conservación de la sintenia en grandes porciones de un cromosoma, y microsintnia , la conservación de la sintenia solo para unos pocos genes a la vez.
La sintenia compartida entre diferentes especies se puede inferir a partir de sus secuencias genómicas. Esto se hace típicamente usando una versión del algoritmo MCScan, que encuentra bloques sinténicos entre especies comparando sus genes homólogos y buscando patrones comunes de colinealidad en una escala cromosómica o de contig . Las homologías se determinan generalmente sobre la base de los aciertos de BLAST de alta puntuación de bits que ocurren entre múltiples genomas. A partir de aquí, se utiliza programación dinámica para seleccionar la mejor ruta de puntuación de genes homólogos compartidos entre especies, teniendo en cuenta la posible pérdida y ganancia de genes que puede haber ocurrido en las historias evolutivas de las especies. [16]