
En genética , un silenciador es una secuencia de ADN capaz de unirse a factores de regulación de la transcripción , llamados represores . El ADN contiene genes y proporciona la plantilla para producir ARN mensajero (ARNm). Ese ARNm luego se traduce en proteínas. Cuando una proteína represora se une a la región silenciadora del ADN, se impide que la ARN polimerasa transcriba la secuencia de ADN en ARN. Al bloquearse la transcripción, la traducción del ARN en proteínas es imposible. Por lo tanto, los silenciadores impiden que los genes se expresen como proteínas. [1]
La ARN polimerasa, una enzima dependiente del ADN, transcribe las secuencias de ADN, llamadas nucleótidos , en la dirección 3' a 5' mientras que el ARN complementario se sintetiza en la dirección 5' a 3'. El ARN es similar al ADN, excepto que el ARN contiene uracilo, en lugar de timina, que forma un par de bases con la adenina. Una región importante para la actividad de represión y expresión génica que se encuentra en el ARN es la región 3' no traducida . Esta es una región en el extremo 3' del ARN que no se traducirá a proteína pero incluye muchas regiones reguladoras.
Aún no se sabe mucho sobre los silenciadores, pero los científicos continúan estudiando con la esperanza de clasificar más tipos, ubicaciones en el genoma y enfermedades asociadas con ellos. [2] [3]

Un silenciador es un elemento específico de una secuencia que induce un efecto negativo en la transcripción de un gen en particular. Hay muchas posiciones en las que se puede ubicar un elemento silenciador en el ADN. La posición más común se encuentra aguas arriba del gen diana, donde puede ayudar a reprimir la transcripción del gen. [4] Esta distancia puede variar en gran medida entre aproximadamente -20 pb y -2000 pb aguas arriba de un gen. Ciertos silenciadores se pueden encontrar aguas abajo de un promotor ubicado dentro del intrón o exón del propio gen. También se han encontrado silenciadores dentro de la región no traducida 3' (UTR 3') del ARNm. [5]
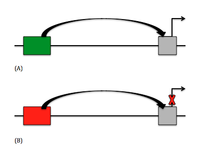
Actualmente, existen dos tipos principales de silenciadores en el ADN, que son el elemento silenciador clásico y el elemento regulador negativo no clásico (NRE). En los silenciadores clásicos, el gen es reprimido activamente por el elemento silenciador, principalmente al interferir con el ensamblaje del factor de transcripción general (GTF). [5] Los NRE reprimen pasivamente el gen, generalmente al inhibir otros elementos que se encuentran aguas arriba del gen. De los NRE, hay ciertos silenciadores que dependen de la orientación, lo que significa que el factor de unión se une en una dirección particular en relación con otras secuencias. Los silenciadores dependientes del promotor se entienden como elementos silenciadores porque dependen de la posición y la orientación, pero también deben utilizar un factor específico del promotor. [5] Recientemente se han descubierto los elementos de respuesta del grupo Polycomb (PRE), que pueden permitir e inhibir la represión dependiendo de la proteína unida a ellos y de la presencia de transcripción no codificante. [4]

Para los silenciadores clásicos, la vía de señalización es relativamente simple. Dado que la represión es activa, los elementos silenciadores se dirigen al ensamblaje de GTF, necesarios para la transcripción del gen. Estos elementos silenciadores se encuentran principalmente aguas arriba del gen y pueden variar entre distancias cortas y largas. Para los silenciadores de largo alcance, se ha observado que el ADN formará un bucle para acercar el silenciador al promotor y hacer un bucle para eliminar el ADN interferente. [4] Los silenciadores también se dirigen a los sitios de helicasa en el ADN que son ricos en adenina y timina (AT) y propensos a desenrollar el ADN, lo que deja espacio para iniciar la transcripción. La actividad de helicasa inhibida conduce a la inhibición de la transcripción. Esto se ve comúnmente en el promotor del gen humano tirotropina-β . Los NRE pueden inducir una curvatura en la región promotora para bloquear las interacciones, como se ve cuando un NRE se une a Yin-Yang 1 ( YY1 ), [5] y flanquean señales reguladoras o regiones promotoras también. Cuando la región silenciadora se encuentra dentro de un intrón, pueden darse dos tipos de represiones. En primer lugar, puede haber un bloqueo físico de un sitio de empalme. En segundo lugar, puede haber una curvatura en el ADN que inhiba el procesamiento del ARN. [5]
Cuando se encuentra en el exón o en la región no traducida, el silenciador será principalmente clásico o dependiente de la posición. Sin embargo, estos silenciadores pueden llevar a cabo su actividad antes de la transcripción. [5] La mayoría de los silenciadores se expresan de forma constitutiva en los organismos, y solo permiten la activación de un gen inhibiendo el silenciador o activando una región potenciadora. El mejor ejemplo de esto es el factor silenciador restrictivo neuronal (NRSF) que produce el gen REST . El gen REST produce NRSF para reprimir la transcripción de genes neuronales que son esenciales para la localización del tejido neuronal. Cuando un silenciador reprime REST , el NRSF también se inhibe, lo que permite la transcripción de genes neuronales. [5]
Otro elemento regulador ubicado aguas arriba del gen es un potenciador . Los potenciadores funcionan como un interruptor de "activación" en la expresión génica y activarán la región promotora de un gen en particular, mientras que los silenciadores actúan como un interruptor de "desactivación". Aunque estos dos elementos reguladores trabajan uno contra el otro, ambos tipos de secuencia afectan a la región promotora de formas muy similares. [4] Debido a que los silenciadores no han sido identificados y analizados a fondo, la amplia investigación sobre potenciadores ha ayudado a los biólogos a comprender la mecánica del silenciador. Los potenciadores se pueden encontrar en muchas de las mismas áreas en las que se encuentran los silenciadores, como aguas arriba del promotor por muchos pares de kilobases, o incluso aguas abajo dentro del intrón del gen. [4] El bucle de ADN también es una función modelo utilizada por los potenciadores para acortar la proximidad del promotor al potenciador. Los potenciadores también funcionan con factores de transcripción para iniciar la expresión, de forma muy similar a como los silenciadores pueden hacerlo con los represores. [4]

Existen varias diferencias en la regulación del control metabólico en eucariotas y en procariotas. Los procariotas varían el número de enzimas específicas producidas en sus células para regular la expresión génica, que es un control metabólico lento, y también regulan las vías enzimáticas a través de mecanismos como la inhibición por retroalimentación y la regulación alostérica , que es un control metabólico rápido. [6] Los genes de los procariotas se agrupan en función de funciones similares en unidades llamadas operones que consisten en un promotor y un operador . El operador es el sitio de unión para el represor y, por lo tanto, tiene una función equivalente a la región silenciadora en el ADN eucariota. Cuando una proteína represora está unida al operador, la ARN polimerasa no puede unirse al promotor para iniciar la transcripción del operón.
El operón lac en la procariota E. coli consiste en genes que producen enzimas para descomponer la lactosa. Su operón es un ejemplo de un silenciador procariota. Los tres genes funcionales en este operón son lacZ, lacY y lacA. [6] El gen represor, lacI, producirá la proteína represora LacI que está bajo regulación alostérica. Estos genes se activan por la presencia de lactosa en la célula que actúa como una molécula efectora que se une a LacI. Cuando el represor está unido a la lactosa, no se unirá al operador, lo que permite que la ARN polimerasa se una al promotor para iniciar la transcripción del operón. Cuando el sitio alostérico del represor no está unido a la lactosa, su sitio activo se unirá al operador para evitar que la ARN polimerasa transcriba los genes del operón lac .
Los eucariotas tienen un genoma mucho más grande y, por lo tanto, tienen diferentes métodos de regulación genética que los procariotas. Todas las células de un organismo eucariota tienen el mismo ADN, pero se especifican a través de la expresión genética diferencial, un fenómeno conocido como totipotencia genética . [7] Sin embargo, para que una célula exprese los genes para su correcto funcionamiento, los genes deben estar estrechamente regulados para expresar las propiedades correctas. Los genes en eucariotas se controlan en los niveles transcripcional , postranscripcional , traduccional y postraduccional . [8] En el nivel transcripcional, la expresión genética se regula alterando las tasas de transcripción. Los genes que codifican proteínas incluyen exones que codificarán los polipéptidos, intrones que se eliminan del ARNm antes de la traducción de las proteínas, un sitio de inicio de la transcripción en el que se une la ARN polimerasa y un promotor. [9]
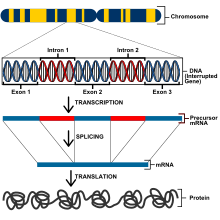
Los genes eucariotas contienen un promotor ascendente y un promotor central también denominado promotor basal. Un promotor basal común es la secuencia TATAAAAAA conocida como caja TATA . La caja TATA es un complejo con varias proteínas diferentes, incluido el factor de transcripción II D (TFIID), que incluye la proteína de unión a TATA (TBP), que se une a la caja TATA junto con otras 13 proteínas que se unen a TBP. Las proteínas de unión a la caja TATA también incluyen el factor de transcripción II B (TFIIB), que se une a las polimerasas de ADN y ARN. [9]
Los silenciadores en eucariotas controlan la expresión génica a nivel transcripcional en el que el ARNm no se transcribe. Estas secuencias de ADN pueden actuar como silenciadores o potenciadores en función del factor de transcripción que se une a la secuencia y la unión de esta secuencia evitará que los promotores, como la caja TATA, se unan a la ARN polimerasa. [7] Una proteína represora puede tener regiones que se unan a la secuencia de ADN, así como regiones que se unan a los factores de transcripción ensamblados en el promotor del gen, lo que crearía un mecanismo de bucle cromosómico. [9] El bucle acerca a los silenciadores a los promotores para garantizar que los grupos de proteínas necesarios para la expresión génica óptima trabajen juntos.

Las mutaciones genéticas ocurren cuando se alteran las secuencias de nucleótidos en un organismo. Estas mutaciones conducen no solo a influencias fenotípicas observables en un individuo, sino también a alteraciones que son indetectables fenotípicamente. Las fuentes de estas mutaciones pueden ser errores durante la replicación, mutaciones espontáneas y mutágenos químicos y físicos ( radiación UV e ionizante , calor). [10] Los silenciadores, al estar codificados en el genoma, son susceptibles a tales alteraciones que, en muchos casos, pueden conducir a graves anomalías fenotípicas y funcionales. En términos generales, las mutaciones en elementos o regiones silenciadoras podrían conducir a la inhibición de la acción del silenciador o a la represión persistente de un gen necesario. Esto puede conducir a la expresión o supresión de un fenotipo no deseado que puede afectar la funcionalidad normal de ciertos sistemas en el organismo. Entre los muchos elementos y proteínas silenciadores, REST/NSRF es un factor silenciador importante que tiene una variedad de impactos, no solo en los aspectos neuronales del desarrollo. De hecho, en muchos casos, REST/NSRF actúa en conjunción con RE-1/NRSE para reprimir e influir en las células no neuronales. [11] Sus efectos varían desde las ranas ( Xenopus laevis ) hasta los humanos, con innumerables efectos en el fenotipo y también en el desarrollo. En Xenopus laevis , el mal funcionamiento o daño de REST/NRSF se ha asociado a un patrón ectodérmico anormal durante el desarrollo y consecuencias significativas en el tubo neural, los ganglios craneales y el desarrollo ocular. [12] En humanos, una deficiencia en el elemento silenciador REST/NSRF se ha correlacionado con la enfermedad de Huntington debido a la disminución en la transcripción de BDNF .
Además, estudios en curso indican que NRSE está involucrado en la regulación del gen ANP, que cuando se sobreexpresa, puede conducir a hipertrofia ventricular . [13] Las mutaciones en los complejos del grupo Polycomb (PcG) también presentaron modificaciones significativas en los sistemas fisiológicos de los organismos. Por lo tanto, la modificación de los elementos y secuencias silenciadores puede resultar en cambios devastadores o imperceptibles.
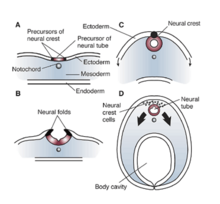
Los efectos e influencias de RE1/NRSE y REST/NRSF son significativos en células no neuronales que requieren la represión o silenciamiento de genes neuronales. Estos elementos silenciadores también regulan la expresión de genes que no inducen proteínas específicas de neuronas y los estudios han demostrado el amplio impacto que estos factores tienen en los procesos celulares. En Xenopus laevis, la disfunción o mutación de RE1/NRSE y REST/NRSF demostró un impacto significativo en el tubo neural , los ganglios craneales y el desarrollo ocular. [12] Todas estas alteraciones se pueden rastrear hasta un patrón inadecuado del ectodermo durante el desarrollo de Xenopus. Por lo tanto, una mutación o alteración en la región silenciadora RE1/NRSE o en el factor silenciador REST/NRSF puede alterar la diferenciación y especificación adecuadas del dominio neuroepitelial y también obstaculizar la formación de piel o ectodermo. [12] La falta de estos factores da como resultado una disminución de la producción de proteína morfogenética ósea (BMP), que se traduce en un desarrollo deficiente de la cresta neural . [12] Por lo tanto, los efectos de NRSE y NRSF son de importancia fundamental para la neurogénesis del embrión en desarrollo, y también en las primeras etapas de la formación de patrones ectodérmicos. En última instancia, el funcionamiento inadecuado de estos factores puede dar lugar a un desarrollo aberrante del tubo neural, de los ganglios craneales y de los ojos en Xenopus .
La enfermedad de Huntington (EH) es un trastorno neurodegenerativo hereditario, cuyos síntomas aparecen durante la adultez media. Los síntomas más notorios de esta enfermedad progresiva son el deterioro cognitivo y motor, así como las alteraciones del comportamiento. [14] Estos deterioros pueden derivar en demencia , corea y, finalmente, la muerte. A nivel molecular, la EH es el resultado de una mutación en la proteína huntingtina (Htt). Más específicamente, hay una repetición anormal de una secuencia CAG hacia el extremo 5' del gen, que luego conduce al desarrollo de un tramo de poliglutamina tóxica (poliQ) en la proteína. La proteína Htt mutada afecta las funciones neuronales adecuadas de un individuo al inhibir la acción de REST/NRSF.
REST/NRSF es un elemento silenciador importante que se une a las regiones reguladoras para controlar la expresión de ciertas proteínas implicadas en las funciones neuronales. Las acciones mecanísticas de la huntingtina aún no se comprenden por completo, pero existe una correlación entre Htt y REST/NRSF en el desarrollo de la EH. Al unirse a REST/NRSF, la proteína huntingtina mutada inhibe la acción del elemento silenciador y lo retiene en el citosol. Por lo tanto, REST/NRSF no puede entrar en el núcleo y unirse al elemento regulador RE-1/NRSE de 21 pares de bases. Una represión adecuada de genes diana específicos es de fundamental importancia, ya que muchos están involucrados en el desarrollo adecuado de receptores neuronales, neurotransmisores , proteínas de vesículas sinápticas y proteínas de canal. Una deficiencia en el desarrollo adecuado de estas proteínas puede causar las disfunciones neuronales observadas en la enfermedad de Huntington. Además de la falta de represión debido al REST/NRSF inactivo, la proteína huntingtina mutada también puede disminuir la transcripción del gen del factor neurotrópico derivado del cerebro (BDNF). El BDNF influye en la supervivencia y el desarrollo de las neuronas del sistema nervioso central y del sistema nervioso periférico. Esta represión anormal se produce cuando la región RE1/NRSE dentro de la región promotora del BDNF se activa por la unión de REST/NRSF, lo que conduce a la falta de transcripción del gen del BDNF. [15] Por lo tanto, la represión anómala de la proteína BDNF sugiere un impacto significativo en la enfermedad de Huntington.
REST/NRSF en conjunción con RE1/NRSE también actúa fuera del sistema nervioso como reguladores y represores. La investigación actual ha vinculado la actividad de RE1/NRSE con la regulación de la expresión del gen del péptido natriurético atrial ( ANP ). [13] Una región reguladora de NRSE está presente en la región no traducida 3' del gen ANP y actúa como mediador para su expresión apropiada. La proteína codificada por el gen ANP es importante durante el desarrollo embrionario para la maduración y el desarrollo de los miocitos cardíacos . Sin embargo, durante la primera infancia y durante la edad adulta, la expresión de ANP se suprime o se mantiene al mínimo en el ventrículo. Por lo tanto, una inducción anormal del gen ANP puede conducir a hipertrofia ventricular y consecuencias cardíacas graves. Para mantener la represión del gen, NRSF (factor silenciador restrictivo neuronal) o REST se une a la región NRSE en la región no traducida 3' del gen ANP . Además, el complejo NRSF-NRSE recluta un correpresor transcripcional conocido como mSin3. [13] Esto conduce a la actividad de la histona desacetilasa en la región y a la represión del gen. Por lo tanto, los estudios han revelado la correlación entre REST/NRSF y RE1/NRSE en la regulación de la expresión del gen ANP en los miocitos ventriculares. Una mutación en NRSF o NRSE puede conducir a un desarrollo indeseable de los miocitos ventriculares, debido a la falta de represión, lo que puede causar hipertrofia ventricular. La hipertrofia ventricular izquierda, por ejemplo, aumenta la probabilidad de muerte súbita de un individuo debido a una arritmia ventricular resultante del aumento de la masa ventricular. [16] Además de la influencia en el gen ANP , la secuencia NRSE regula otros genes embrionarios cardíacos, como el péptido natriurético cerebral BNP, la α-actina esquelética y la subunidad α3 de la Na, K – ATPasa. [13] Por lo tanto, la actividad reguladora tanto de NRSE como de NRSF en mamíferos previene no sólo disfunciones neuronales sino también anomalías fisiológicas y fenotípicas en otras regiones no neuronales del cuerpo.

Los complejos reguladores del grupo Polycomb (PcG) son conocidos por su influencia en la regulación epigenética de las células madre, especialmente en las células madre hematopoyéticas . El complejo represor Polycomb 1 (PRC 1) está directamente involucrado en el proceso de hematopoyesis, y funciona junto con, por ejemplo, el gen PcG “Bmi1”. Estudios en ratones indican que los organismos con “Bmi1” mutado demuestran un funcionamiento mitocondrial deficiente, y también obstaculizan la capacidad de las células hematopoyéticas para auto-renovarse. Asimismo, las mutaciones en los genes PRC2 se relacionaron con afecciones hematológicas como la leucemia linfoblástica aguda (LLA), que es una forma de leucemia. Por lo tanto, los genes y proteínas del grupo Polycomb están involucrados en el mantenimiento adecuado de la hematopoyesis en el cuerpo. [17]